-
氟喹诺酮类药物因其广谱性和疗效好而被广泛应用于兽医临床治疗,目前使用最多的是第3代产品,包括氧氟沙星,诺氟沙星(NOR)、恩诺沙星(ENR)和环丙沙星等。进入动物体内的药物不能被完全吸收,50%以上会随动物粪尿排出体外,最终导致大量氟喹诺酮类化合物进入环境中。有研究[1-3]表明,此类药物可以在许多环境基质中检出,甚至在一些居民生活供水中也有少量存在。在对广州多处饮用水进行药物检测分析时发现,氟喹诺酮药物的浓度为1.0~679.7 ng·L−1 [4];FICK等[5]从多个饮用水井取样检测,结果显示其中某些氟喹诺酮类药物浓度高达1 µg·L−1。不同环境基质中的氟喹诺酮类药物可能会影响环境过程、破坏生态系统服务并导致氟喹诺酮类耐药基因的产生和传播[6],最终会对人类健康造成威胁。为使畜禽粪便在作为肥料资源利用之前能够尽可能地去除其中残留的抗生素药物,通常利用高温堆肥工艺对其进行处理,但氟喹诺酮类药物因喹诺酮环的存在而表现出较高的稳定性,特别是耐水解和耐高温等性质,普通的堆肥很难实现该类药物的完全去除。而且,氟喹诺酮类化合物作为抗菌药物,还可抵抗微生物的降解转化[7]。据报道[8],氟喹诺酮类药物在高温堆肥中具有较强的抗逆性,因此,其在堆肥产品中的大量残留已成为一个急待解决的问题。
近年来,研究者探索了电化学氧化[9]、高级氧化[10]、光降解[11]、材料吸附[12]和生物降解法[13]等多种方法以去除环境中的抗生素污染。生物降解法作为一种环境友好且有效的抗生素去除方法受到了广泛的关注,而微生物在环境污染物的生物降解中起着重要的作用。有研究[14]发现,堆肥中氟喹诺酮类药物的完全去除可通过接种微生物来实现。目前,已发现多种微生物具有降解氟喹诺酮类药物的能力,如微杆菌属的细菌可降解NOR,白腐真菌(Irpex lacteus, Panus tigrinus, Dichomitus squalens等)能降解转化NOR、氧氟沙星和环丙沙星[15-18]。另外,为了确定该类药物的生物降解模式,一些研究[19-20]分析了典型的氟喹诺酮类药物生物降解后的产物。据报道[21],ENR可以通过木腐真菌转化为CO2和其他一些代谢物;环丙沙星也可被木腐菌通过羟基化、脱羧、脱氟和哌嗪环降解等途径转化。然而以上关于微生物降解转化氟喹诺酮类药物性能和降解机制的实验均是在常温(25~30 ℃)的条件下进行,在与堆肥温度相近的热环境中(70 ℃)的相关研究报道较少。
本研究以前期从药厂污泥筛选得到的嗜热菌Thermus sp. C419(CGMCC 1.16184, GenBank登录号: KY784655C419)[22]为降解菌株,探究其在70 ℃的高温条件下对常用的2种氟喹诺酮类药物(NOR和ENR)单独和混合存在时的生物降解情况,并对药物的降解动力学、生物降解产物和可能的代谢途径进行研究分析,最后通过鉴定生物降解后药物的残留抗菌活性,分析其对微生物的毒性大小。本研究探索了氟喹诺酮类药物在高温条件下的降解转化,以期为畜禽粪便高温堆肥工艺提供可降解氟喹诺酮类药物的堆肥菌剂,实现堆肥过程畜禽粪便中氟喹诺酮类药物的高效降解。
全文HTML
-
本实验过程中所用的NOR和ENR购于上海阿拉丁生化科技股份有限公司。实验采用无机盐培养基(MMSM)进行生物降解实验,主要成分包括FeSO4·7H2O 0.013 g·L−1、CaCl2·2H2O 0.013 g·L−1、Na2EDTA·2H2O 0.018 g·L−1、MgSO4·7H2O 0.25 g·L−1、KH2PO4 5 g·L−1、NH4NO3 5 g·L−1、Na2HPO4 7.5 g·L−1、酵母提取物0.6 g·L−1、乙酸钠0.5 g·L−1、NOR/ENR 5 mg·L−1或10 mg·L−1。采用Luria-Bertani(LB)培养基(胰蛋白胨10 g·L−1、NaCl 5 g·L−1、酵母提取物5 g·L−1)进行细菌细胞的增殖培养。
-
1)生物降解实验。250 mL的锥形瓶中添加100 mL的MMSM培养基(乙酸钠浓度0.5 g·L−1),接种3%的菌株C419菌悬液,并根据畜禽粪便中抗生素的典型浓度(1~10 mg·kg−1)[23],设定降解单一药物实验中的低剂量组NOR/ENR药物浓度为5 mg·L−1,高剂量组NOR/ENR药物浓度为10 mg·L−1;2种药物混合降解实验体系中,NOR/ENR药物浓度各为5 mg·L−1。培养液置于70 ℃、转速为150 r·min−1的摇床避光培养,在0、12、24、48、72、120、168和216 h取样测定药物残留浓度以及菌体生长量。实验中通过称重法确定培养期间蒸发水的量,并在取样前添加无菌水至原重量。取样后,样品利用0.45 µm针式滤膜过滤,所获液体保存在棕色色谱瓶中并存放在4 ℃的冰箱中待测。
利用一级动力学模型拟合菌株C419对氟喹诺酮类生物降解动力学,计算方法见式(1)。
式中:C0为初始浓度,mg·L−1;C为实验时间t时的药物浓度,mg·L−1;k为降解速率常数,h−1。
生物降解的半衰期t1/2的计算方法如式(2)所示。
2)生物降解代谢物的提取。取氟喹诺酮药物生物降解后的上清液进行离心(8 000 r·min−1),并用针式滤膜(0.22 µm)过滤。用与样品等量的乙酸乙酯提取样品中的降解产物,提取步骤重复3次。收集乙酸乙酯,用氮吹仪将其吹干,最后用甲醇溶解提取物,所获液体保存在棕色色谱瓶中并存放在4 ℃的冰箱中待测。
3)残留抗菌活性测定。通过改进的平板扩散药敏实验,利用枯草芽孢杆菌(Bacillus subtilis,革兰氏阴性)和大肠杆菌K12 (Escherichia coli,革兰氏阴性)对氟喹诺酮类药物及其代谢物的残留抗菌活性进行检测[18]。主要实验步骤:将1 mL的菌悬液(OD600 = 1.0)接种到LB半固体培养基(琼脂含量0.8%)中混合均匀,并分装至培养皿中。待培养基凝固后,在培养基上放置4个直径为6 mm的牛津杯,并在杯子中加入样品和氟喹诺酮类药物(5 mg·L−1)各200 µL。将培养皿置于在37 ℃培养箱中,20 h之后取出测定抑菌圈的大小。通过对比原始药物的抑菌圈与降解后样品抑菌圈的大小得到相对的抑制率,以此来评估实验样品残留的抗菌活性。
-
利用高效液相色谱(Hitachi L-2000, 日本)测定各氟喹诺酮类抗生素的浓度。检测条件如下:流动相为0.02 mol·L−1的三氯乙酸、乙腈和甲醇(74∶22∶4,体积比),利用安捷伦C-18色谱柱(250 mm×4.6 mm,5 µm)进行色谱分离,柱温设定为30 ℃,激发波长设定为278 nm,进样量为10 µL。
利用超高效液相色谱串联质谱(UPLC-MS/MS, AB Sciex 6500,美国)对氟喹诺酮类抗生素的微生物降解代谢产物进行分析。色谱柱为岛津C18色谱柱(20 mm×75 mm, 1.6 µm,日本),色谱分离温度为30 ℃。流动相由0.1%甲酸水溶液(A)和0.1%甲酸乙腈溶液(B)组成。流速为0.3 mL·min−1,样品注入量为10 µL。洗脱程序为:B相在20 min内完成10%~100%的梯度洗脱,随后以10%的B相等度洗脱5 min。该质谱仪配备了一个电喷雾接口(ESI),ESI源设定为正离子模式,温度为350 ℃,电压设置为4.5 kV。检测器于质荷比为50~700时对样品进行全面扫描,获得代谢物。检测结束后,利用分析软件Analyst 1.6.3对结果进行分析。根据查阅的文献和EAWAG-BBD代谢通路预测系统(http://eawag-bbd.ethz.ch/UNK t/)的预测路径确定降解产物,并在Low-MS条件下对产物进行鉴定,进一步证实了降解产物的结构。
1.1. 药品与培养基
1.2. 实验方法
1.3. 分析方法
-
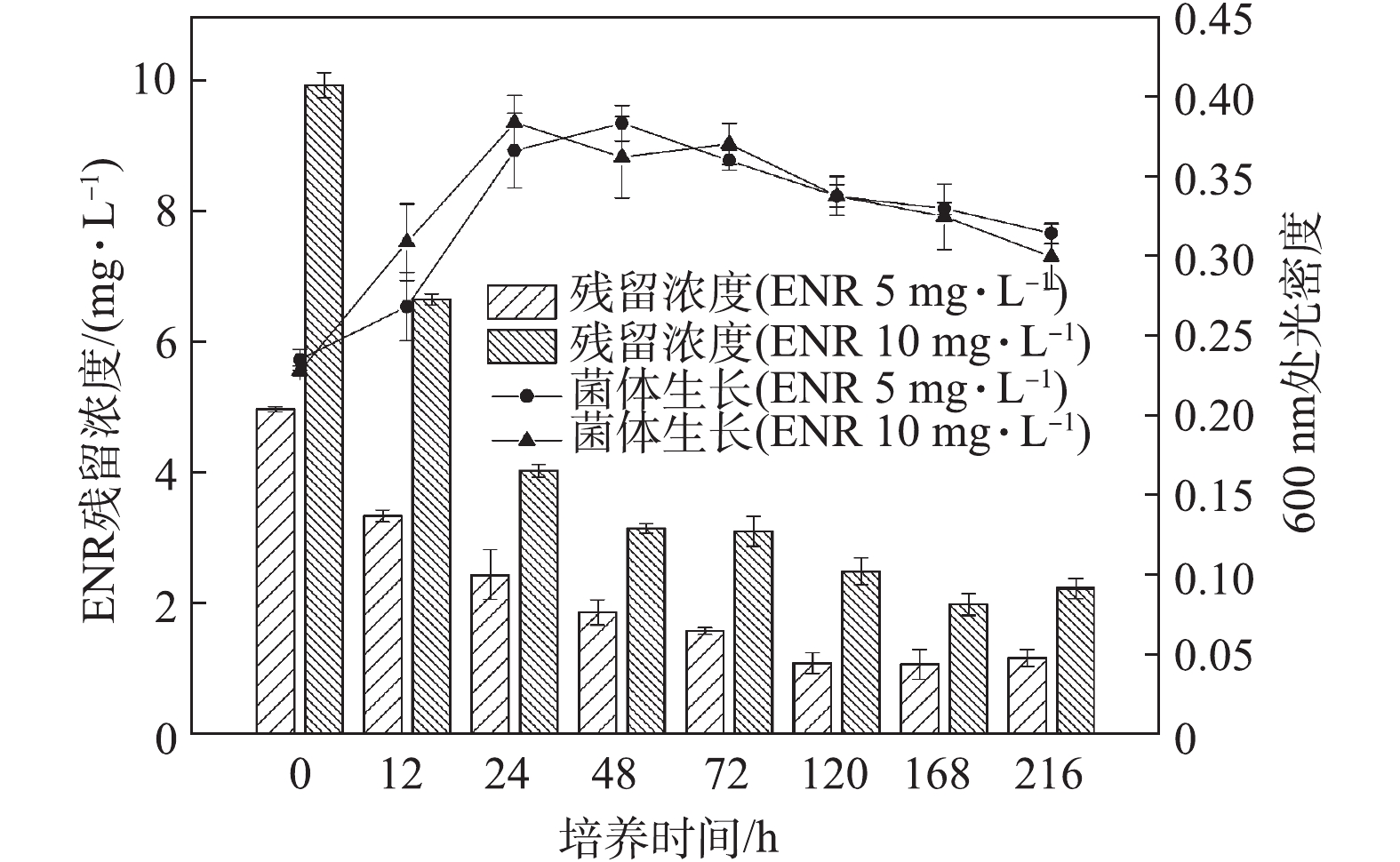
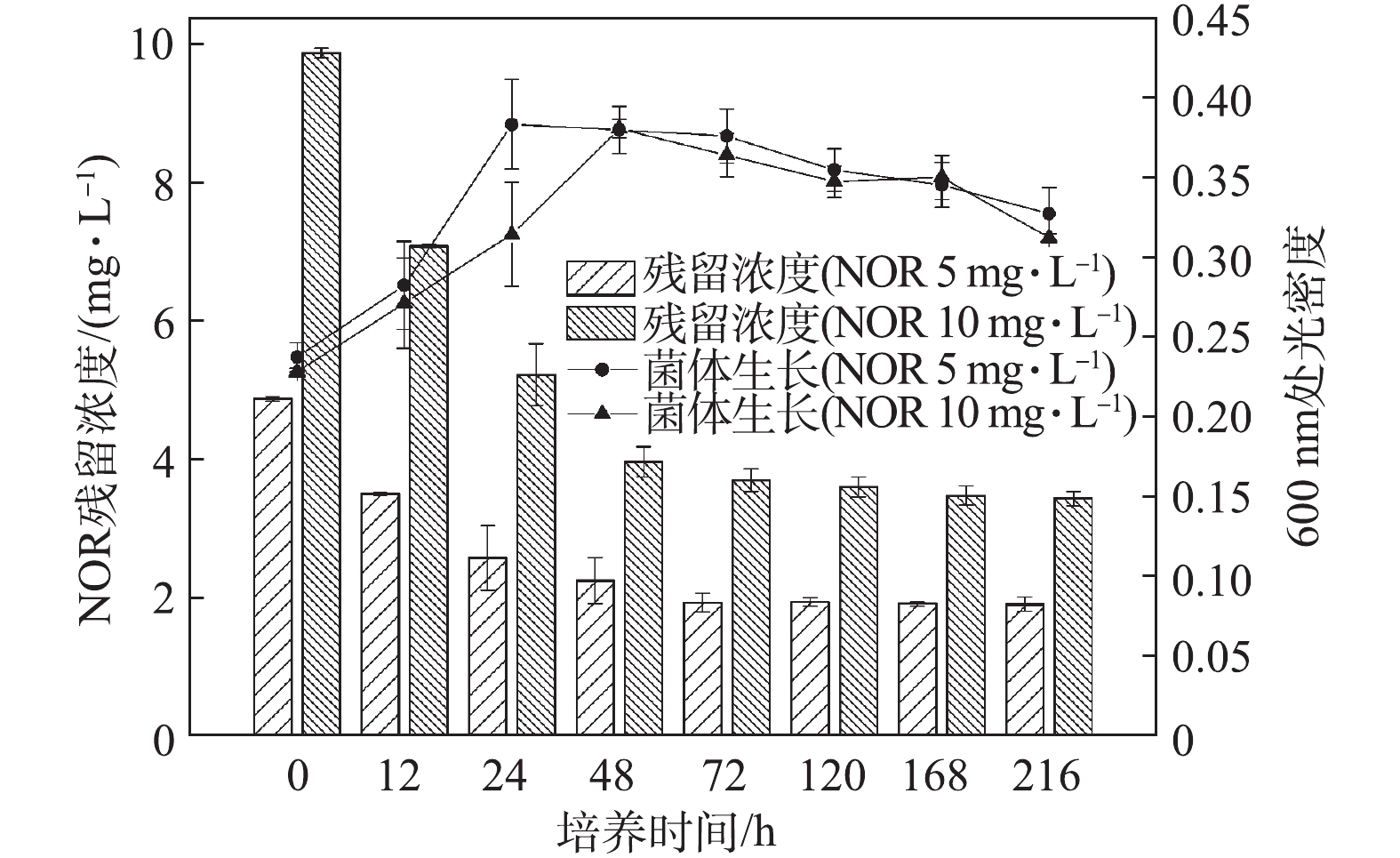
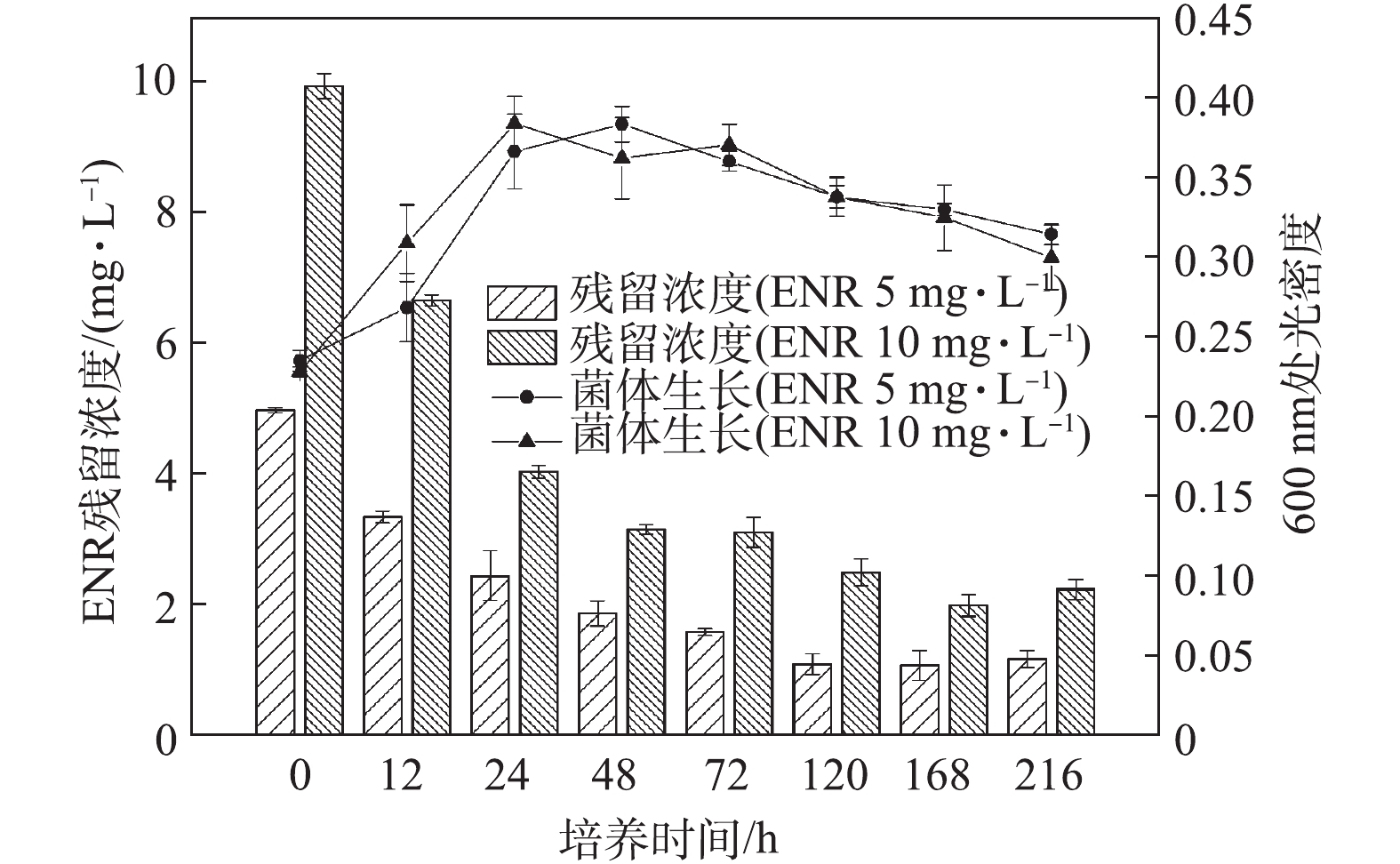
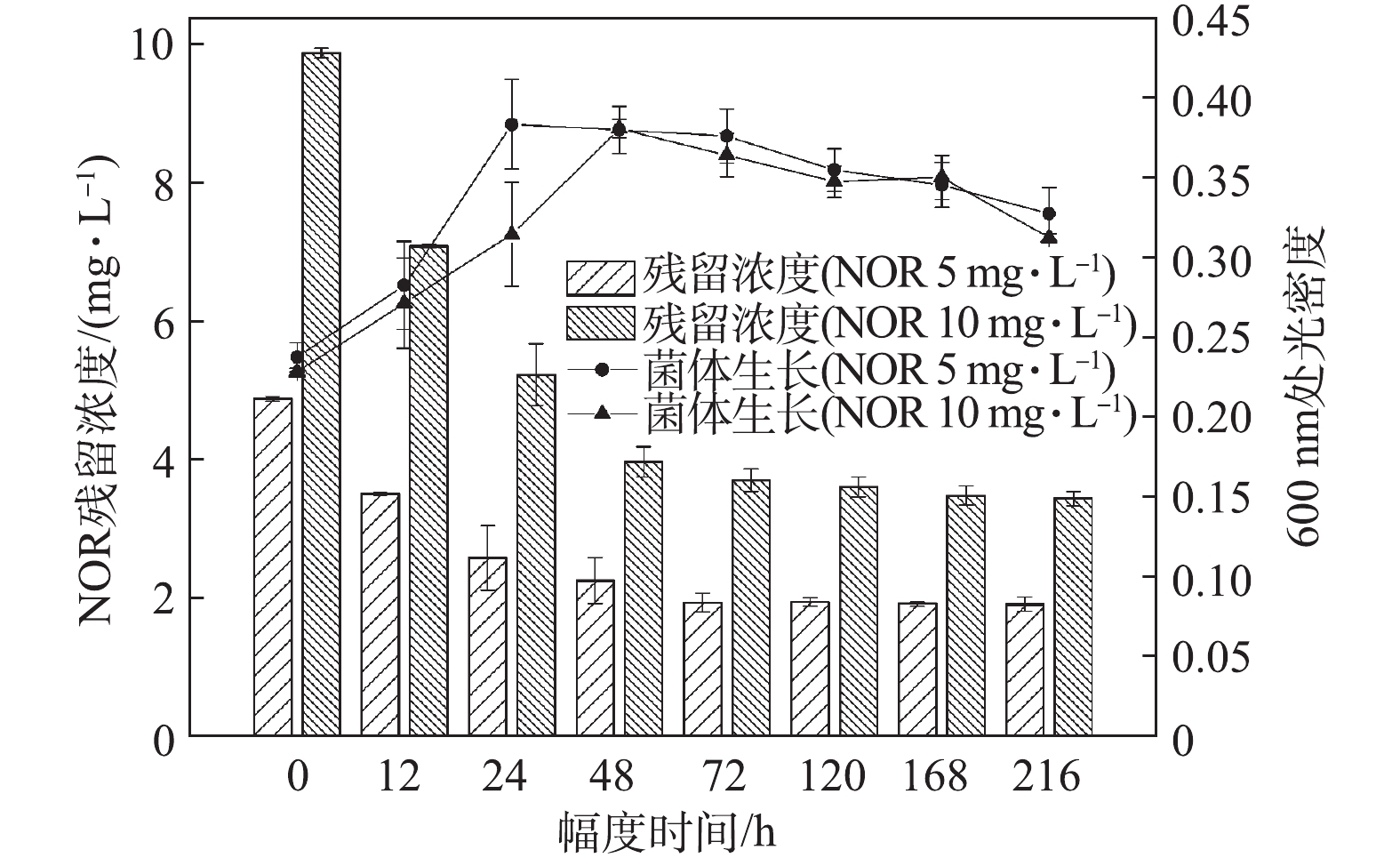
菌株C419对单一药物NOR的降解情况如图1所示。低剂量(5 mg·L−1)处理组中,菌株C419在初始24 h的生长速度快于高剂量(10 mg·L−1)处理组,但2组的最大生长量无明显差异。高、低剂量处理组在72 h内,对NOR的降解率分别为66%和60%;72 h后,NOR浓度基本不再降低。出现以上现象的主要原因可能是,在72 h后,菌体的生物量开始下降,菌体不再生长或者繁殖速率低于菌体的死亡速率,进而说明NOR降解主要发生在菌体生物量增长阶段。图2反映了菌株C419降解单一药物ENR的结果。低剂量(5 mg·L−1)处理组中,ENR的生物降解在初始48 h逐渐增加,与细菌的生长趋势一致;高剂量(10 mg·L−1)处理组也出现类似的现象。随着降解时间继续延长,虽然菌体生长量开始下降,但是ENR的降解还在继续,120 h后低剂量组达到最高降解率(75%),168 h后高浓度处理组达到最高降解率(80%)。由以上结果可知,ENR的降解不局限于菌体生物量的增长阶段,高浓度ENR的降解需要更长时间,且最终的降解率更高;同时,结果也表明菌株C419在氟喹诺酮药物高剂量处理组中表现出更强的降解能力。
对比NOR与ENR的降解,菌株C419对单一药物NOR或ENR(5 mg·L−1)的降解率分别为60%和75%,这说明菌株C419对ENR的降解效率更高,但是需要的时间更长。对比该菌株降解环丙沙星的效率(57%)[22],本研究中菌株C419对NOR与ENR的降解效率更高,这主要是因为不同类型氟喹诺酮类药物降解效果与药物分子结构的差异及其对微生物的毒性有关[23]。在3种氟喹诺酮药物(NOR、ENR、环丙沙星)中,环丙沙星对菌株C419的降解活性的抑制毒性最强,NOR次之,ENR最弱。该结果与AMORIM等[17]的研究结果一致。
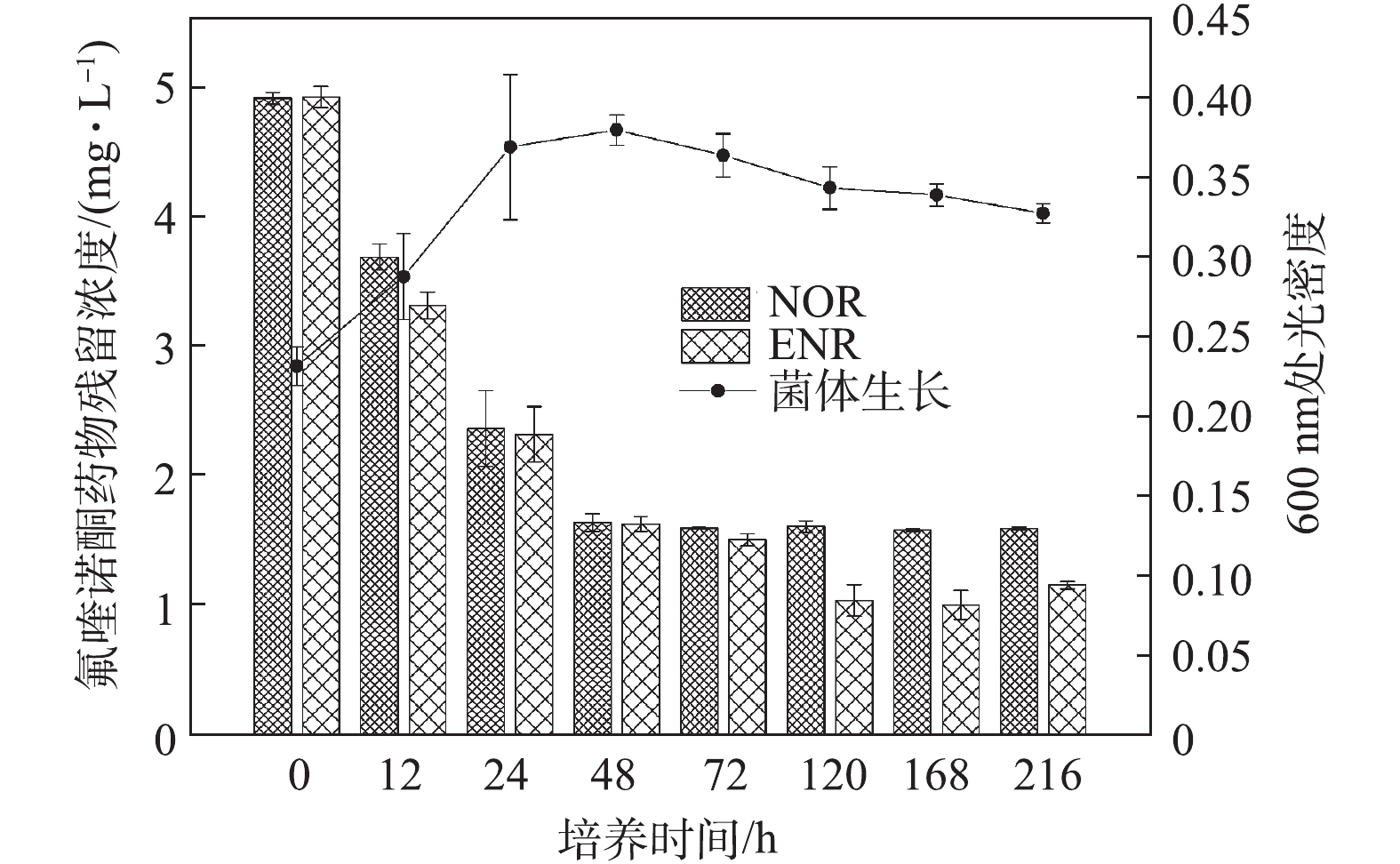
在环境基质中,氟喹诺酮类化合物通常是同时存在的,因此,评价菌株对混合药物的生物降解能力具有重要意义。图3反映了在2种氟喹诺酮类药物(NOR和ENR)混合体系中,菌株C419对他们的降解以及菌体生长的情况。在混合降解体系中,65%以上的NOR在培养48 h后被去除;ENR降解率则在培养120 h后达到最大值77%。与单一抗生素降解结果(5 mg·L−1 NOR:72 h降解60%;5 mg·L−1 ENR:120 h降解75%)相比,混合降解体系中各抗生素的降解效率更高,降解完成的时间更短。
综合以上结果可知,培养基中氟喹诺酮类药物种类越多或者抗生素浓度越高,菌株对抗生素的降解效率越高。其原因可能是,本研究中利用的菌株Thermus sp. C419是一种被证明具有合成淀粉酶、漆酶、锰过氧化物酶等耐热酶能力的嗜热菌[24]。根据已有研究[18, 25],这些酶广泛存在于真菌、细菌和植物中,并已被用于降解各种有机污染物,包括氟喹诺酮类药物,而且酶降解和修饰是抗生素药物等有机污染物生物降解的重要机制。由此推断,菌株C419对氟喹诺酮药物的降解是酶的作用结果。但是底物浓度过低将无法刺激微生物降解酶的产生,这会导致生物降解受到限制[26]。另有研究[27]指出,底物浓度的增加可以提高降解酶的活力。因此,高浓度或者种类多样的氟喹诺酮药物可以促进菌株C419合成更高酶活的降解酶,从而提高生物降解效率。另外,氟喹诺酮类药物的降解速率在菌体生长初期速度较快,这可能是由于乙酸钠作为外加碳源的促进作用导致的。随着乙酸钠的逐渐消耗,氟喹诺酮类药物的降解随之减弱。有研究[28]表明,易降解碳源的存在可以增加菌株生物量,并诱导参与化合物降解的特定酶的合成。而且,营养物质可以明显改善微生物生长和关键酶的活性,进一步增强微生物的共代谢作用[29]。
-
一级动力学模型通常被用于描述抗生素的降解行为[13, 17, 30],因此,本研究采用该模型拟合NOR和ENR的生物降解过程。由表1可以看出,各处理组的可决系数(R2)为0.815 6~0.950 4,这说明本实验数据与一级动力学模型拟合较好。不同浓度的NOR和ENR的降解速率常数(k)为0.015 6~0.023 2 h−1;2种药物混合体系中的降解速率常数分别为0.022 8 h−1和0.022 3 h−1;菌株C419在不同实验条件下对于这2种氟喹诺酮类药物的降解半衰期为29.9~44.4 h。对于单一药物的降解而言,低剂量处理组的NOR和ENR的半衰期都高于高剂量处理组,而降解速率常数均小于高剂量处理组,这说明高浓度下2种氟喹诺酮类药物的去除速率更快。此外,当菌株C419降解NOR和ENR混合液时,这2种药物的半衰期均低于单一药物降解时的半衰期,同时,降解速率常数均大于单一药物降解情况。
-
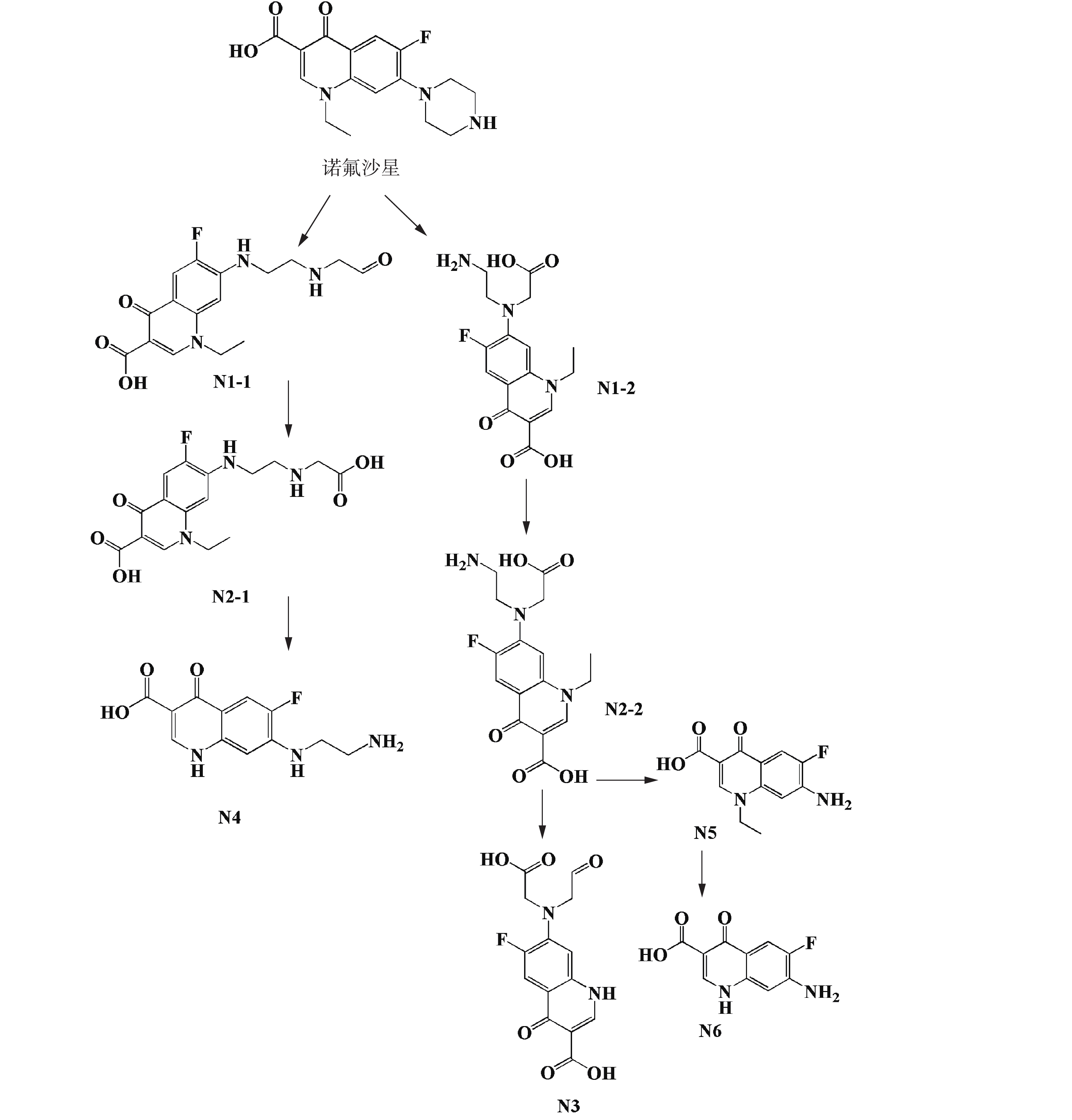
利用超高效液相色谱串联质谱(UPLC-MS/MS)对NOR和ENR的微生物降解代谢产物进行分析。相关产物的结构是基于参考文献中的方法[16-19]和EAWAG-BBD途径预测系统所得出,并通过产物离子分析进一步确认。NOR和ENR产物的质谱数据、相关的化学式和产物分子离子碎片损失结果见表2和表3。根据这些结果,提出了降解NOR和ENR可能的代谢途径(如图4和图5所示),氟喹诺酮类化合物有多条微生物降解途径,这与已有的研究结果[31]一致。
菌株C419降解NOR的代谢途径如图4所示。NOR的哌嗪环通过生物降解后由2个不同的途径开环并转化为2种不同的化合物(N1-1和N1-2)。产物N1-1和N1-2可被进一步氧化生成N2-1和N2-2[16]。在N2-1到N4的过程中,发生了一系列反应,包括脱羧反应和侧链基团的去除。虽然N1-1和N4之间存在中间产物,但由于这些化合物的不稳定性,因此,质谱检测并未获得相关信息。从N2-2到N4和N5的反应是通过胺氧化去除一个“R基团”的方式进行的。产物N6是由N5的吡啶环去除C2H5而获得的。上述代谢途径主要是基于EAWAG-BBD途径预测系统提出的,N1、N2、N3、N4和N6这5种降解产物首次在本研究中被提出。产物N1(C16H18FN3O4, m/z 336)和N5(C12H11FN2O3, m/z 251)曾在白腐真菌降解NOR的实验中[16]被检测出。另外,在菌株Labrys portucalensis F11 降解 NOR 生成的中间体中首次发现产物 N7(C14H16FN3O2, m/z 278) 和 N8(C16H19N3O4, m/z 318)。产物N9(C17H18FN3O4, m/z 348)降解木质素真菌的研究[18]表明其为代谢产物之一。
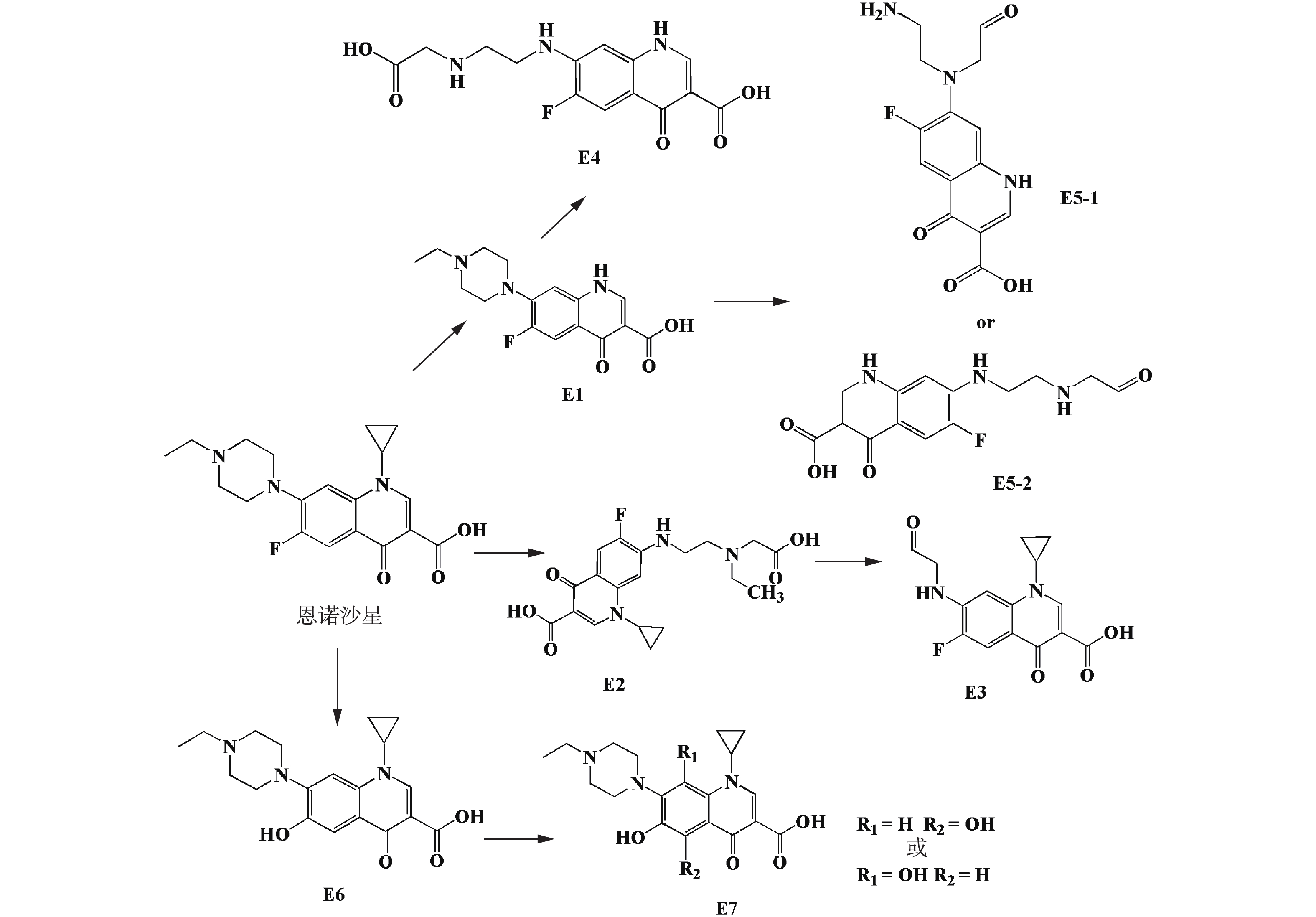
菌株C419降解ENR的代谢途径如图5所示。首先产物E1是通过氧化去除吡啶环上的环丙基而得到的,然后将E1的哌嗪环氧化开环生成产物E4和E5。E5有2种可能的结构,包括E5-1和E5-2。ENR也可能被氧化成E2,并通过氧化去除胺的“R基团”的方式进一步转化为E3。氟喹诺酮类化合物的转化都有相同的趋势,即哌嗪取代基始终是酶的作用位点[17]。产物E6(C19H23FN3O4, m/z 358)和E7(C19H23N3O5, m/z 374)也为褐腐菌降解ENR的产物[19]。产物E6可能是由羟基自由基对初始的ENR分子上氟位点的攻击所产生的,产物E7是E6进一步羟基化所生成的。由于ENR的代谢反应复杂,只有稳定的中间体才能被分离和鉴定出。
-
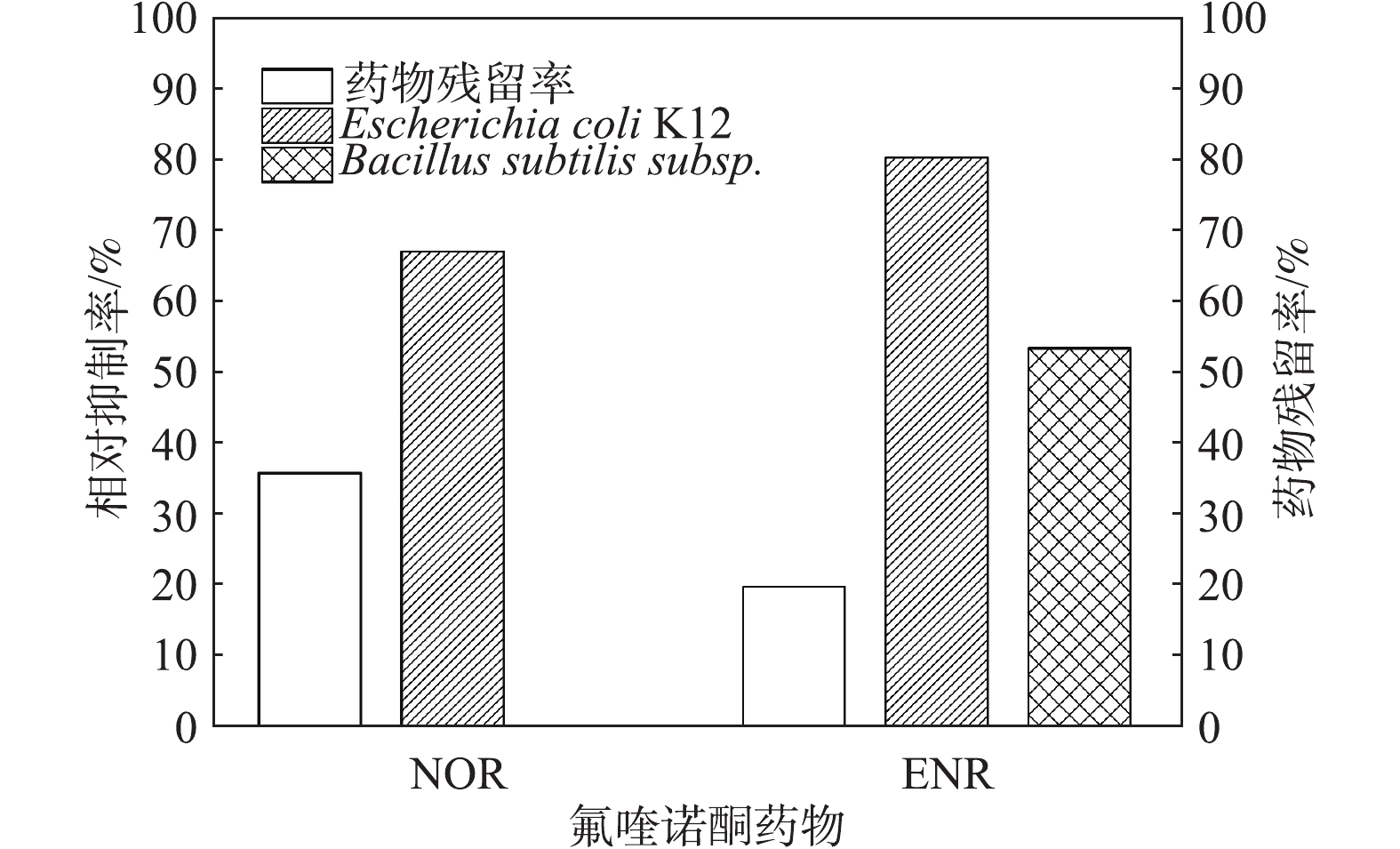
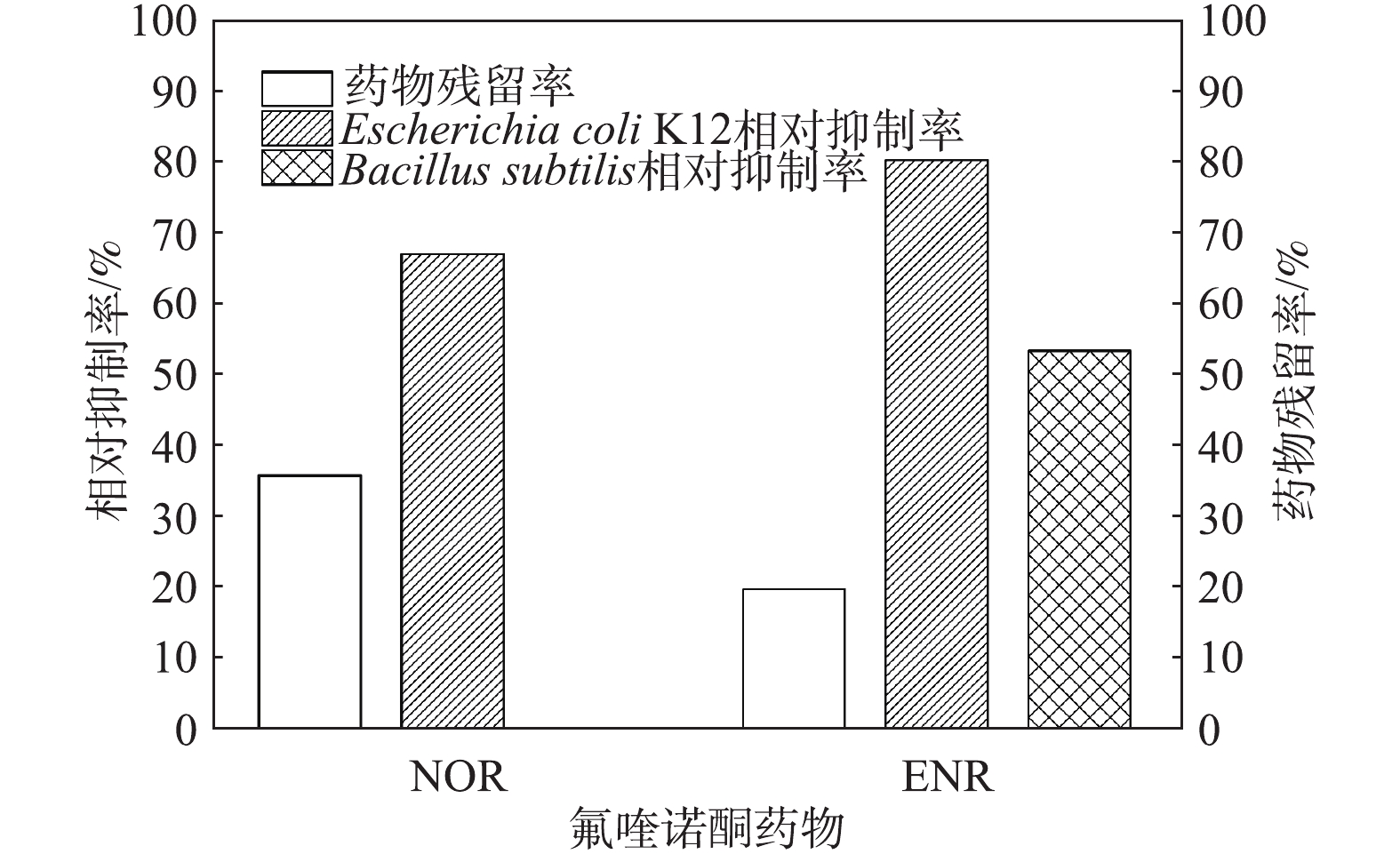
本研究利用革兰氏阴性菌-大肠杆菌K12和革兰氏阳性菌-枯草芽孢杆菌作为检测降解后药物的残留抗菌活性的目标微生物,通过比较氟喹诺酮类原药和生物降解后样品的抑菌圈大小得出样品对微生物的相对抑制率,来评估降解产物的残留抗菌活性。图6展示了生物降解后氟喹诺酮药物的残留浓度以及对枯草芽孢杆菌和大肠杆菌K12的相对抑制作用,结果表明,与母体化合物抑菌活性(100%)相比,2种氟喹诺酮类药物通过生物降解之后,其抑菌活性均有一定程度的降低。实验发现,NOR溶液和生物降解后的NOR样品对枯草芽孢杆菌均无抑制作用,因此,图6中没有显示相应结果。生物降解后的NOR样品对大肠杆菌K12的抑制作用与原始的NOR溶液相比降低了30%左右。而经生物降解后的ENR对大肠杆菌K12的抑制活性仍较高,与母体化合物相比仅降低20%,对枯草芽孢杆菌的抑制降低了45%。
由以上实验结果可知,生物降解之后的喹诺酮类药物仍然具有一定的抗菌活性。氟喹诺酮类药物的降解不彻底是其抗菌活性高的原因之一。氟喹诺酮类药物的抑菌活性主要在于哌嗪环和氟取代基[15]。虽然哌嗪环通常是降解酶的作用点,但本研究中其代谢产物结构复杂,一些活性基团未被完全清除,因此,其抗菌活性并未完全消失。同时,培养基中未被生物降解的母体化合物同样会造成较高的抗菌活性。ČVANČAROVA等[18]利用从环境中获得的多种微生物检测降解之后的氟喹诺酮药物的毒性,发现所有被测微生物均被高度抑制,这表明代谢产物仍然具有很高的抗菌活性。BECKER等[32]利用真菌漆酶去除废水中的抗生素(包括10种氟喹诺酮类抗生素),发现抗生素的毒性仅略有下降。因此,生物降解并不能完全去除氟喹诺酮类药物的毒性,一些物理化学降解方法也是如此[33]。由此推断,如果氟喹诺酮类未被彻底矿化,复杂的降解产物的残留抗菌活性仍不可低估。
2.1. NOR和ENR的生物降解
2.2. NOR和ENR的生物降解动力学分析
2.3. 生物降解产物的鉴定
2.4. 残留抗菌活性分析
-
1)菌株C419具有降解NOR和ENR的能力。C419单独降解NOR时,在高剂量和低剂量处理组中,NOR的去除率分别为66%和60%;C419单独降解ENR时,在高剂量和低剂量处理组中,ENR的去除率分别为80%和75%。
2)菌株C419降解NOR和ENR混合物时,对2种药物的去除率分别为65%和77%,均比降解单一药物时的去除率高。
3)NOR和ENR的生物降解遵循一级动力学模型。通过模型解析可以发现,培养基中氟喹诺酮类药物浓度越高或者混合降解时,药物的半衰期越短,降解效率越高。
4)利用UPLC-MS/MS确定了NOR和ENR可能的生物降解产物,并根据文献和代谢途径预测系统提出2种药物可能的代谢途径。另外,生物降解后的氟喹诺酮类药物抗菌活性减弱,但并未完全消失。因此,须进一步减少代谢产物的活性,以实现该菌株的工程应用。



 下载:
下载: