-
当前人工合成化学物被大量生产使用,给食品安全风险评估带来严峻挑战. 传统的化学物风险评估数据主要依赖于动物试验及体外试验等各类测试方法,但测试成本、试验周期、动物伦理以及“3R”原则的限制,导致了无法开展高通量、快速的风险评估. 随着毒理学测试方法的发展和风险评价策略的调整,2007年美国国家研究委员会提出“21 世纪的毒性试验:愿景与策略”(Toxicity Testing in the 21st Century: A Vision and a Strategy),强调了未来化学物毒性测试应借助计算毒理学和基于人源细胞、细胞系或细胞组分体外测试的组合方法[1],即新策略方法(New Approach Methodologies,NAMs),包括我国在内的很多国家和国际组织正在开展下一代风险评估技术研发[2 − 5],而NAMs是下一代风险评估的技术支持.
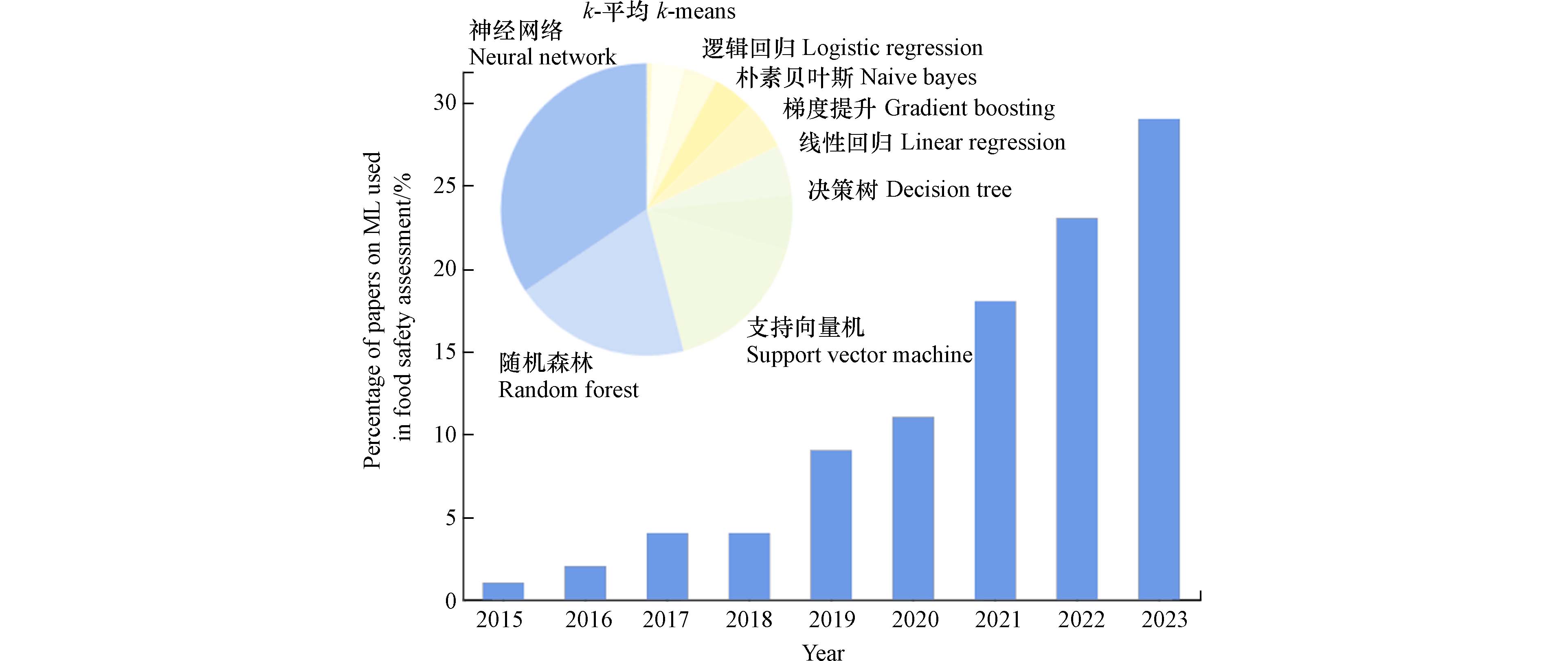
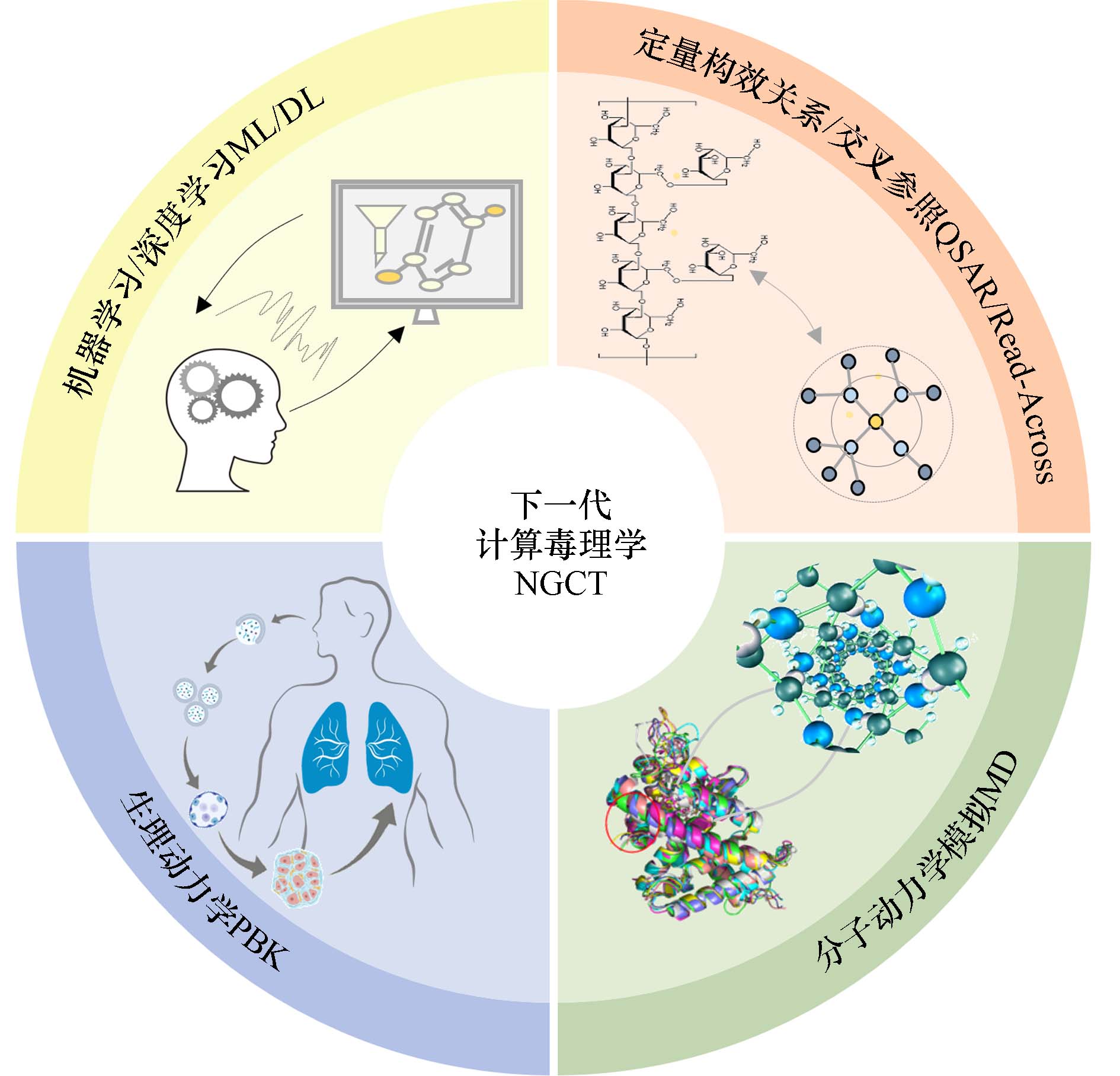
随着人工智能(Artificial Intelligence,AI)的快速发展,基于机器学习(Machine Learning,ML)和深度学习(Deep Learning,DL)的下一代计算毒理学(Next-Generation Computational Toxicology,NGCT)逐渐兴起(图1),为化学物的高通量测试提供技术支撑[6]. NGCT通过挖掘化学、毒理学、医学、多组学、系统生物学等在内的多模态大数据,对化学物的各类属性以及毒性等进行高通量预测. 目前NGCT被逐渐用于开发化学物风险评估的NAMs[7],美国食品和药品管理局关于建模和仿真的相关报告中重点强调了NAMs的应用,如基于生理的动力学模型(Physilogically Based Kinetics,PBK)建模、定量构效关系(Quantitative Structure-Activity Relationship,QSAR)分析、交叉参照分析(Read Across)用于风险评估的概率和统计建模等. 为进一步促进NGCT在化学物风险评估中的应用,本文介绍NGCT主要方法及其在食品安全风险评估中的应用,为化学物安全风险评估研究提供借鉴.
-
ML和DL在食品安全领域具有多种用途,常被用于预测食品安全风险. 随着AI的快速发展和大型数据库的建设,基于ML和DL的预测方法整合了多种NAMs,在化学物风险评估中发挥重要作用,近年关于机器学习的食品安全风险评估研究论文数量持续增加(图2).
ML和DL模型被广泛用于食品及食品相关化学物的安全性评估[8],可用于构建针对外源性化学物暴露的毒性筛查模型,包括贝叶斯网络[9]、神经网络[10,11]、支持向量机[12]和决策树[13,14]等算法常被用于食品安全风险分析及预警. 针对食品添加剂的潜在神经毒性,基于神经网络的ML模型建立了通用的食品有害因素分类评估分数,为大规模进行食品中具有神经毒性物质的风险评估提供有效手段[15]. 基于DL开发的DeepCarc模型运用碱基分类选择策略,实现了高准确率的化合物致癌风险优先级的筛选[16]. 通过亚临床和癌症前期阶段获得的数据构建ML模型,将有助于建立与饮食和儿童患传染性单核细胞增多症的关联,确定食品加工过程中产生的具有不良作用的化合物摄入的风险阈值[17]. 针对食品及食品添加剂的危害水平预测,将基于膳食暴露评估和暴露限值建立的风险评估模型,以及熵权-层次分析法计算食品中化学成分的风险值,进一步利用反向传播神经网络建立安全风险预警模型,预测食品的生产和加工过程是否存在安全风险,从而从源头上进行管控.
AI可结合电子鼻、电子舌、气相色谱-质谱、质谱代谢组学技术等检测方法,用于食品安全质量分析,特别是食品中特定成分或污染物的追踪[18]. 基于贝叶斯框架的稳定同位素混合模型通过建立同位素指纹、化学指纹等分析食品样品中的稳定同位素比例,实现食品中痕量污染物的溯源. 基于神经网络的稳定同位素混合模型,可以预测出不同品种、产地蜂蜜的地理来源,准确率达90%以上[19]. 针对食品中痕量或微量的化学残留分析,稳定同位素混合模型结合质谱分析技术可检测食物中的残留农药和未公开的添加剂[20],并对其空间分布进一步预测[21],这为食品添加剂的监管提供了技术支撑.
-
QSAR是基于化学物分子结构与生物活性/毒性建立的定量关系(图3). 数据集及分子特征描述的限制导致QSAR模型的应用域及泛化能力有限[22],而基于ML/DL的QSAR模型能够完全由数据驱动,数据量越大,模型的预测能力越强. 随着高通量试验的快速发展,大量的测试数据可用于训练深度QSAR模型[23].
随着AI的快速发展,围绕受体相关的关键分子靶点,利用化学物与受体分子交互作用的数据信息,可构建基于ML的QSAR模型[24]. 目前,基于ML/DL的QSAR模型已经用于食品-药物相互作用[25]、受体的结合活性[26-27]、肝脏毒性[28]及其他多种毒性终点的预测[29-30],在食品安全风险评估中显示出巨大应用前景.
ML/DL及QSAR模型常在数据量充足时表现出良好的预测性能,当化学物数据不足时,通常采用Read-Across作为数据缺口填补方法,用于预测目标物质的相同终点[31]. 基于ML的Read-Across工具能够估计所选结构特征的相关性[32],实现小型数据集上的高准确率预测效果. 将Read-Across集成到基于ML的QSAR模型中,通过数据融合为每种化学物质构建基于相似性的大型特征向量,结合QSAR和Read-Across的优点,使用多种相似性规则进行计算和加权,提高了模型的预测效率、可靠性和可解释性[33]. 如定量跨结构活性关系模型,使用最小二乘法消除了描述符之间的关联,且采用了ML方法对超参数进行优化,实现了高准确度的化学物心脏毒性预测[34]. 基于ML的Read-Across方法有助于解决相似性假说的不确定性,提高论据的充分性和可靠性、判断证据权衡结果是否明确已被广泛应用于化学物风险评估[35].
-
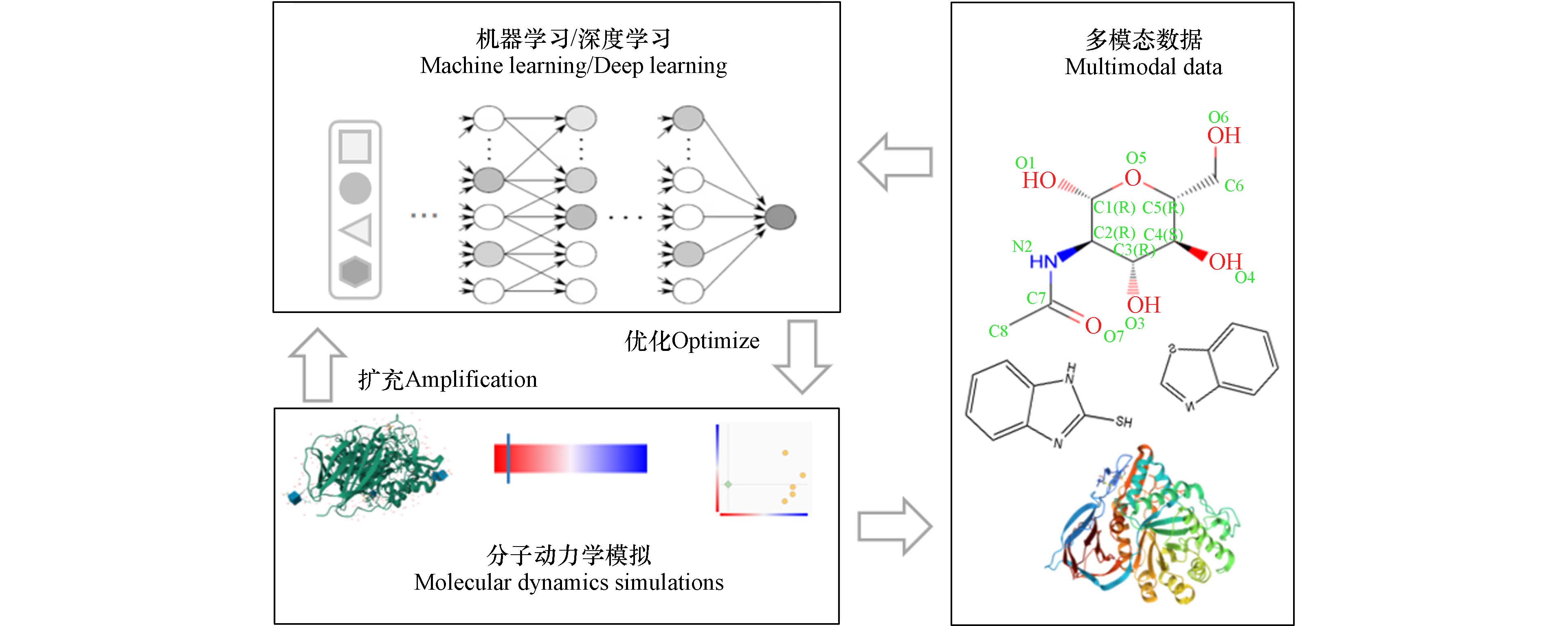
分子动力学(Molecular Dynamics,MD)模拟用于识别化学物质和生物大分子间的动态分子交互作用,为化学物风险评估提供更为全面的相互作用信息[36]. 分子模拟技术由于受到计算资源和算法的限制,精度和效率通常难以兼得,而将ML模型引入到分子动力学模拟中,能够在满足第一性原理精度的同时,计算速度媲美经验式模型,满足快速、低成本且高通量的预测要求(图4).
研究污染物与受体的交互作用有利于从蛋白质分子水平识别分子起始事件(Molecular initiation events,MIE),有利于深入了解MIE、关键事件(Key events,KE)和有害结局(Adverse outcome,AO)的关联,促进深入了解污染物毒性作用机制. 基于ML/DL的蛋白质-配体相互作用预测目前已显示出巨大的潜力[37]. 基于图学习的自适应机器能够适应多类数据集,比传统的分子动力学模拟展现出更好的预测性能[38];基于X射线结构中蛋白质-配体相互作用信息,利用六种图神经网络算法构建的模型准确预测了配体的亲和力[39]. 目前,AlphaFold 3模型在蛋白质-配体相互作用方面的预测性能皆优于其他传统分子模拟方法,能够预测包括蛋白质、核酸和小分子及多种复合物的结构,为化学物和受体相互作用研究提供更加准确全面的受体结构信息. 通过基于ML的原子间相互作用建模对食品成分及添加剂中的分子进行精确模拟,有利于预测由外源化合物暴露导致的内源性小分子的相互作用以及对人体健康的潜在影响[40].
随着大规模计算资源的持续增加,针对大量化学物开展MD模拟逐渐普及[41]. 基于分子交互作用信息,加强多模态数据扩充,构建融合MD重要特征的ML/DL预测模型,成为当前研究的新方法. 强化ML和MD领域之间的联合应用,构建集成式分子模拟及结构预测的综合性平台,将是人工智能与分子物理交叉领域荟萃方法的全新变革.
-
PBK模型可根据器官或组织内化学物及其代谢物浓度的时间变化,评估其在人体内的分布,模拟肝脏等代谢转化功能,提供毒物在体内的生物转化数据(图5). 在评估食品安全时,通过化学-生物相互作用的机理获得相关剂量-反应数据,借助PBK模型来预测人体体内的暴露量,最后进行风险表征.
PBK模型广泛用于预测特定人群通过食品摄入化学物质的暴露量,可预测化学物质在动物及人体内的分布和浓度,包括在血液、组织、乳汁等中的水平[42 − 44]. 欧洲食品安全局对食品中全氟烷基化合物对人类健康的风险进行评估时使用了PBK模型[42],能准确预测人类器官浓度-时间曲线、药代动力学行为和化学物的每日摄入剂量. PBK模型还能用于经由膳食摄入的化学物质到人体内浓度的推导[43],如预测乳汁中的化学物暴露,预测目标化合物的母体血浆暴露量和婴儿日摄入量[44].
传统的PBK模型受到动物试验伦理和低通量的限制,难以推广使用. 基于AI建模的下一代PBK模型克服了传统模型的技术瓶颈问题,可广泛应用于高通量化学物风险评估. 基于微生理系统的PBK模型用于皮肤-肝脏-甲状腺模型的开发,通过研究微流体循环连接的“器官”间的相互作用,预测几种典型的化学物在特定器官中的暴露[45],通过由皮肤和肝脏中的代谢及甲状腺激素表征化学物的生物效应. 与普通PBK模型相比,基于AI的PBK模型能够进一步预测器官水平的化学物质暴露,定性及定量描述化学物在体内的生物转化数据. 目前,通过使用计算机模拟、体外和人体生物监测数据对化学物的人体PBK模型进行细化和校准[46],极大促进从机制上理解食品安全风险,并提高预测结果的准确性.
定量危害表征依赖于对体外试验读数的解释,这需基于体外基准浓度将体外浓度-效应数据外推至体内剂量-效应数据,即定量体外-体内外推(Quantitative in vitro to in vivo Extrapolation,QIVIVE),通过体外组学数据预测体内毒性,区别于传统的动物实验,更加符合下一代风险评估的要求. 基于AI的PBK模型整合了关于化学物在人体或生物体中的吸收、分布、代谢和排泄的知识,成为体外-体内外推的有效工具,能够实现跨物种、途径和各种暴露情景预测[47]. 基于深度学习的Deep-PK模型能够识别影响化合物属性的关键分子区域,其在分子层面上的识别和预测性能远超常规的生理动力学模型[48]. 基于人工神经网络并融合化学物质文本描述和结构信息的多模态模型,能够实现同时预测不同物种、组织、测量方法和血液基质的组织-血液分配系数,为化学物内暴露评估提供了有效手段[49]. 将QIVIVE用于化学物的暴露,能够帮助深入理解化学物毒性的分子机制[50].
-
大量人工合成化学物被生产、投入使用并不断产生相关测试新数据,亟需为其开发通用的评估模型. 包括我国在内的很多国家和国际组织正在探索下一代风险评估方法,将整合有关分子系统生物学的信息纳入未来的风险评估范畴. NGCT将海量多模态数据有机整合,可实现对于化学物多种性质的高通量预测,揭示化学物质如何在生物体内引发不良健康效应,为食品安全风险评估提供深层次的机制性理解,能够推动化学物食品安全风险评估研究新范式的建立. 目前大语言模型(Large Language Model,LLM)在提取和整合高通量数据上表现突出,能够处理来自高通量筛选、组学、生物信息学和其他来源的大量复杂数据,其中垂直大模型能够专注于特定领域的数据处理和分析,通过自然语言处理技术更深层次地理解、整合并分析多模态数据,训练数据中的模式和关系,对新化学物的行为进行高精度预测,将促进化学物风险评估的变革性发展. 未来需要进一步结合LLM在自然语言处理、预测分析和决策制定方面的先进能力,提高风险评估的准确性和透明度.
下一代计算毒理学在食品安全风险评估中的应用
The application of next-generation computational toxicology in food safety risk assessment
-
摘要: 随着人工智能的迅速发展,基于机器学习和深度学习的下一代计算毒理学(Next-Generation Computational Toxicology,NGCT)逐渐兴起. NGCT通过整合化学、毒理学和系统生物学等多模态大数据,实现对化学物属性和毒性的高通量预测,克服了传统风险评估方法的局限性,推动了化学物的高通量风险评估. 本文总结了NGCT在融合分子交互特征、预测化学物属性和暴露数据、以及食品安全风险评估领域的应用. 鉴于大语言模型对研究新范式的推动,进一步展望了大语言模型在食品安全领域自然语言处理、预测分析和决策制定方面的潜力,并简述了基于NGCT的食品安全风险评估新范式.Abstract: With the rapid development of artificial intelligence, Next-Generation Computational Toxicology (NGCT) based on machine learning and deep learning is gradually emerging. NGCT integrates multimodal big data from chemistry, toxicology, and systems biology to achieve high-throughput predictions of chemical properties and toxicity, overcoming the limitations of traditional risk assessment methods and promoting high-throughput safety risk assessment of chemicals. This article summarizes the applications of NGCT in integrating molecular interaction features, predicting chemical properties and exposure data, and assessing food safety risks. Given the transformative impact of large language models (LLMs) on new research paradigms, the potential of LLMs in natural language processing, predictive analysis, and decision-making in the field of food safety is further discussed, and a new paradigm for food safety risk assessment based on NGCT is briefly described.
-
Key words:
- chemicals /
- food safety /
- computational toxicology /
- machine learning /
- high-throughput /
- risk assessment
-
当前人工合成化学物被大量生产使用,给食品安全风险评估带来严峻挑战. 传统的化学物风险评估数据主要依赖于动物试验及体外试验等各类测试方法,但测试成本、试验周期、动物伦理以及“3R”原则的限制,导致了无法开展高通量、快速的风险评估. 随着毒理学测试方法的发展和风险评价策略的调整,2007年美国国家研究委员会提出“21 世纪的毒性试验:愿景与策略”(Toxicity Testing in the 21st Century: A Vision and a Strategy),强调了未来化学物毒性测试应借助计算毒理学和基于人源细胞、细胞系或细胞组分体外测试的组合方法[1],即新策略方法(New Approach Methodologies,NAMs),包括我国在内的很多国家和国际组织正在开展下一代风险评估技术研发[2 − 5],而NAMs是下一代风险评估的技术支持.
随着人工智能(Artificial Intelligence,AI)的快速发展,基于机器学习(Machine Learning,ML)和深度学习(Deep Learning,DL)的下一代计算毒理学(Next-Generation Computational Toxicology,NGCT)逐渐兴起(图1),为化学物的高通量测试提供技术支撑[6]. NGCT通过挖掘化学、毒理学、医学、多组学、系统生物学等在内的多模态大数据,对化学物的各类属性以及毒性等进行高通量预测. 目前NGCT被逐渐用于开发化学物风险评估的NAMs[7],美国食品和药品管理局关于建模和仿真的相关报告中重点强调了NAMs的应用,如基于生理的动力学模型(Physilogically Based Kinetics,PBK)建模、定量构效关系(Quantitative Structure-Activity Relationship,QSAR)分析、交叉参照分析(Read Across)用于风险评估的概率和统计建模等. 为进一步促进NGCT在化学物风险评估中的应用,本文介绍NGCT主要方法及其在食品安全风险评估中的应用,为化学物安全风险评估研究提供借鉴.
1. 基于人工智能的安全性评估(AI-based safety assessment)
ML和DL在食品安全领域具有多种用途,常被用于预测食品安全风险. 随着AI的快速发展和大型数据库的建设,基于ML和DL的预测方法整合了多种NAMs,在化学物风险评估中发挥重要作用,近年关于机器学习的食品安全风险评估研究论文数量持续增加(图2).
ML和DL模型被广泛用于食品及食品相关化学物的安全性评估[8],可用于构建针对外源性化学物暴露的毒性筛查模型,包括贝叶斯网络[9]、神经网络[10,11]、支持向量机[12]和决策树[13,14]等算法常被用于食品安全风险分析及预警. 针对食品添加剂的潜在神经毒性,基于神经网络的ML模型建立了通用的食品有害因素分类评估分数,为大规模进行食品中具有神经毒性物质的风险评估提供有效手段[15]. 基于DL开发的DeepCarc模型运用碱基分类选择策略,实现了高准确率的化合物致癌风险优先级的筛选[16]. 通过亚临床和癌症前期阶段获得的数据构建ML模型,将有助于建立与饮食和儿童患传染性单核细胞增多症的关联,确定食品加工过程中产生的具有不良作用的化合物摄入的风险阈值[17]. 针对食品及食品添加剂的危害水平预测,将基于膳食暴露评估和暴露限值建立的风险评估模型,以及熵权-层次分析法计算食品中化学成分的风险值,进一步利用反向传播神经网络建立安全风险预警模型,预测食品的生产和加工过程是否存在安全风险,从而从源头上进行管控.
AI可结合电子鼻、电子舌、气相色谱-质谱、质谱代谢组学技术等检测方法,用于食品安全质量分析,特别是食品中特定成分或污染物的追踪[18]. 基于贝叶斯框架的稳定同位素混合模型通过建立同位素指纹、化学指纹等分析食品样品中的稳定同位素比例,实现食品中痕量污染物的溯源. 基于神经网络的稳定同位素混合模型,可以预测出不同品种、产地蜂蜜的地理来源,准确率达90%以上[19]. 针对食品中痕量或微量的化学残留分析,稳定同位素混合模型结合质谱分析技术可检测食物中的残留农药和未公开的添加剂[20],并对其空间分布进一步预测[21],这为食品添加剂的监管提供了技术支撑.
2. ML赋能的 QSAR /Read-Across研究(ML-aided QSAR /Read-Across research)
QSAR是基于化学物分子结构与生物活性/毒性建立的定量关系(图3). 数据集及分子特征描述的限制导致QSAR模型的应用域及泛化能力有限[22],而基于ML/DL的QSAR模型能够完全由数据驱动,数据量越大,模型的预测能力越强. 随着高通量试验的快速发展,大量的测试数据可用于训练深度QSAR模型[23].
随着AI的快速发展,围绕受体相关的关键分子靶点,利用化学物与受体分子交互作用的数据信息,可构建基于ML的QSAR模型[24]. 目前,基于ML/DL的QSAR模型已经用于食品-药物相互作用[25]、受体的结合活性[26-27]、肝脏毒性[28]及其他多种毒性终点的预测[29-30],在食品安全风险评估中显示出巨大应用前景.
ML/DL及QSAR模型常在数据量充足时表现出良好的预测性能,当化学物数据不足时,通常采用Read-Across作为数据缺口填补方法,用于预测目标物质的相同终点[31]. 基于ML的Read-Across工具能够估计所选结构特征的相关性[32],实现小型数据集上的高准确率预测效果. 将Read-Across集成到基于ML的QSAR模型中,通过数据融合为每种化学物质构建基于相似性的大型特征向量,结合QSAR和Read-Across的优点,使用多种相似性规则进行计算和加权,提高了模型的预测效率、可靠性和可解释性[33]. 如定量跨结构活性关系模型,使用最小二乘法消除了描述符之间的关联,且采用了ML方法对超参数进行优化,实现了高准确度的化学物心脏毒性预测[34]. 基于ML的Read-Across方法有助于解决相似性假说的不确定性,提高论据的充分性和可靠性、判断证据权衡结果是否明确已被广泛应用于化学物风险评估[35].
3. 融合分子交互作用特征的ML模型(ML models with molecular interaction features)
分子动力学(Molecular Dynamics,MD)模拟用于识别化学物质和生物大分子间的动态分子交互作用,为化学物风险评估提供更为全面的相互作用信息[36]. 分子模拟技术由于受到计算资源和算法的限制,精度和效率通常难以兼得,而将ML模型引入到分子动力学模拟中,能够在满足第一性原理精度的同时,计算速度媲美经验式模型,满足快速、低成本且高通量的预测要求(图4).
研究污染物与受体的交互作用有利于从蛋白质分子水平识别分子起始事件(Molecular initiation events,MIE),有利于深入了解MIE、关键事件(Key events,KE)和有害结局(Adverse outcome,AO)的关联,促进深入了解污染物毒性作用机制. 基于ML/DL的蛋白质-配体相互作用预测目前已显示出巨大的潜力[37]. 基于图学习的自适应机器能够适应多类数据集,比传统的分子动力学模拟展现出更好的预测性能[38];基于X射线结构中蛋白质-配体相互作用信息,利用六种图神经网络算法构建的模型准确预测了配体的亲和力[39]. 目前,AlphaFold 3模型在蛋白质-配体相互作用方面的预测性能皆优于其他传统分子模拟方法,能够预测包括蛋白质、核酸和小分子及多种复合物的结构,为化学物和受体相互作用研究提供更加准确全面的受体结构信息. 通过基于ML的原子间相互作用建模对食品成分及添加剂中的分子进行精确模拟,有利于预测由外源化合物暴露导致的内源性小分子的相互作用以及对人体健康的潜在影响[40].
随着大规模计算资源的持续增加,针对大量化学物开展MD模拟逐渐普及[41]. 基于分子交互作用信息,加强多模态数据扩充,构建融合MD重要特征的ML/DL预测模型,成为当前研究的新方法. 强化ML和MD领域之间的联合应用,构建集成式分子模拟及结构预测的综合性平台,将是人工智能与分子物理交叉领域荟萃方法的全新变革.
4. AI辅助的PBK模型(AI-aided PBK model)
PBK模型可根据器官或组织内化学物及其代谢物浓度的时间变化,评估其在人体内的分布,模拟肝脏等代谢转化功能,提供毒物在体内的生物转化数据(图5). 在评估食品安全时,通过化学-生物相互作用的机理获得相关剂量-反应数据,借助PBK模型来预测人体体内的暴露量,最后进行风险表征.
PBK模型广泛用于预测特定人群通过食品摄入化学物质的暴露量,可预测化学物质在动物及人体内的分布和浓度,包括在血液、组织、乳汁等中的水平[42 − 44]. 欧洲食品安全局对食品中全氟烷基化合物对人类健康的风险进行评估时使用了PBK模型[42],能准确预测人类器官浓度-时间曲线、药代动力学行为和化学物的每日摄入剂量. PBK模型还能用于经由膳食摄入的化学物质到人体内浓度的推导[43],如预测乳汁中的化学物暴露,预测目标化合物的母体血浆暴露量和婴儿日摄入量[44].
传统的PBK模型受到动物试验伦理和低通量的限制,难以推广使用. 基于AI建模的下一代PBK模型克服了传统模型的技术瓶颈问题,可广泛应用于高通量化学物风险评估. 基于微生理系统的PBK模型用于皮肤-肝脏-甲状腺模型的开发,通过研究微流体循环连接的“器官”间的相互作用,预测几种典型的化学物在特定器官中的暴露[45],通过由皮肤和肝脏中的代谢及甲状腺激素表征化学物的生物效应. 与普通PBK模型相比,基于AI的PBK模型能够进一步预测器官水平的化学物质暴露,定性及定量描述化学物在体内的生物转化数据. 目前,通过使用计算机模拟、体外和人体生物监测数据对化学物的人体PBK模型进行细化和校准[46],极大促进从机制上理解食品安全风险,并提高预测结果的准确性.
定量危害表征依赖于对体外试验读数的解释,这需基于体外基准浓度将体外浓度-效应数据外推至体内剂量-效应数据,即定量体外-体内外推(Quantitative in vitro to in vivo Extrapolation,QIVIVE),通过体外组学数据预测体内毒性,区别于传统的动物实验,更加符合下一代风险评估的要求. 基于AI的PBK模型整合了关于化学物在人体或生物体中的吸收、分布、代谢和排泄的知识,成为体外-体内外推的有效工具,能够实现跨物种、途径和各种暴露情景预测[47]. 基于深度学习的Deep-PK模型能够识别影响化合物属性的关键分子区域,其在分子层面上的识别和预测性能远超常规的生理动力学模型[48]. 基于人工神经网络并融合化学物质文本描述和结构信息的多模态模型,能够实现同时预测不同物种、组织、测量方法和血液基质的组织-血液分配系数,为化学物内暴露评估提供了有效手段[49]. 将QIVIVE用于化学物的暴露,能够帮助深入理解化学物毒性的分子机制[50].
5. 结论与展望(Conclusions and prospects)
大量人工合成化学物被生产、投入使用并不断产生相关测试新数据,亟需为其开发通用的评估模型. 包括我国在内的很多国家和国际组织正在探索下一代风险评估方法,将整合有关分子系统生物学的信息纳入未来的风险评估范畴. NGCT将海量多模态数据有机整合,可实现对于化学物多种性质的高通量预测,揭示化学物质如何在生物体内引发不良健康效应,为食品安全风险评估提供深层次的机制性理解,能够推动化学物食品安全风险评估研究新范式的建立. 目前大语言模型(Large Language Model,LLM)在提取和整合高通量数据上表现突出,能够处理来自高通量筛选、组学、生物信息学和其他来源的大量复杂数据,其中垂直大模型能够专注于特定领域的数据处理和分析,通过自然语言处理技术更深层次地理解、整合并分析多模态数据,训练数据中的模式和关系,对新化学物的行为进行高精度预测,将促进化学物风险评估的变革性发展. 未来需要进一步结合LLM在自然语言处理、预测分析和决策制定方面的先进能力,提高风险评估的准确性和透明度.
-
-
[1] Toxicity testing in the 21st century: a vision and a strategy[M]. Washington, DC: National Academies Press, 2007. [2] YANG D Y, YANG H, SHI M Y, et al. Advancing food safety risk assessment in China: Development of new approach methodologies (NAMs)[J]. Frontiers in Toxicology, 2023, 5: 1292373. doi: 10.3389/ftox.2023.1292373 [3] Supporting Individual Risk Assessment during COVID-19[M]. Washington D C : National Academies Press, 2022. [4] HAN P L, LI X H, YANG J Y, et al. Advancing toxicity predictions: A review on in vitro to in vivo extrapolation in next-generation risk assessment[J]. Environment & Health, 2024, 2(7): 499-513. [5] HSCHMEISSER S,MICCOLI A,von BERGEN M,et al. New approach methodologies in human regulatory toxicology–Not if,but how and when![J]. Environment International, 2023(178): 108082. [6] DENG X Y, CAO S H, HORN A L. Emerging applications of machine learning in food safety[J]. Annual Review of Food Science and Technology, 2021, 12: 513-538. doi: 10.1146/annurev-food-071720-024112 [7] ALEXANDER-WHITE C, BURY D, CRONIN M, et al. A 10-step framework for use of read-across (RAX) in next generation risk assessment (NGRA) for cosmetics safety assessment[J]. Regulatory Toxicology and Pharmacology, 2022, 129: 105094. doi: 10.1016/j.yrtph.2021.105094 [8] LIM K, PAN K, YU Z, et al. Pattern recognition based on machine learning identifies oil adulteration and edible oil mixtures[J]. Nature Communications, 2020, 11(1): 5353. doi: 10.1038/s41467-020-19137-6 [9] BOUZEMBRAK Y, MARVIN H J P. Impact of drivers of change, including climatic factors, on the occurrence of chemical food safety hazards in fruits and vegetables: A Bayesian Network approach[J]. Food Control, 2019, 97: 67-76. doi: 10.1016/j.foodcont.2018.10.021 [10] ZHENG C, SONG Y H, MA Y P. Public opinion prediction model of food safety events network based on BP neural network[J]. IOP Conference Series: Materials Science and Engineering, 2020, 719(1): 012078. doi: 10.1088/1757-899X/719/1/012078 [11] ZHANG R F, ZHOU L, ZUO M, et al. Prediction of dairy product quality risk based on extreme learning machine[C]. 2018 2nd International Conference on Data Science and Business Analytics (ICDSBA), Changsha, China, 2018: 448-456. [12] MA Y, J HOU Y Y, LIU Y S, et al. Research of food safety risk assessment methods based on big data[C]. IEEE International Conference on Big Data Analysis (ICBDA), Hangzhou, China, 2016: 1-5. [13] CHANG W T, Yeh Y P, WU H Y, et al. An automated alarm system for food safety by using electronic invoices[J]. PLoS One, 2020, 15(1): e0228035. doi: 10.1371/journal.pone.0228035 [14] SCHILTER B, BENIGNI R, BOOBIS A, et al. Establishing the level of safety concern for chemicals in food without the need for toxicity testing[J]. Regulatory Toxicology and Pharmacology, 2014, 68(2): 275-296. doi: 10.1016/j.yrtph.2013.08.018 [15] LI X L, CHENG W, YANG S F, et al. Establishment of a 13 genes-based molecular prediction score model to discriminate the neurotoxic potential of food relevant-chemicals[J]. Toxicology Letters, 2022, 355: 1-18. doi: 10.1016/j.toxlet.2021.10.013 [16] LI T, TONG W D, ROBERTS R, et al. DeepCarc: Deep learning-powered carcinogenicity prediction using model-level representation[J]. Frontiers in Artificial Intelligence, 2021, 4: 757780. doi: 10.3389/frai.2021.757780 [17] RUIZ-SAAVEDRA S, GARCÍA-GONZÁLEZ H, ARBOLEYA S, et al. Intestinal microbiota alterations by dietary exposure to chemicals from food cooking and processing. Application of data science for risk prediction[J]. Computational and Structural Biotechnology Journal, 2021, 19: 1081-1091. doi: 10.1016/j.csbj.2021.01.037 [18] WANG C C, LIANG Y C, WANG S S, et al. A machine learning-driven approach for prioritizing food contact chemicals of carcinogenic concern based on complementary in silico methods[J]. Food and Chemical Toxicology, 2022, 160: 112802. doi: 10.1016/j.fct.2021.112802 [19] 佘僧, 李熠, 宋洪波, 等. 稳定同位素技术在蜂蜜真实性溯源中的研究进展[J]. 食品工业科技, 2018, 39(17): 300-304,308. SHE S, LI Y, SONG H B, et al. Research progress of stable isotope ratio mass spectrometry for authenticity and traceability in honey[J]. Science and Technology of Food Industry, 2018, 39(17): 300-304,308 (in Chinese).
[20] 杜晓宁, 张鹏帅, 雷雯, 等. 稳定同位素技术在我国食品安全检测领域的应用进展[J]. 同位素, 2019, 32(3): 231-243. doi: 10.7538/tws.2019.32.03.0231 DU X N, ZHANG P S, LEI W, et al. Application of stable isotope technique in food safety field[J]. Journal of Isotopes, 2019, 32(3): 231-243 (in Chinese). doi: 10.7538/tws.2019.32.03.0231
[21] ERDÉLYI D, KERN Z, NYITRAI T, et al. Predicting the spatial distribution of stable isotopes in precipitation using a machine learning approach: A comparative assessment of random forest variants[J]. GEM - International Journal on Geomathematics, 2023, 14(1): 14. doi: 10.1007/s13137-023-00224-x [22] 王中钰, 陈景文, 傅志强, 等. QSAR模型应用域的表征方法[J]. 科学通报, 2022, 67(3): 255-266. doi: 10.1360/TB-2021-0406 WANG Z Y, CHEN J W, FU Z Q, et al. Characterization of applicability domains for QSAR models[J]. Chinese Science Bulletin, 2022, 67(3): 255-266 (in Chinese). doi: 10.1360/TB-2021-0406
[23] TROPSHA A, ISAYEV O, VARNEK A, et al. Integrating QSAR modelling and deep learning in drug discovery: The emergence of deep QSAR[J]. Nature Reviews. Drug Discovery, 2024, 23(2): 141-155. doi: 10.1038/s41573-023-00832-0 [24] BHHATARAI B, WILSON D M, PRICE P S, et al. Evaluation of OASIS QSAR models using ToxCast™ in vitro estrogen and androgen receptor binding data and application in an integrated endocrine screening approach[J]. Environmental Health Perspectives, 2016, 124(9): 1453-1461. doi: 10.1289/EHP184 [25] RYU J Y, KIM H U, LEE S Y. Deep learning improves prediction of drug-drug and drug-food interactions[J]. Proceedings of the National Academy of Sciences of the United States of America, 2018, 115(18): E4304-E4311. [26] RUSSO D P, ZORN K M, CLARK A M, et al. Comparing multiple machine learning algorithms and metrics for estrogen receptor binding prediction[J]. Molecular Pharmaceutics, 2018, 15(10): 4361-4370. doi: 10.1021/acs.molpharmaceut.8b00546 [27] IDAKWO G, THANGAPANDIAN S, LUTTRELL J 4th, et al. Deep learning-based structure-activity relationship modeling for multi-category toxicity classification: A case study of 10K Tox21 chemicals with high-throughput cell-based androgen receptor bioassay data[J]. Frontiers in Physiology, 2019, 10: 1044. doi: 10.3389/fphys.2019.01044 [28] WANG H, LIU R F, SCHYMAN P, et al. Deep neural network models for predicting chemically induced liver toxicity endpoints from transcriptomic responses[J]. Frontiers in Pharmacology, 2019, 10: 42. doi: 10.3389/fphar.2019.00042 [29] FERNANDEZ M, BAN F Q, WOO G, et al. Toxic colors: The use of deep learning for predicting toxicity of compounds merely from their graphic images[J]. Journal of Chemical Information and Modeling, 2018, 58(8): 1533-1543. doi: 10.1021/acs.jcim.8b00338 [30] GOH G B, SIEGEL C, VISHNU A, et al. Chemception: A deep neural network with minimal chemistry knowledge matches the performance of expert-developed QSAR/QSPR models[EB/OL]. 2017: 1706.06689. [31] van LEEUWEN K, SCHULTZ T W, HENRY T, et al. Using chemical categories to fill data gaps in hazard assessment[J]. SAR and QSAR in Environmental Research, 2009, 20(3/4): 207-220. [32] CHATTERJEE M, ROY K. Chemical similarity and machine learning-based approaches for the prediction of aquatic toxicity of binary and multicomponent pharmaceutical and pesticide mixtures against Aliivibrio fischeri[J]. Chemosphere, 2022, 308: 136463. doi: 10.1016/j.chemosphere.2022.136463 [33] HARTUNG T. Making big sense from big data in toxicology by read-across[J]. ALTEX, 2016, 33(2): 83-93. [34] BANERJEE A, ROY K. Machine-learning-based similarity meets traditional QSAR: “q-RASAR” for the enhancement of the external predictivity and detection of prediction confidence outliers in an hERG toxicity dataset[J]. Chemometrics and Intelligent Laboratory Systems, 2023, 237: 104829. doi: 10.1016/j.chemolab.2023.104829 [35] YANG C, RATHMAN J F, MOSTRAG A, et al. High throughput read-across for screening a large inventory of related structures by balancing artificial intelligence/machine learning and human knowledge[J]. Chemical Research in Toxicology, 2023, 36(7): 1081-1106. doi: 10.1021/acs.chemrestox.3c00062 [36] 潘柳萌, 吕翾, 庄树林. 分子动力学模拟在有机污染物毒性作用机制中的应用[J]. 科学通报, 2015, 60(19): 1781-1788. doi: 10.1360/N972015-00118 PAN L M, LÜ X, ZHUANG S L. The application of molecular dynamics simulations in mechanism of toxicity of organic contaminants[J]. Chinese Science Bulletin, 2015, 60(19): 1781-1788 (in Chinese). doi: 10.1360/N972015-00118
[37] ZHANG Y J, LI S Y, MENG K, et al. Machine learning for sequence and structure-based protein-ligand interaction prediction[J]. Journal of Chemical Information and Modeling, 2024, 64(5): 1456-1472. doi: 10.1021/acs.jcim.3c01841 [38] LI Y Q, HSIEH C Y, LU R Q, et al. An adaptive graph learning method for automated molecular interactions and properties predictions[J]. Nature Machine Intelligence, 2022, 4: 645-651. doi: 10.1038/s42256-022-00501-8 [39] MASTROPIETRO A, PASCULLI G, BAJORATH J. Learning characteristics of graph neural networks predicting protein–ligand affinities[J]. Nature Machine Intelligence, 2023, 5: 1427-1436. doi: 10.1038/s42256-023-00756-9 [40] 张家晨, 张良, 庄树林. 分子起始事件在计算毒理学中的研究展望[J]. 环境化学, 2021, 40(9): 2629-2632. doi: 10.7524/j.issn.0254-6108.2021032602 ZHANG J C, ZHANG L, ZHUANG S L. Perspective of molecular initiating events in computational toxicology[J]. Environmental Chemistry, 2021, 40(9): 2629-2632 (in Chinese). doi: 10.7524/j.issn.0254-6108.2021032602
[41] 谭皓月, 张荣, 陈钦畅, 等. 基于计算毒理的环境污染物-生物大分子的相互作用研究[J]. 科学通报, 2022, 67(35): 4180-4191. doi: 10.1360/TB-2022-0613 TAN H Y, ZHANG R, CHEN Q C, et al. Computational toxicology studies on the interactions between environmental contaminants and biomacromolecules[J]. Chinese Science Bulletin, 2022, 67(35): 4180-4191 (in Chinese). doi: 10.1360/TB-2022-0613
[42] PANEL ON CONTAMINANTS IN THE FOOD CHAIN (EFSA CONTAM PANEL) E F S A, SCHRENK D, BIGNAMI M, et al. Risk to human health related to the presence of perfluoroalkyl substances in food[J]. EFSA Journal. European Food Safety Authority, 2020, 18(9): e06223. [43] SHARMA R, KAMBLE S S, GUNASEKARAN A, et al. A systematic literature review on machine learning applications for sustainable agriculture supply chain performance[J]. Computers & Operations Research, 2020, 119: 104926. [44] PANSARI A, FAISAL M, JAMEI M, et al. Prediction of basic drug exposure in milk using a lactation model algorithm integrated within a physiologically based pharmacokinetic model[J]. Biopharmaceutics & Drug Disposition, 2022, 43(5): 201-212. [45] TAO T P, MASCHMEYER I, LeCLUYSE E L, et al. Development of a microphysiological skin-liver-thyroid Chip3 model and its application to evaluate the effects on thyroid hormones of topically applied cosmetic ingredients under consumer-relevant conditions[J]. Frontiers in Pharmacology, 2023, 14: 1076254. doi: 10.3389/fphar.2023.1076254 [46] McNALLY K, LOIZOU G. Refinement and calibration of a human PBPK model for the plasticiser, Di-(2-propylheptyl) phthalate (DPHP) using in silico, in vitro and human biomonitoring data[J]. Frontiers in Pharmacology, 2023, 14: 1111433. doi: 10.3389/fphar.2023.1111433 [47] NAJJAR A, PUNT A, WAMBAUGH J, et al. Towards best use and regulatory acceptance of generic physiologically based kinetic (PBK) models for in vitro-to-in vivo extrapolation (IVIVE) in chemical risk assessment[J]. Archives of Toxicology, 2022, 96(12): 3407-3419. doi: 10.1007/s00204-022-03356-5 [48] MYUNG Y, de SÁ A G C, ASCHER D B. Deep-PK: Deep learning for small molecule pharmacokinetic and toxicity prediction[J]. Nucleic Acids Research, 2024, 52(W1): W469-W475. doi: 10.1093/nar/gkae254 [49] ZHANG S Y, WANG Z Y, CHEN J W, et al. Multimodal model to predict tissue-to-blood partition coefficients of chemicals in mammals and fish[J]. Environmental Science & Technology, 2024, 58(4): 1944-1953. [50] ALGHARABLY E A, Di CONSIGLIO E, TESTAI E, et al. Prediction of in vivo prenatal chlorpyrifos exposure leading to developmental neurotoxicity in humans based on in vitro toxicity data by quantitative in vitro-in vivo extrapolation[J]. Frontiers in Pharmacology, 2023, 14: 1136174. doi: 10.3389/fphar.2023.1136174 -



 下载:
下载: