-
环境内分泌干扰物(EDCs)[1]是20世纪90年代以来引起广泛关注的一类新型化学物质.分为天然和人工合成两类,除铅、汞、砷等金属类外,其余均为有机化学物.按其用途可分为工业原材料、洗涤用品、化妆品、农药、塑料包装和儿童玩具等,因工业生产和使用被不断释放到空气、水体、土壤等环境介质中,可经呼吸空气、饮水、食物摄入及皮肤接触等途径进入人体,影响生物体内激素的合成、释放和代谢,甚至可与激素竞争结合相应受体,或者影响激素受体上下游的调控基因,干扰激素正常的调节功能,造成内分泌系统的紊乱,从而引发各种疾病,对人类健康产生潜在的危害和不利影响[2].
已有大量研究报道EDCs可产生类雌激素效应[3],如邻苯二甲酸脂类、多氯联苯类、双酚类等化合物.这些EDCs在环境浓度下发挥着与天然雌激素类似的生物学活性,从而影响女性的生殖健康[4].已知的不利健康危害包括:月经周期变化、子宫内膜异位症、子宫肌瘤、多囊卵巢综合征、不孕不育、乳腺癌、子宫内膜癌及卵巢癌.此外,近年来的流行病学调查 报告进一步证实女性雌激素敏感癌症发病率与环境中EDCs的浓度存在密切相关性.
EDCs诱导产生的类雌激素效应主要通过经典的核雌激素受体ERα与ERβ介导[5].随着结构生物学研究的深入,有报道证实存在膜雌激素受体(GPER),属于7-跨膜G蛋白偶联受体(GPCR)家族[6]的一员,作为雌激素信号的靶蛋白,参与了雌激素在生殖、神经、内分泌、免疫和心血管系统中的介导作用,对于包括癌症在内的一系列疾病,GPER正成为一种新的治疗靶点和预后指标[7-9].G蛋白偶联雌激素受体(GPER)最初被称为GPR30[10],其结构未被结晶实验确认,激动和拮抗的分子机制及其结构特征的研究仍处在初步阶段.不同于经典的核雌激素受体亚型,GPER被认为是介导快速细胞信号的媒介[11],参与介导了雌激素快速非基因组效应[12].内源性化合物,如雌激素可与细胞膜上的GPER结合,激活快速细胞效应反应[13],包括环磷酸腺苷的产生、细胞内钙离子的动员、多种激酶的激活,如细胞外信号调节激酶、肌醇磷脂3激酶、离子通道以及内皮型一氧化氮合酶等途径.影响下游效应分子在相应的靶组织中发挥其生物学效应[14],从而导致细胞的增值与分化.类似地,环境污染物也可模拟内源性物质结合GPER[15].如BPA可在低浓度下激活GPER,产生较强的雌激素效应[16].
因此,构建快速筛查环境污染物是否结合GPER的分类预测模型,可为理论评估化合物健康风险与毒性效用提供重要的依据.虽然针对环境污染物的毒性预测,已有报道显示机器学习算法可表现出良好的分类预测性能 [17-20],但是结合GPER的小分子却未有可用的分子数据库和已知的预测模型,这限制了构建针对GPER分类预测模型的发展.为了解决这一问题,本研究系统地总结了已报道的有机小分子结合GPER的数据,并进一步评测了随机森林(random forest,RF)、支持向量机(support vector machine,SVM)、神经网络(neural network,ANN)、K-最近邻(K-nearest neighbour,KNN)、朴素贝叶斯(naive bayes,NB)、逻辑回归(logistic,LG)等6种典型机器学习算法的分类预测性能,其中基于RF算法构建的分类模型展示出了优秀的分类预测表现.
-
从文献中查询了130种对GPER检测结合能力的化合物[6, 11, 16, 21-38],分别包括7种双酚类化合物、30种多溴联苯类化合物、18种类固醇类化合物、15种植物激素、30种吲哚及其衍生物、10种农药杀虫剂以及20种GPER选择性配体化合物.其中具有结合能力的化合物有91种;不具有结合能力的化合物有39种.将所有化合物以4∶1的比例随机分成训练集和测试集,其中训练集具有结合能力的有73种,不具有结合能力的有31种;测试集具有结合能力的有18种,不具有结合能力的有8种.利用训练集构建分类预测模型,通过测试集定性评估模型的预测能力.
-
利用软件ChemDraw程序包绘制测试化合物相应的2D分子结构,随后通过Chem 3D程序包转化为相应的3D分子结构,并对其进行能量最小化优化处理.基于优化后的SDF格式的分子结构文件,通过 PaDEL软件计算得到1538个1D和2D分子描述符.根据以下两个原则进行初步的描述符筛选:(1)剔除常数和至少有一个缺失值的描述符;(2)剔除描述符之间相关系数大于0.9的描述符,最终得到369个分子描述符特征.随后利用RF算法和递归特征删减法的描述符重要性评价,将重要性前五位的描述符保留,作为构建机器学习分类预测模型的特征值.
-
为了系统地比较不同机器学习算法的表现,筛选最优算法构建GPER配体分类预测模型,我们选择了6种机器学习算法进行模型的构建[17],分别为:(1)RF算法,是一种集成分类器,它将大量的决策树拟合到一个数据集中,然后将所有树的预测组合起来;(2)SVM算法,通过核函数将输入向量映射到高维特征空间,优化特征空间内的线性划分,构造出具有最大边距的超平面来分离不同类别的数据;(3)ANN算法,采用最常见的前馈反向传播算法,其主要思想是:输入学习样本,使用反向传播算法对网络的权值和偏差进行反复的调整训练,使输出的向量与期望向量尽可能地接近;(4)KNN算法,考虑到数据中的所有情况,并根据局部邻域来获得分类预测.局部领域的大小由参数k决定,即k个最近邻数据点,而类别则由K-最近邻邻居的多数票决定;(5)NB算法,是基于现有的特征并假设特征独立,判断分类类别的概率,进行分类;(6)LG算法,主要研究二分类的响应变量与相应特征值之间的相互关系,并建立相应的预测模型.
所有的数据处理和模型建立都是基于统计编程环境R(版本4.0.2)实现.分别使用randomForest和caret程序包执行特征选择.使用randomForest、kernlab、nnet、kknn、e1071程序包执行RF、SVM、ANN、KNN、NB和LG算法的分类模型构建.
对于分类模型的评估[17],将模型输出的预测分类与实际结果相比,利用混淆矩阵产生真阳性(true positive,TP)、真阴性(true negative,TN)、假阳性(false positive,FP)、假阴性(false negative,FN),并计算相应指标敏感性(Sensitivity,Sn)、特异性(Specificity,Sp)、精确度(Accuracy,Q)以及马修斯相关系数(matthews correlation coefficient,MCC),相应计算公式如下:
敏感性S、特异性Sp、精确度Q以及马修斯相关系数MCC的值越高模型评价越好,精确度在0.9以上为预测结果较好.
而受试者工作特征曲线(receiver operator characteristic curve,ROC)则是另外一个评价二分类模型的重要指标.使用pROC程序包生成ROC曲线并计算每个分类模型的训练集和预测集的AUC值.此外,我们也采用10折交叉验证,来评估模型的精度.最终采用AMBIT Discover软件计算训练集和测试集中化合物的欧几里德距离,评价基于分子描述符的预测模型的应用域.
-
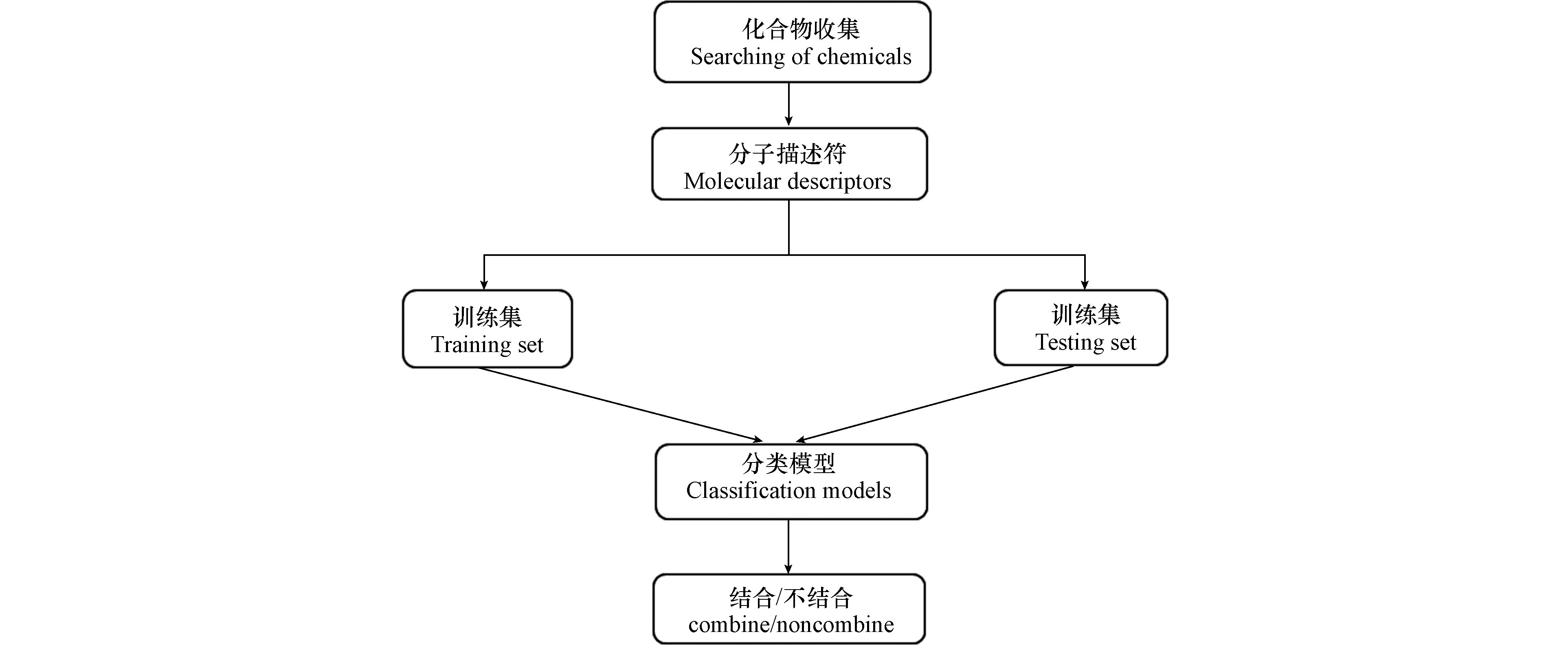
采用10折交叉验证来评估模型的可靠性和预测性能,图1显示了整个分类模型构建的流程.
-
联合RF算法的重要性评价和caret程序包的递归特征删减法进行描述符选取,得到如表1所示的5个最重要的描述符作为模型特征值.
其中,自变量GATS4c、GATS4s、AATSC6i和MATS2i为基于 Geary自相关指数、Broto-Moreau自相关指数以及Moran自相关指数的描述符.自相关指数反应原子性质沿拓扑结构的分布,其中GATS4c和 GATS4s描述符反映了化合物的静电性质对于结合的影响.AATSC6i和MATS2i描述符代表了化合物第一电离能的影响,ETA_Eta表示扩展拓扑化学原子指数对于分子结合能力的影响.
-
对于不同机器学习算法,采用caret程序包的格点搜索法进行超参数的优化,提高分类模型的统计学评价标准.
对于RF模型,通过调整参数trees和mtry的值,改变森林“树”的数量,从而达到对模型的优化效果.trees表示森林“树”的数量,mtry表示每次迭代变量抽样的数量.最终选定RF模型的参数为ntree=500、mtry=5,此时模型预测精度最优.
对于SVM模型,采用高斯RBF核函数进行训练.参数gamma决定了数据映射到新的特征空间后的分布;而参数cost表示为惩罚因子,即对误差的容忍度.最终选定SVM模型的参数为gamma=0.35、cost=10.
针对人工神经网络,分别对size、decay和maxit 等3个参数进行优化,其中size代表隐藏层神经元数,decay代表输入权重的修正参数、maxit代表最大迭代次数.最终选择ANN模型的参数为size=17、decay=0.01、maxit=1000.
对于K-最近邻,通过对邻居数量k的优化,从而增加模型的精度.最终选择5个邻居数的KNN算法建立模型,精确度较高.
对于NB模型,为避免由于样本数据较少而导致整个数据零概率问题的出现,使用拉普拉斯技术做平滑处理,避免零概率问题.
对于LG模型,我们使用逐步回归的方法进行变量重要性的筛选,得到最优模型.
-
通过对模型参数的优化,最终得到最优模型预测结果,如表2所示.
根据结果,表3列举了模型RF、SVM、ANN和KNN的4组预测结果较好的数据,其中Y表示分子结合GPER、N表示不结合.可以看到,测试集的α-E2、训练集的2'-OH-BDE-003和BPF被四个模型全部预测错误.推测原因为α-E2和BPF在结构上与具有结合效应的分子E2和BPA有相似之处,2'-OH-BDE-003在结构上与不具有结合效应的分子BDE-003有相似之处,由此造成了本研究中的所有分类模型都不能对其进行正确的分类.
依据表4,计算模型评价指标Sn、Sp、Q和MCC值,如下所示.可以看到RF的精确度最高,训练集达到99%,测试集达到88.5%,敏感性和特异性也较高,说明RF的预测效果优秀.SVM、ANN、KNN的精度次之,训练集精确度分别达到97.1%、95.2%、95.2%;测试集精确度分别达到88.5%、84.6%、84.6%,敏感性和特异性也较高.而NB和LG的预测结果较差,训练集精度分别为81.7%和79.8%;测试集精度分别为84.6%和88.5%,由此得到最优模型为RF.同时其它模型的统计学评价指标(Sn、Sp、Q和MCC)也显示为RF为最优的分类预测模型.
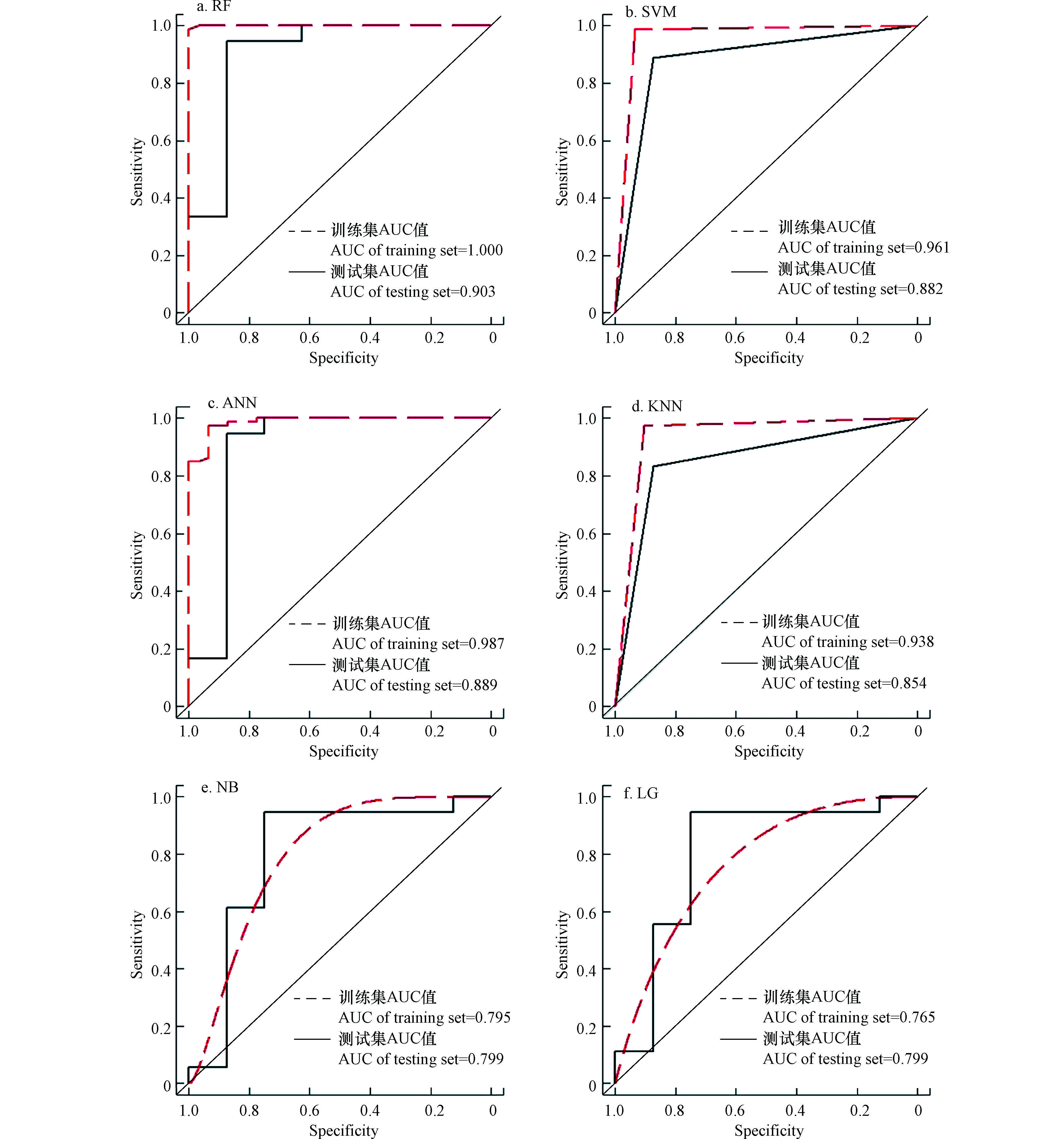
此外通过受试者工作特征(ROC)曲线以及AUC曲下面积对模型评价进行进一步说明.AUC曲下面积越接近1模型性能越高.由图2可知,RF训练集和测试集的AUC值均在0.9以上,说明模型性能最优;SVM、ANN、KNN次之,训练集AUC值分别达到0.961、0.987、0.938,测试集AUC分别达到0.882、0.889、0.854,模型性能较好;NB和LG的训练集AUC值分别为0.795和0.765,测试集AUC均为0.799,说明模型预测性能较差.
最后,通过10折交叉验证来计算模型的精度,其中RF、SVM、ANN、KNN精度分别为81%、81%、81%、80%,而NB和LG的精度较低,分别为75%和72%,说明RF和SVM模型的精度较高,这和前面模型性能评估得出的结论是一致的.
-
本文使用基于欧几里得距离的标准来探索化合物是否适用于所建模型,利用训练集计算可知化合物中最大的欧几里得距离为0.88(截断值),而计算得到的测试集中所有化合物的欧几里得距离均小于0.88,表明测试集化合物均适用于预测模型.这也为测试活性未知的化合物是否适用于所建模型提供了评价标准.
-
目前对于外源性化合物对膜雌激素受体GPER的干扰效应研究方兴未艾,仍有大量潜在的化合物有待验证.因此,在我们的研究中,搜集了130种已知的化合物结合GPER的情况,评价了RF、SVM、ANN、KNN、NB、LG的6种机器学习算法构建的分类预测模型的表现.通过特征选择、模型优化以及模型评价,结果显示RF为最优模型,为评价环境污染物是否通过结合GPER而产生内分泌干扰效应提供了快速筛查的预测模型与理论支撑.
基于膜雌激素受体(GPER)结合化合物能力的分类预测模型
Classification prediction model based on GPER binding ability of membrane estrogen receptor
-
摘要: 近年来,计算毒理学的方法被广泛应用于潜在的环境内分泌干扰物(EDCs)的筛选.膜雌激素受体(GPER),作为一种可以快速响应内源性配体雌激素的关键靶蛋白,调控其介导的多项生理学功能.但是针对GPER的化合物毒性预测模型仍未见报道.因此,本研究收集了130个化合物对GPER的结合活性数据,主要包括双酚类、多溴联苯类以及农药杀虫剂类环境污染物.利用随机森林(RF)、支持向量机(SVM)、人工神经网络(ANN)、K最近邻(KNN)、朴素贝叶斯(NB)以及逻辑回归(LG)等6种机器学习算法构建二分类模型.结果显示,所有被测试算法的测试集准确率均达到85%以上,其中SVM、RF、ANN、KNN等4种算法的训练集准确率高于93%,10折交叉验证准确率高于80%,说明得到的模型具有优秀的分类预测性能.因此,本研究基于机器学习算法构建的分类模型,可以用来快速、准确地预测环境污染物是否通过结合GPER产生内分泌干扰效应.为评估环境污染物的潜在健康风险提供了理论依据.Abstract: In recent years, computational toxicology has been widely applied to the screening of potential environmental endocrine disruptors(EDCs).Membrane estrogen receptor(GPER), as a key target protein that can rapidly respond to endogenous ligand estrogen, regulates its mediated multiple physiological functions. However, the prediction model of compound toxicity for GPER has not been reported. Therefore, the binding activity data of 130 compounds against GPER were collected in this study, mainly including bisphenols, polybrominated biphenyls and environmental pollutants like pesticides and insecticides.Six machine learning algorithms, including random forest (RF), support vector machine(SVM), artificial neural network(ANN), K-nearest neighbor(KNN), Naive Bayes(NB)and logistic regression(LG), were used to construct the dichotomous model.The results showed that the test set accuracy of all the algorithms tested reached more than 85%, among which the training set accuracy of SVM、RF、ANN and KNN algorithm was 93% higher than that of the four algorithms, and the 10-fold cross validation accuracy was 80% higher than that of the four algorithms, indicating that the model obtained had excellent classification prediction performance. Therefore, the classification model built based on machine learning algorithm in this study can be used to quickly and accurately predict whether environmental pollutants will produce endocrine disrupting effects by combining with GPER.It provides a theoretical basis for evaluating the potential health risks of environmental pollutants.
-
随着矿区农业、采矿业以及化工生产业的不断发展,污染物不断地排放,导致矿区地区浅层地下水不同程度的污染[1-3]。监测显示,某矿区地下水中超标的污染物有重金属Cr、阴离子
SO2−4 、F−等。Cr(Ⅵ)在环境中呈流动态,毒性很高,很容易穿透细胞壁,在细胞代谢过程中,可引起DNA氧化和非氧化2种形式的损坏,从而导致突变和染色体断裂,影响DNA的自然复制和转录,并能引起突变,主要导致肝细胞功能、肾脏和肺部的癌变[4-6];长期饮用高氟水,轻者牙齿产生斑釉、关节疼痛,重者会影响骨骼发育,甚至丧失劳动力[7-9]。目前,我国有400余个城市以地下水为供水水源[10],有些城市地下水甚至成为唯一供水水源。地下水关乎人民健康,一旦受到污染,造成的危害将无法估量。因此,寻找合适的污染地下水治理技术显得尤为重要。硫酸盐还原菌(SRB)价格低廉,是去除重金属离子非常有效的方法之一。董慧等[11]利用SRB去除矿山废水中污染物,在进水pH为3.0、水温为26~27 ℃、进水Fe2+的质量度低于450 mg·L−1、mCOD/m硫酸根离子>2.0的条件下,
SO2−4 平均去除率在80%以上,且对水中耗氧有机污染物(以COD计)有较好的去除效果,对重金属平均去除率在99%以上。董艳荣等[12]研究了SRB分离及处理煤矿酸性废水工艺,结果表明,在接种量为10%、接种时间为5 d条件下,对煤矿酸性废水中SO2−4 和Fe2+的去除率分别为74.71%和99.18%。SRB虽然在处理污染水方面具有一定的优势,但SRB需要充足碳源,且易受外界因素干扰,单独作用效果差。而SRB固定化技术是将其高度密集于一个有限的空间内,使其保持一定活性,具有处理污水效果好、利于固液分离、可重复利用、回收方便和抗重金属离子抑制能力强等优点[13-14]。安文博等[15]利用生铁屑固定SRB的实验表明,SRB颗粒能够抵抗pH=4的酸溶液,并在碱、盐溶液中能够保持较好稳定性,对Mn2+的吸附容量符合Freundlich等温吸附方程(R2=0.988 68,1/n=0.489 6),吸附动力学符合Elovich动力学模型(R2=0.996 4)。有机-无机杂化材料是一种介于有机聚合物和无机聚合物之间的一种新型纳米复合材料[16-17],其兼具两者的优点,目前,已有研究将其用于水处理技术中。邱迅[18]研究了一种基于二氧化硅的有机-无机杂化材料,将其用于处理水中低浓度的Cu2+、Cr6+等重金属离子,结果表明,该种杂化材料对Cu2+具有一定的吸附选择性,且在中性条件下吸附效果较好,可将50 mg·L−1以下的K2Cr2O7溶液中的Cr(Ⅵ)几乎完全还原并吸附。该矿区地下水污染成分复杂,单一杂化材料无法使出水Cr(Ⅵ)、
SO2−4 浓度满足要求,单一SRB无法使F−有效去除,目前,很少有研究可同时去除该地区多种污染成分的材料。所以,为克服单一处理方法的局限性,考虑将杂化材料与SRB结合,实现对污染物的有效去除。参考周彩华等[19]利用溶胶-凝胶工艺制备氧化锆溶胶、王国祥[20]利用二氧化钛与丙烯酰胺杂化制备杂化材料的实验方法,本研究选择ZrOCl2与丙烯酰胺单体杂化聚合,得到纳米ZrO2-聚丙烯酰胺杂化材料,利用该杂化材料中聚丙烯酰胺这一中间物质对SRB进行固定化处理,形成纳米ZrO2-SRB颗粒。该颗粒对水中污染物具有还原和吸附双重作用,可以同时去除铬和氟。1. 材料与方法
1.2 硫酸盐还原菌的富集、分离与鉴定
实验所用菌株取自阜新市皮革园区生化池。以乙醇为碳源、按5%接种量接入菌株进行富集培养,直至其适应新碳源环境,并能够大量繁殖;采用叠皿夹层培养法对菌株进行纯化分离,直至得到形态单一菌落,将其继续培养即得到纯化的菌株;对菌株分别进行革兰氏染色、芽孢染色、在1 600倍油镜下镜检观察;将菌株置于2份等量的浅层液体培养基中培养:1份进行摇床振荡好氧培养,1份在液体培养基液面滴加石蜡油置于厌氧培养箱中进行厌氧培养。3 d后分别进行基因测序,并利用透射电镜在放大30 000倍条件下进行镜检观察。
1.2 纳米ZrO2-聚丙烯酰胺杂化材料制备
室温下,称取2 g氧氯化锆,溶于200 mL质量分数为95%的乙醇溶液中,ZrOCl2在乙醇溶液中进行水解和缩聚反应,反应如式(1)和式(2)所示。
Zr−Cl+H2O→Zr−OH+HCl (1) Zr−OH+HO−Zr−O→Zr+H2O (2) 在得到无色透明的纳米二氧化锆明胶后,向200 mL溶胶中加入0.6 g丙烯酰胺单体、0.05 g亚硫酸氢钠和过硫酸钾作为引发剂,将混合溶液充分搅拌均匀,在25 ℃下,进行聚合反应30 min,得到纳米ZrO2-聚丙烯酰胺无机-有机杂化材料。
1.3 硫酸盐还原菌的固定化
称取质量分数为2.5%的海藻酸钠于300 mL蒸馏水中,充分溶胀后,加入200 mL纳米ZrO2-聚丙烯酰胺杂化材料混匀溶解,密封并于室温下存放8~12 h,再向混合溶液中加入质量分数为2.5%的制孔剂聚乙二醇以及100 mL经驯化培养后处于对数期生长的菌液(平板计数法得到菌液对数期的菌密度为3×108个·mL−1),充分混合、搅拌均匀后,利用注射器滴入到pH=6的2%CaCl2饱和硼酸溶液中,期间利用搅拌器以100 r·min−1的搅拌速率进行交联。4 h后取出颗粒,用0.9%生理盐水进行冲洗,再吸干表面水分,重复3遍。在小球使用前,再放入富集培养基中激活12 h。
1)机械强度测试。将固定化细菌颗粒放于100 mL的玻璃注射器中,向玻璃注射器施加一定的压力,观察颗粒的破损情况;同时,用手捏固定好的细菌颗粒,根据整个过程细菌颗粒的变化情况来描述其机械强度,从颗粒的硬度以及弹性对其进行强度分级:当颗粒较软时,认为其强度等级较差;当颗粒具有一定的硬度、弹性较差时,认为其强度等级中等;当颗粒具有一定的硬度且弹性好时,认为其强度等级良好;当颗粒硬度大且易碎时,认为其强度等级为优。
2)传质性能测试。将固定化的细菌颗粒加入到一定量的滴有墨水的蒸馏水中,2 h后取出,观察颗粒颜色进入颗粒的深度,与未加入墨水的固定化颗粒进行对比,确定其传质性能,传质性能分级如下:当颗粒仅有表面变黑且颜色较浅时,认为其传质能力较差;当距离颗粒中心约1/2处变黑且颜色较深时,认为其传质能力中等;当颗粒中心变黑、颜色较浅时,认为其传质能力良好;当颗粒中心变黑、颜色较深时,认为其传质能力为优。
3)成球性能测试。根据固定化过程肉眼判断成球状况的规则性,根据颗粒成球的黏连性判断颗粒的成球性能。成球性能分级如下:当难于成球、黏连严重时,认为其成球性能较差;当成球的形状不规则、部分黏连时,认为其成球性能中等;当成球形状规则、部分黏连时,认为其成球性能良好;当成球形状规则、无黏连时,认为其成球性能为优。
4)细菌活性测试。取一定量的细菌颗粒,置于上述配置的细菌富集培养基中,并向培养基中加入浓度为500 mg·L−1的
SO2−4 ,隔一段时间后,观察培养基的颜色变化情况,测定SO2−4 的浓度变化,根据是否产生臭鸡蛋味的气体情况来判断固定化细菌的活性。细菌活性分级如下:当溶液颜色无明显变化、SO2−4 去除率<20%、产生极少臭鸡蛋气味气体时,认为其活性较差;当溶液颜色较浅、SO2−4 去除率为40%~60%、产生少量臭鸡蛋气味气体时,认为其活性中等;当溶液变为较黑色、SO2−4 去除率60%~80%、产生较多臭鸡蛋气味气体时,认为其活性良好;当溶液变为深黑色、SO2−4 去除率80%~95%、产生大量的臭鸡蛋气味气体时,认为其活性为优。1.4 动态实验
设计6组直径为50 mm、高为50 cm、总容积为0.98 L的动态柱,底部0~3 cm填有进水炉渣含水层,含水层以上30 cm填充反应层,反应层以上设有3 cm炉渣过滤层,如图1所示。1#柱反应层采用纳米ZrO2-SRB颗粒,颗粒中包含200 mL杂化材料和100 mL菌液,进水水力负荷为2.935 m3·(m2·d)−1,进水成分近似模拟该地区地下水的成分:5 mg·L−1 F−、10 mg·L−1 Cr(Ⅵ)、10 mg·L−1 Cr(Ⅲ)、500 mg·L−1
SO2−4 、pH=4.6;2#柱反应层采用与1#柱相同密度的SRB,进行挂膜处理,且在2#柱中加入与1#柱相同量的杂化材料;3#、4#柱进水水力负荷分别为1.468、4.403 m3·(m2·d)−1,5#柱进水成分中将Cr(Ⅵ)提高为50 mg·L−1,6#柱进水成分中将F−提高为10 mg·L−1;各柱中保持纳米ZrO2-SRB颗粒数量以及其他进水条件均与1#柱相同。连续测定出水各个污染物的浓度及pH的提升效果。1.5 再生实验
利用0.1 mol·L−1 HCl、0.2 mol·L−1乙醇和质量分数为2.5%硫脲作为洗脱液,将吸附污染离子后的纳米ZrO2-SRB颗粒加入50 mL洗脱液,并在35 ℃下180 r·min−1下振荡处理60 min,再放入富集培养基中激活12 h。脱附完成后,再次进行吸附,如此吸附-脱附重复3次,并计算每次再生后颗粒对Cr(Ⅵ)、Cr(Ⅲ)、
SO2−4 、F−的去除率。1.6 测定方法
pH采用玻璃电极法测定;Cr(Ⅵ)采用二苯碳酰二肼分光光度法测定;Cr(Ⅲ)采用高锰酸钾氧化-二苯碳酰二肼分光光度法测定;
SO2−4 采用铬酸钡分光光度法测定;F−采用离子选择电极法测定。2. 结果与分析
2.1 硫酸盐还原菌的鉴定结果与分析
1 600倍油镜下镜检SRB的革兰氏染色、经番红复染的芽孢染色、SRB透射电镜放大30 000倍的检测结果如图2所示。由图2(a)可看出,经革兰氏染色后,SRB被染为红色,初步判断该菌株呈阴性;由图2(b)可看出,经番红复染后被染为红色,说明该菌株无芽孢;由图2(c)可明显看出,该菌株呈杆状,且具有鞭毛。
好氧和厌氧条件下培养的菌株经DNA测序后,测序结果相同,说明该菌株生化类型为兼性厌氧型。基因测序以及BLAST基因库比对、序列同源性分析如表1所示,可看出,该兼性厌氧菌与Citrobacter amalonaticus TB10的相似性最高,相似度达99.93%,说明该菌株与Citrobacter amalonaticus TB10属于同一性质的菌株,均为柠檬酸性杆菌。并利用MEGA 6.0软件得到所测菌株序列与其他物质的亲缘关系;得到的进化树结果如图3所示。
表 1 序列同源性分析Table 1. Sequence homology analysis菌属 菌株 相似度/% Citrobacter amalonaticus TB10 99.93 Citrobacter amalonaticus HAMBI 1296 99.86 Citrobacter amalonaticus LMG 7873 99.78 Uncultured Citrobacter sp. clone F2AUG.11 99.71 Citrobacter farmeri CIP 104553 99.64 Citrobacter farmeri 17.7 KSS 99.57 Uncultured bacterium clone KSR-CFL3 99.49 Citrobacter amalonaticus OFF7 99.42 Citrobacter sp CF3-C 99.35 Citrobacter sp. enrichment culture clone TB39-15 99.28 2.2 纳米ZrO2-聚丙烯酰胺杂化材料的结构表征分析
将制得的纳米ZrO2-聚丙烯酰胺杂化材料在60 ℃条件下烘干,采用SEM在放大倍数为5 000倍下观察其表观结构,并进行EDS能谱和FT-IR红外光谱分析,结果如图4所示。可以看出,纳米ZrO2-聚丙烯酰胺杂化材料表面孔隙明显,质地均匀,分散性较好;主要含N—H、C—H、C=O、C—N、Zr—O—Zr特征峰,说明杂化材料中既有有机物吸收峰又有无机物吸收峰,由此可见,ZrO2与聚丙烯酰胺间是通过共价键连接。
2.3 纳米ZrO2-SRB颗粒的性能测试
固定化细菌颗粒如图5所示。通过对其做系列性能分析后,发现其在成球过程中形状规则且无黏连,说明其成球性好;在玻璃注射器中施加一定的压力后不易破损,压力增大,破损程度增大,说明其具有一定的硬度、弹性较好;将其加入到滴有墨水的蒸馏水中,2 h取出后发现其中心颜色变黑,且颜色较深,说明其传质性能良好;将其放于培养基中一段时间后,发现培养基颜色变深,且有黑色沉淀生成,会产生一种臭鸡蛋气味的气体产生,此时测定硫酸根的去除率为69.9%,说明其活性良好。
2.4 动态实验结果分析
6个动态柱的出水情况如图6~图11所示。对比1#、2#动态柱出水情况,可以看出,在SRB和杂化材料投加量相同条件下,纳米ZrO2-SRB颗粒反应层对Cr(Ⅵ)、Cr(Ⅲ)、
SO2−4 、F−的去除效果要好于挂膜的SRB,对溶液中Cr(Ⅵ)、Cr(Ⅲ)、SO2−4 的有效去除时间要长于挂膜的SRB反应层,这说明纳米ZrO2-SRB颗粒可以利用杂化材料中的乙醇作碳源。纳米ZrO2-SRB颗粒对溶液中Cr(Ⅵ)、Cr(Ⅲ)、SO2−4 的作用包括SRB和纳米ZrO2的双重作用,而F−的去除主要依靠纳米ZrO2的吸附作用。Cr(Ⅵ)、Cr(Ⅲ)、SO2−4 、F−的最大去除率分别为99.7%、98.8%、70.4%、92.4%;单独的SRB对Cr(Ⅵ)、Cr(Ⅲ)、SO2−4 的最大去除率分别为99.3%、72.4%、71.2%,对F−没有去除效果。且可以看出,2种反应层对pH的提升效果影响较小,这说明溶液中的pH主要靠SRB的作用,纳米ZrO2对溶液pH没有提升作用。对比1#、3#、4#动态柱的出水情况,可以看出,不同进水水力负荷均不会影响到纳米ZrO2-SRB颗粒对Cr(Ⅵ)、Cr(Ⅲ)、
SO2−4 、F−的最大去除率,对Cr(Ⅵ)、Cr(Ⅲ)、SO2−4 、F−的最大去除率分别为99.7%、98.7%、71.2%、93.7%,但随着进水负荷的增大,维持污染物最大去除率的时间较短,pH最大提升水平维持的时间也有所缩短。在进水水力负荷为2.935 m3·(m2·d)−1、反应进行1~14 d时,F−的去除率可以维持在最大水平,7~23 d期间对Cr(Ⅵ)和SO2−4 的去除率可以维持在最大水平;而当水力负荷为4.403 m3·(m2·d)−1时,对F−的去除率仅在4 d前可维持最大,对Cr(Ⅵ)和SO2−4 的去除率仅在4.5~8.5 d时保持最大,可看出,能够保证各个污染物有效去除的时间明显缩短了。这是因为在反应层高度相同时,进水流速越大,对反应层的传质推动力越大,导致污染物与反应层的接触时间缩短,污染物未来得及和反应层充分接触便流出动态柱,但进水流速也不宜太小,太小的进水流速会延长接触时间,在相同的处理时间内处理的水量小,所以最佳进水水力负荷选择2.935 m3·(m2·d)−1较为适宜。对比1#、5#、6# 3个动态柱内的出水情况,可以看出,当Cr(Ⅵ)的浓度增加到50 mg·L−1时,纳米ZrO2-SRB颗粒对Cr(Ⅵ)的最大去除率仍然可维持在99.7%,但在初始1~3 d时,由于SRB的活性较低,5#动态柱出水中Cr(Ⅵ)的去除率仅为62.3%,相比于1#动态柱去除率91.8%,明显有所下降。这说明纳米ZrO2对高浓度Cr(Ⅵ)的选择吸附性较低,但是靠SRB对Cr(Ⅵ)的还原作用仍然可使出水浓度维持在较佳水平,且当Cr(Ⅵ)浓度增大后,不会影响到纳米ZrO2对F−和Cr(Ⅲ)的吸附效果,但对
SO2−4 的去除效果会有一定影响。由此可见,纳米ZrO2对F−和Cr(Ⅲ)的吸附选择性优于Cr(Ⅵ)优于SO2−4 ;当F−浓度增加到10 mg·L−1时,对比1#和6#动态柱内的出水情况,可以看出,6#动态柱中在反应1~3 d时,对F−、Cr(Ⅵ)、SO2−4 的去除率较1#动态柱中的去除率有所变化,对F−的去除率由93.7%上升为96.7%,对Cr(Ⅵ)的去除率由原来的91.8%下降为87.8%,对SO2−4 的去除率由原来的30.2%降为17.5%,对Cr(Ⅲ)的去除效果基本上没有变化,说明纳米ZrO2对F−的吸附性能优于Cr(Ⅲ)、Cr(Ⅵ)和SO2−4 。2.5 吸附再生实验结果分析
纳米ZrO2-SRB颗粒经过0、1、2、3次脱附再生后,对Cr(Ⅵ)、Cr(Ⅲ)、
SO2−4 、F−的去除结果如图12所示。由图12可看出,经过3次循环再生后,较最初对Cr(Ⅵ)、Cr(Ⅲ)、SO2−4 、F−的去除率仅分别降低了1.8%、4.0%、1.5%、4.2%。由此可见,SRB在经过加入碳源乙醇和培养基活化后可以恢复其活性,颗粒可以达到较好的再生效果。这说明0.1 mol·L−1 HCl、0.2 mol·L−1乙醇、质量分数为2.5%硫脲和培养基的活化作用对于纳米ZrO2-SRB颗粒是一种良好的再生剂。2.6 纳米ZrO2-SRB颗粒处理铬和氟污染地下水的机理分析
1)微观结构表征。将包埋后得到的纳米ZrO2-SRB颗粒和处理不含Cr(Ⅲ)的污染地下水后得到的颗粒分别在60 ℃条件下烘干,采用SEM在放大倍数为2 000倍下观察材料的表观结构和XRD分析,结果如图13所示。可以看出,处理污染物前,细菌颗粒呈现明显的微球状,孔道通畅,表面较为光滑,主要含有的成分是ZrO2和一种有机物CH4N2O·C2H2O4。吸附处理污染水后的细菌颗粒形状变得不为明显,且表面变得粗糙,出现大量的凸形褶皱;处理污染水后的颗粒成分主要有C、O、Zr、S、H、Cr、F等元素;处理不含Cr(Ⅲ)的污水后,出现了ZrCr2H10、C6Cr2O12、ZrS0.67、ZrO0.67F2、Cr(OH)3新物质,Cr最终以Cr(Ⅵ)和Cr(Ⅲ)形式存在,说明SRB可将溶液中的
SO2−4 还原为S2-、将Cr(Ⅵ)还原为Cr(Ⅲ),最终以ZrCr2H10、Cr(OH)3、ZrS0.67的形式被去除,且ZrS0.67是硫化物的最终去向,残留在颗粒中;最终产物中含有Cr(Ⅵ),说明ZrO2-SRB处理污染地下水不但具有还原过程还存在纳米ZrO2的吸附过程,可吸附水中的Cr(Ⅵ)和F−,最终分别以C6Cr2O12和ZrO0.67F2形式被去除。2)等温吸附实验。取100 mL含10 mg·L−1 Cr(Ⅵ)、10 mg·L−1 Cr(Ⅲ)、5 mg·L−1 F−、500 mg·L−1
SO2−4 的溶液9份,每份分别加入质量为0.83、1.66、2.49、3.32、4.15、4.98、5.81、6.64、7.47 g纳米ZrO2-聚丙烯酰胺杂化材料,调节原始溶液至pH=7,置于温度为25 ℃条件下,振荡反应20 min后取出,经过滤后分别测定溶液中Cr(Ⅵ)、Cr(Ⅲ)、F−和SO2−4 浓度。Langmuir和Freundlich模型的方程式分别如式(3)和式(4)所示。
CeQe=1bQm+CeQm (3) lnQe=lnKf+1nlnCe (4) 式中:
Ce 为平衡浓度,mg·L−1;b 为Langmuir 吸附常数,L·mg−1;Qm 为达到饱和时的吸附量,mg·g−1;Qe 为达到动态平衡时的吸附量,mg·g−1。Kf 为Freundlich 吸附常数;n 为经验常数。F−、Cr(Ⅵ)、Cr(Ⅲ)、
SO2−4 4种离子的Langmuir模型和Freundlich模型拟合结果如表2所示。由表2可知,Freundlich模型(R2=0.997 3、0.991 6、0.998 1、0.991 1)相比于Langmuir模型(R2=0.883 9、0.790 0、0.723 2、0.639 6)可以更好地拟合杂化材料对Cr(Ⅵ)、Cr(Ⅲ)、F−、SO2−4 的吸附过程,这说明吸附不仅仅是均匀的单层吸附,更主要的是多层吸附过程。表 2 吸附等温线拟合方程及相关系数Table 2. Adsorption isotherm fitting equation and correlation coefficients离子类型 Langmuir Freundlich 拟合方程 R2 拟合方程 R2 F- 
0.883 9 
0.997 3 Cr(Ⅵ) 
0.790 0 
0.991 6 Cr(Ⅲ) 
0.723 2 
0.998 1 

0.639 6 
0.991 1 3. 结论
1)室内动态柱实验结果表明:纳米ZrO2-SRB颗粒为反应层、进水水力负荷2.935 m3·(m2·d)−1时对污染物的去除效果更好;且ZrO2-SRB颗粒对F−的吸附选择性优于Cr(Ⅲ)、Cr(Ⅵ)和
SO2−4 。2)结构表征结果表明:纳米ZrO2-SRB颗粒处理污染物后出现大量凸形褶皱,且颗粒组成中出现S、Cr、F元素。
3)纳米ZrO2-SRB颗粒处理污染物的机理为:SRB对Cr(Ⅵ)、
SO2−4 存在还原作用,杂化材料对Cr(Ⅵ)、Cr(Ⅲ)、F−存在吸附作用;且吸附等温线符合Freundlich模型,这说明吸附过程是多层吸附。4) 0.1 mol·L−1 HCl、0.2 mol·L−1乙醇、质量分数为2.5%硫脲和培养基的活化共同作用对于纳米ZrO2-SRB颗粒的再生具有良好的效果。
-
表 1 模型选定的描述符
Table 1. Selected descriptors for models
变量Variables 描述Description GATS4c Geary autocorrelation - lag 4 / weighted by charges GATS4s Geary autocorrelation - lag 4 / weighted by I-state ETA_Eta Composite index Eta AATSC6i Average centered Broto-Moreau autocorrelation - lag 6 / weighted by first ionization potential MATS2i Moran autocorrelation - lag 2 / weighted by first ionization potential 表 2 模型预测结果
Table 2. Model prediction results
数据集Dataset 化合物数Chemical number(n) 真阳性True positive(TP) 真阴性True negative(TN) 假阴性False negative(FN) 假阳性False positive(FP) 模型 RF 训练集 104 73 30 0 1 测试集 26 17 6 1 2 模型 SVM 训练集 104 72 29 1 2 测试集 26 16 7 2 1 模型ANN 训练集 104 72 27 1 4 测试集 26 15 7 3 1 模型 KNN 训练集 104 71 28 2 3 测试集 26 15 7 3 1 模型 NB 训练集 104 69 16 4 15 测试集 26 16 6 2 2 模型 LG 训练集 104 70 13 3 18 测试集 26 16 7 2 1 表 3 模型化合物预测情况
Table 3. Prediction of model compounds
序号No. 化合物Compound 参考文献Reference GPER配体分子(是/否) GPER ligand molecule(Y/N) 观测值Observed SVM RF ANN KNN 1 SK0* [25] Y Y Y Y Y 2 SK0P [25] Y Y Y Y Y 3 G-1 [6, 21] Y Y Y Y Y 4 G-15 [6,21] Y Y Y Y Y 5 G-36 [6,21] Y Y Y Y Y 6 Oleuropein [24] Y Y Y Y Y 7 Hydroxytyrosol [24] Y Y Y Y Y 8 MIBE [24] Y Y Y Y Y 9 4-hydroxytamoxifen [24] Y Y Y Y Y 10 GPER-L1 [24] Y Y Y Y Y 11 GPER-L2 [24] Y Y Y Y Y 12 17β-estradiol [22] Y Y Y Y Y 13 E3 [24] N N N Y N 14 Tamoxifen [22] Y Y Y Y Y 15 Fulvestrant [22] Y Y Y Y Y 16 Epi* [27] Y Y Y Y Y 17 Epi-prop [27] Y Y Y Y Y 18 Epi-4-prop [27] Y Y Y Y Y 19 Epi-5-prop [27] Y Y Y Y Y 20 Epi-Ms [27] N N N Y N 21 C4PY [26] Y Y Y Y Y 22 7β-OH-EpiA* [24] Y Y Y Y Y 23 G-DOTA [37] Y Y Y Y Y 24 G-Bz-DOTA [37] N N N N N 25 G-Bz-DTPA [37] N N N N N 26 Atrazine [24] Y Y Y Y Y 27 PBX1 [34] Y Y Y Y Y 28 PBX2 [34] Y Y Y Y Y 29 ZINC65156419(1) [29] Y Y Y Y N 30 ZINC65156419(2) [29] N N N N N 31 ZINC65156419(3) [29] N N N N N 32 ZINC65156419(4) [29] N N N N N 33 ZINC65156419(5) [29] Y Y Y Y Y 34 ZINC65156419(6) [29] N N N N N 35 ZINC65156419(7) [29] N N N N N 36 ZINC65156419(8) [29] N N N N N 37 ZINC65156419(9)* [29] Y Y Y N Y 38 E2-NH3+ [13] Y Y Y Y Y 39 E2-COO- [13] Y Y Y Y Y 40 E2-NMe3+ [13] Y Y Y Y Y 41 E2-NB [13] Y Y Y Y Y 42 o,p'-DDE [6] Y Y Y Y Y 43 E1* [24] N N Y N N 44 α-E2 [24] N Y Y Y Y 45 Genistein [6] Y Y Y Y Y 46 p,p'-DDT [6] Y Y Y Y Y 47 BPA* [6] Y Y Y Y Y 48 quercetin [24] Y Y Y Y Y 49 Resveratrol* [24] Y Y Y Y Y 50 Raloxifene [24] Y Y Y Y Y 51 zearalonone [6] Y Y Y Y Y 52 Nonylphenol [6] Y Y Y Y Y 53 kepone [6] Y Y Y Y Y 54 STX [24] Y Y Y Y Y 55 PPT* [24] Y Y Y Y Y 56 2,2',5'-PCB-4-OH [6] Y Y Y Y Y 57 equol [24] Y Y Y Y Y 58 2-methoxye stradiol [24] Y Y Y Y Y 59 niacin [24] Y Y Y Y Y 60 daidzein [24] Y Y Y Y Y 61 BDE-003 [28] N N N N N 62 BDE-007* [28] N N N N N 63 BDE-028 [28] N N N N N 64 BDE-047 [28] N N N N N 65 BDE-049* [28] N N N N N 66 BDE-085* [28] N N N N N 67 BDE-099* [28] N N N N N 68 BDE-100 [28] N N N N N 69 BDE-154 [28] N N N N N 70 BDE-180 [28] N N N N N 71 BDE-187 [28] N N N N N 72 BDE-201 [28] N N N N N 73 2'-OH-BDE-003* [28] Y N N N N 74 3'-OH-BDE-007 [28] Y Y Y Y Y 75 3'-OH-BDE-028 [28] Y Y Y Y Y 76 3'-OH-BDE-047* [28] Y Y Y Y Y 77 3'-OH-BDE-154 [28] Y Y Y Y Y 78 4'-OH-BDE-049 [28] Y Y Y Y Y 79 5'-OH-BDE-099* [28] Y Y Y Y Y 80 2'-OH-BDE-007 [28] N N N N N 81 2'-OH-BDE-028* [28] N N N N N 82 3-OH-BDE-100 [28] Y N Y N Y 83 4-OH-BDE-187 [28] Y Y Y Y Y 84 4'-OH-BDE-201 [28] Y Y Y Y Y 85 5-OH-BDE-047 [28] N N N N N 86 5'-OH-BDE-100 [28] N N N N N 87 6-OH-BDE-047 [28] N N N N N 88 6-OH-BDE-085* [28] N N N N N 89 6'-OH-BDE-099 [28] N N N N N 90 6-OH-BDE-180 [28] Y Y Y Y N 91 BPAF [16] Y Y Y Y Y 92 BPB* [16] Y Y Y Y Y 93 BPF* [16] N Y Y Y Y 94 BPS* [16] Y Y Y N N 95 TBBPA [16] N N N N Y 96 TCBPA [16] Y Y Y Y Y 97 Diethylstilbestro [24] N N N N N 98 2-Hydroxy stradiol* [24] Y N Y Y N 99 Aldosterone [24] Y Y Y Y Y 100 Tectoridin [24] Y Y Y Y Y 101 Apigenin* [24] Y Y Y Y Y 102 Methoxychlor [24] Y Y Y Y Y 103 p,p'-DDE* [24] Y Y Y Y Y 104 o,p'-DDT* [24] Y Y Y Y Y 105 DPN [24] Y Y Y Y Y 106 Ethynylestradiol [24] Y Y Y Y Y 107 3MC [32] Y Y Y Y Y 108 AB-1 [31] N N N N N 109 CIMBA-5* [36] Y Y Y Y Y 110 CIMBA-6 [36] Y Y Y Y Y 111 CIMBA-7 [36] Y Y Y Y Y 112 CIMBA-8 [36] Y Y Y Y Y 113 CIMBA-9 [36] N Y N Y N 114 CIMBA-10 [36] Y Y Y Y Y 115 CIMBA-11 [36] Y Y Y Y Y 116 CIMBA-12 [36] Y Y Y Y Y 117 CIMBA-13 [36] Y Y Y Y Y 118 CIMBA-14 [36] N N N N N 119 CIMBA-15 [36] Y Y Y Y Y 120 CIMBA-16 [36] N N N N Y 121 CIMBA-17 [36] Y Y Y Y Y 122 CIMBA-18 [36] Y Y Y Y Y 123 CIMBA-19 [36] N N N N N 124 CIMBA-20 [36] Y Y Y Y Y 125 CIMBA-21 [36] Y Y Y Y Y 126 CIMBA-22 [36] Y Y Y Y Y 127 CIMBA-23 [36] Y Y Y Y Y 128 CIMBA-24 [36] Y Y Y Y Y 129 CIMBA-25 [36] Y Y Y Y Y 130 Carbhydraz [35] Y Y Y Y Y 注:*测试集化合物. Note:*Testing set compounds. 表 4 模型评价
Table 4. Model performance
数据集Dataset 化合物数Chemical number(n) 敏感性Sensitivity(Sn) 特异性Specificity(Sp) 精确度Accuracy(Q) 马修斯相关系数Matthews correlation coefficient(MCC) 模型 RF 训练集 104 1 0.968 0.99 0.977 测试集 26 0.944 0.75 0.885 0.723 模型 SVM 训练集 104 0.986 0.935 0.971 0.931 测试集 26 0.889 0.875 0.885 0.741 模型ANN 训练集 104 0.986 0.871 0.952 0.884 测试集 26 0.833 0.875 0.846 0.672 模型 KNN 训练集 104 0.972 0.903 0.952 0.884 测试集 26 0.833 0.875 0.846 0.672 模型 NB 训练集 104 0.945 0.516 0.817 0.535 测试集 26 0.889 0.75 0.846 0.639 模型 LG 训练集 104 0.959 0.419 0.798 0.480 测试集 26 0.889 0.875 0.885 0.741 -
[1] FENICHEL P, CHEVALIER N. Is testicular germ cell cancer estrogen dependent? The role of endocrine disrupting chemicals [J]. Endocrinology, 2019, 160(12): 2981-2989. doi: 10.1210/en.2019-00486 [2] BALABANIC D, RUPNIK M S, KLEMENCIC A K. Negative impact of endocrine-disrupting compounds on human reproductive health [J]. Reproduction Fertility and Development, 2011, 23(3): 403-416. doi: 10.1071/RD09300 [3] BRAUN J M. Early-life exposure to EDCs: Role in childhood obesity and neurodevelopment [J]. Nature Reviews Endocrinology, 2017, 13(3): 161-173. doi: 10.1038/nrendo.2016.186 [4] FOWLER P A, BELLINGHAM M, SINCLAIR K D, et al. Impact of endocrine-disrupting compounds (EDCs) on female reproductive health [J]. Molecular and Cellular Endocrinology, 2012, 355(2): 231-239. doi: 10.1016/j.mce.2011.10.021 [5] HARRIS H A, KATZENELLENBOGEN J A, KATZENELLENBOGEN B S J E. Characterization of the biological roles of the estrogen receptors, ERα and ERβ, in estrogen target tissues in vivo through the use of an ERα-selective ligand [J]. Endocrinology, 2002(11): 4172-4177. [6] THOMAS P, DONG J. Binding and activation of the seven-transmembrane estrogen receptor GPR30 by environmental estrogens: A potential novel mechanism of endocrine disruption [J]. The Journal of Steroid Biochemistry and Molecular Biology, 2006, 102(1): 175-179. [7] PROSSNITZ E R, BARTON M. The G-protein-coupled estrogen receptor GPER in health and disease [J]. Nature Reviews Endocrinology, 2011, 7(12): 715-726. doi: 10.1038/nrendo.2011.122 [8] PROSSNITZ E R, BARTON M. Signaling, physiological functions and clinical relevance of the G protein-coupled estrogen receptor GPER [J]. Prostaglandins Other Lipid Mediat, 2009, 89(3-4): 89-97. doi: 10.1016/j.prostaglandins.2009.05.001 [9] WANG C, LI J, YE S, et al. Oestrogen inhibits VEGF expression and angiogenesis in triple-negative breast cancer by activating GPER-1 [J]. Journal of Cancer, 2018, 9(20): 3802-3811. doi: 10.7150/jca.29233 [10] NIRO S, PEREIRA E, PELISSIER M A, et al. The DHEA metabolite 7β-hydroxy-epiandrosterone exerts anti-estrogenic effects on breast cancer cell lines [J]. Steroids, 2012, 77(5): 542-551. doi: 10.1016/j.steroids.2012.01.019 [11] PROSSNITZ E, BARTON M. Estrogen biology: New insights into GPER function and clinical opportunities [J]. Molecular and Cellular Endocrinology, 2014, 389(1): 71-83. [12] ALBANITO L, MADEO A, LAPPANO R, et al. G protein-coupled receptor 30 (GPR30) mediates gene expression changes and growth response to 17β-estradiol and selective GPR30 ligand G-1 in ovarian cancer cells [J]. Cancer Research, 2007, 67(4): 1859-1866. doi: 10.1158/0008-5472.CAN-06-2909 [13] REVANKAR C M, MITCHELL H D, FIELD A S, et al. Synthetic estrogen derivatives demonstrate the functionality of intracellular GPR30 [J]. Acs Chemical Biology, 2007, 2(8): 536-544. doi: 10.1021/cb700072n [14] TSUGAWA Y, HIRAMOTO M. IMAI T. Estrogen induces estrogen receptor α expression and hepatocyte proliferation in late pregnancy [J]. Biochemical & Biophysical Research Communications, 2019, 511(3): 592-596. [15] PROSSNITZ E R. GPER modulators: Opportunity nox on the heels of a class akt [J]. The Journal of Steroid Biochemistry and Molecular Biology, 2018, 176: 73-81. doi: 10.1016/j.jsbmb.2017.03.005 [16] CAO L Y, REN X M, LI C H, et al. Bisphenol AF and bisphenol B exert higher estrogenic effects than bisphenol A via G protein-coupled estrogen receptor pathway [J]. Environmental Science & Technology, 2017, 51(19): 11423-11430. [17] RUSSO D P, ZORN K M, CLARK A M, et al. Comparing multiple machine learning algorithms and metrics for estrogen receptor binding prediction [J]. Molecular Pharmaceutics, 2018, 15(10): 4361-4370. doi: 10.1021/acs.molpharmaceut.8b00546 [18] GRISONI F, CONSONNI V. BALLABIO D. Machine learning consensus to predict the binding to the androgen receptor within the compara project [J]. Journal of Chemical Information and Modeling, 2019, 59(5): 1839-1848. doi: 10.1021/acs.jcim.8b00794 [19] SHEFFIELD T, JUDSON R. Ensemble QSAR modeling to predict multispecies fish toxicity lethal concentrations and points of departure [J]. Environmental Science & Technology, 2019, 53(21): 12793-12802. [20] WANG Z, CHEN J, HONG H. Applicability domains enhance application of PPARγ agonist classifiers trained by drug-like compounds to environmental chemicals [J]. Chemical Research in Toxicology, 2020, 33(6): 1382-1388. doi: 10.1021/acs.chemrestox.9b00498 [21] ARNATT C K, ZHANG Y. G protein-coupled estrogen receptor (GPER) agonist dual binding mode analyses toward understanding of its activation mechanism: a comparative homology modeling approach [J]. Molecular Informmatics, 2013, 32(7): 647-658. doi: 10.1002/minf.201200136 [22] WANG D, HU L, ZHANG G, et al. G protein-coupled receptor 30 in tumor development [J]. Endocrine, 2010, 38(1): 29-37. doi: 10.1007/s12020-010-9363-z [23] BARTON M, PROSSNITZ E R. Emerging roles of GPER in diabetes and atherosclerosis [J]. Trends in Endocrinology & Metabolism, 2015, 26(4): 185-192. [24] PROSSNITZ E, ARTERBURN J. International union of basic and clinical pharmacology. XCVII. G protein-coupled estrogen receptor and its pharmacologic modulators [J]. Pharmacological Reviews, 2015, 67(3): 505-540. doi: 10.1124/pr.114.009712 [25] KHAN S U, AHEMAD N, CHUAH L H, et al. Sequential ligand and structure-based virtual screening approach for the identification of potential G protein-coupled estrogen receptor-1 (GPER-1) modulators [J]. Rsc Advances, 2019, 9(5): 2525-2538. doi: 10.1039/C8RA09318K [26] LAPPANO R, ROSANO C, PISANO A, et al. A calixpyrrole derivative acts as an antagonist to GPER, a G-protein coupled receptor: mechanisms and models [J]. Disease models & mechanisms, 2015, 8(10): 1237-1246. [27] SARMIENTO V, SANCHEZ T R, ULLOA A M, et al. Synthesis of novel (-) -epicatechin derivatives as potential endothelial GPER agonists: Evaluation of biological effects [J]. Bioorganic & Medicinal Chemistry Letters, 2018, 28(4): 658-663. [28] CAO L Y, REN X M, YANG Y, et al. Hydroxylated polybrominated biphenyl ethers exert estrogenic effects via non-genomic G protein-coupled estrogen receptor mediated pathways [J]. Environmental Health Perspectives, 2018, 126(5): 057005. doi: 10.1289/EHP2387 [29] O’DEA A, SONDERGARD C, SWEENEY P, et al. A series of indole-thiazole derivatives act as GPER agonists and inhibit breast cancer cell growth [J]. Acs Medicinal Chemistry Letters, 2018, 9(9): 901-906. doi: 10.1021/acsmedchemlett.8b00212 [30] ALBANITO L, LAPPANO R, MADEO A, et al. Effects of atrazine on estrogen receptor α and G protein-coupled receptor 30-mediated signaling and proliferation in cancer cells and cancer-associated fibroblasts [J]. Environmental Health Perspectives, 2015, 123(5): 493-499. doi: 10.1289/ehp.1408586 [31] REVANKAR C M, BOLOGA C G, PEPERMANS R A, et al. A selective ligand for estrogen receptor proteins discriminates rapid and genomic signaling [J]. Cell Chemical Biology, 2019, 26(12): 1692-1702. doi: 10.1016/j.chembiol.2019.10.009 [32] CIRILLO F, LAPPANO R, BRUNO L, et al. AHR and GPER mediate the stimulatory effects induced by 3-methylcholanthrene in breast cancer cells and cancer-associated fibroblasts (CAFs) [J]. Journal of Experimental & Clinical Cancer Research, 2019, 38(1): 335-353. [33] ALDO M U, DAVID M L, ERNESTO B R, et al. The effects of (-)-epicatechin on endothelial cells involve the G protein-coupled estrogen receptor (GPER) [J]. Pharmacological Research, 2015, 100: 309-320. doi: 10.1016/j.phrs.2015.08.014 [34] MAGGIOLINI M, SANTOLLA M, AVINO S, et al. Identification of two benzopyrroloxazines acting as selective GPER antagonists in breast cancer cells and cancer-associated fibroblasts [J]. Future medicinal chemistry, 2015, 7(4): 437-448. doi: 10.4155/fmc.15.3 [35] ROSANO C, PONASSI M, SANTOLLA M F, et al. Macromolecular modelling and docking simulations for the discovery of selective GPER ligands [J]. The AAPS Journal, 2016, 18(1): 41-46. doi: 10.1208/s12248-015-9844-3 [36] DELEON C, WANG H, GUNN J, et al. A novel GPER antagonist protects against the formation of estrogen-induced cholesterol gallstones in female mice [J]. Journal of Lipid Research, 2020, 61(5): 767-777. doi: 10.1194/jlr.RA119000592 [37] NAYAK T K, DENNIS M K, RAMESH C, et al. Influence of charge on cell permeability and tumor imaging of GPR30-targeted 111in-labeled nonsteroidal imaging agents [J]. ACS Chemical Biology, 2010, 5(7): 681-690. doi: 10.1021/cb1000636 [38] LUO L J, LIU F, LIN Z K, et al. Genistein regulates the IL-1 beta induced activation of MAPKs in human periodontal ligament cells through G protein-coupled receptor 30 [J]. Archives of Biochemistry and Biophysics, 2012, 522(1): 9-16. doi: 10.1016/j.abb.2012.04.007 -




 下载:
下载: