-
莫诺拉韦(Molnupiravir)和巴瑞替尼(Baricitinib)是常见的抗病毒药物[1]. 其中,莫诺拉韦具有广谱的抗RNA病毒活性,对新型冠状病毒也有显著抑制作用;巴瑞替尼是口服选择性JAK1/2 抑制剂,具有抗癌、免疫调节和抗炎活性. 自新冠肺炎疫情爆发以来,莫诺拉韦和巴瑞替尼迅速被许多国家纳入疾病治疗指南,并大量投入到新冠肺炎患者的治疗当中. 莫诺拉韦和巴瑞替尼等抗病毒药物可通过医疗应用、药物处置和不完全代谢等途径进入天然水体,导致药物在污水中的浓度升高[2]. 在水生环境中的质量浓度范围在ng·L−1—μg·L−1[3]. 这些药物的持续排放不仅会威胁生态环境健康,还会对城市生活饮用水安全构成潜在风险[4]. 因此,迫切需要探索去除水生环境中残留莫诺拉韦、巴瑞替尼的新技术.
传统的污水处理方法(如混凝、沉淀、过滤和消毒)对其去除效果有限[5]. Kuroda[6]的研究表明,传统污水处理厂对羟氯喹(Hydroxychloroquine)、瑞德西韦(Remdesivir)、利巴韦林(Ribavirin)等抗新冠肺炎药物及其代谢物的去除率小于20%. 近年来,高级氧化工艺(AOPs)因其具有将人为和生物难降解有机物转化为矿物质的潜力而越来越普遍地应用于饮用水消毒及污染物质的去除[7]. 紫外辐射具有提高自由基量子产率以及降低其选择性的能力. 因此,将紫外辐射结合氧化剂在AOPs中产生活性氧自由基已成为一种常用的催化活化方法[8]. 紫外/过氧化氢(UV/H2O2)和紫外/过硫酸盐(UV/PDS)可以有效降解药品和个人护理产品(PPCPs),具有高效、低耗的特点[9]. 然而,在极大促进污染物去除的同时,也出现了一些局限性,比如:UV/H2O2体系中H2O2难以消耗完全,因此需要一定的后续工艺来清理残留物,由此会造成成本的增加[10];而UV/PDS体系中,活性物质的产率低限制了其在水处理中的应用[11]. 紫外/氯工艺因其具有明显的节能优势和较高的吸光度、量子产率和氯残留,目前已被广泛应用于饮用水的消毒和循环水的净化[12].
一般来说,常规的氯化物包括次氯酸盐和单氯胺. Wang等[13]将传统的紫外/氯工艺(紫外/次氯酸盐(UV/HClO)、紫外/单氯胺(UV/NH2Cl))在成本效益、吸光度、自由基产率、去除新污染物效率方面与紫外/二氯异氰尿酸盐(UV/Dichloroisocyanurate, NaDCC)体系进行了对比. 结果表明,使用UV/NaDCC在纯水中实现ECs去除的成本比使用UV/HClO低4%—53%,比使用UV/NH2Cl低75%—95%,而使用UV/NaDCC在实际水中实现新污染物(ECs)去除的成本比使用UV/HClO低26%—91%,比使用UV/NH2Cl低89%—96%;与HClO和NH2Cl相比,NaDCC是一种平衡良好的化合物,具有较强的紫外吸收能力和中等的量子产率;UV/NaDCC覆盖了UV/HClO工艺中的所有自由基,且大多数自由基浓度在UV/NaDCC中显著高于其他紫外/氯工艺;在去除新污染物效率方面,UV/NaDCC在多种水基质中的降解性能优于传统的紫外/氯工艺,具有优异的降解效率. 该研究为水中污染物的去除提供了一个新的研究方向,但是在实验中验证该体系对抗病毒药物的降解往往耗时耗力,因此本研究以模型预测的方式探究了莫诺拉韦、巴瑞替尼在紫外/二氯异氰尿酸盐体系下的降解路径及中间产物的毒性.
本研究以UV/NaDCC作为降解体系,采用Gaussian和Multiwfn计算了莫诺拉韦、巴瑞替尼的福井函数及简缩双描述符,预测了其在UV/NaDCC体系下的反应位点及降解路径,采用毒性评估软件ECOSAR、T.E.S.T.对莫诺拉韦、巴瑞替尼及其降解中间体和产物的急性毒性、慢性毒性、生物蓄积性、发育毒性进行定量构效关系(QSAR)预测.
-
本研究通过GaussView5.0构建分子结构,计算任务为频率优化加结构分析(Opt+Freq),方法选择B3LYP,基组设置为6-311G,将计算任务提交Gaussian开始计算. 基于密度泛函理论(DFT)用Multiwfn[14]计算了Molnupiravir、Baricitinib的福井函数[15],根据福井函数的数值可以推测易发生亲电(f −)、亲核(f +)或者自由基攻击(f 0)的位点[16].
-
双描述符(DD)[17]是在概念密度泛函理论框架下定义的一种实空间函数,用于显示活性位点. DD为负值的区域易受亲电攻击,DD为正值的区域易受亲核攻击,在实际分析中,一个位点上的DD越正(负),该位点与亲核试剂(亲电试剂)反应的倾向就越大[18]. 双描述符与福井函数的区别是:双描述符可以同时展现亲电与亲核反应位点而不需要分别考察f +和f −.
-
ECOSAR和Toxicity Estimation Software Tool(T.E.S.T)是由United States Environmental Protection Agency(EPA,美国环境保护署)开发的毒性评估软件. ECOSAR作为一种有效的预测程序已成功地应用于有机污染物的生态毒性评估[19],广泛用于水生生物毒性的估计;T.E.S.T能够使用定量结构活性关系(QSAR)方法估计化学品的毒性,预测结果与实测结果保持大体一致,具有较好的准确性[20]. 本研究利用ECOSAR程序对莫诺拉韦、巴瑞替尼分子及其降解产物进行毒性评估,预测了莫诺拉韦、巴瑞替尼及其降解产物对鱼类、绿藻和水蚤的急性毒性(LC50、EC50)和慢性毒性(Chv),毒性分为剧毒、有毒、有害和无害;本文还利用T.E.S.T软件(层级分析法)对其生物蓄积性、发育毒性进行了预测.
-
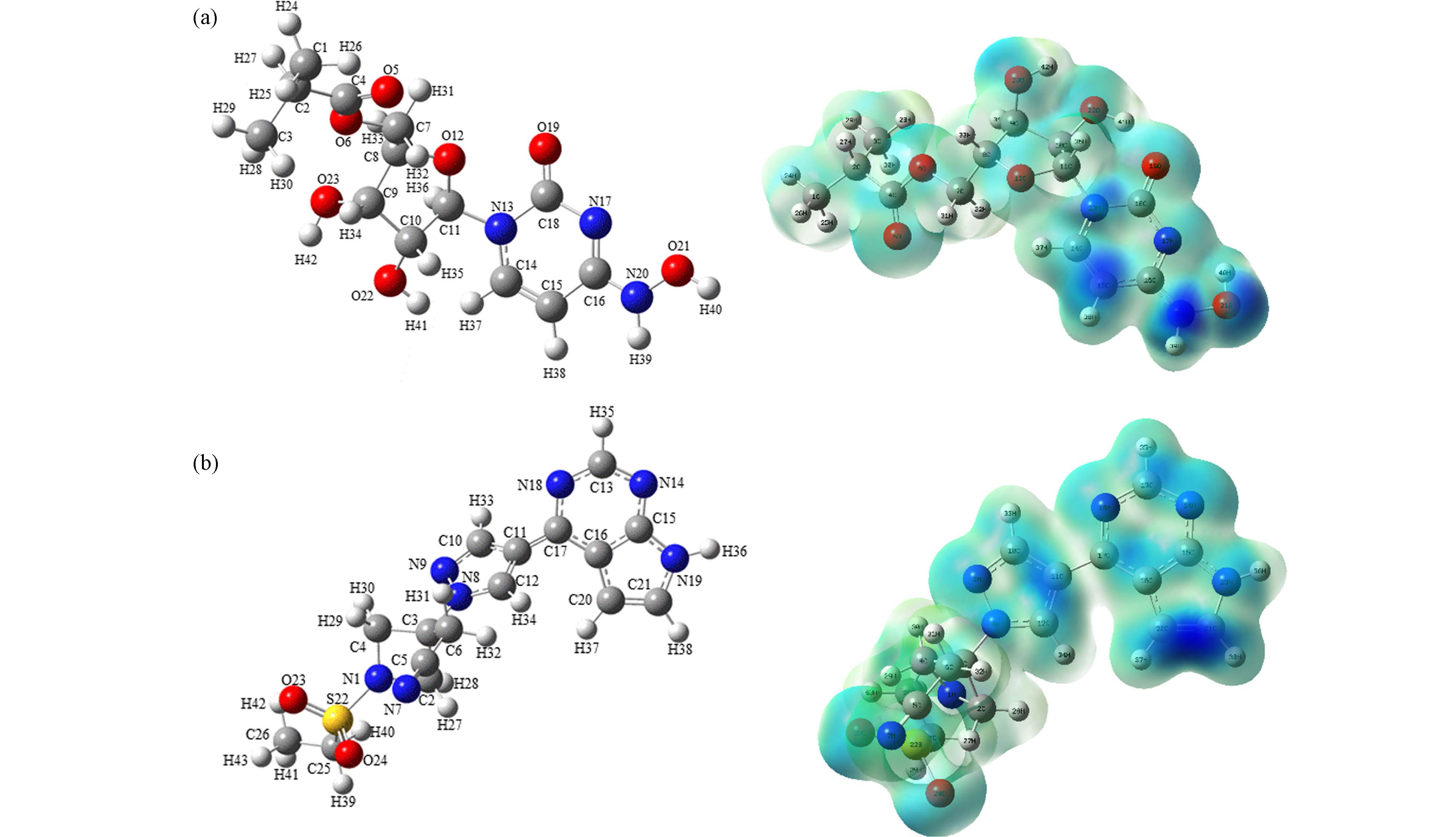
基于福井函数的对比分析可以预测出反应的活性位点,其中,f −(f +)越大的位点,越容易受到亲电(亲核)攻击[21],而较高的f 0值表示容易受到自由基攻击. 在AOPs领域,自由基被认为是最重要的活性物质,因此f 0被广泛使用. 然而值得注意的是,f 0同时考虑了自由基的亲电性和亲核性,当自由基在反应中主要表现出亲电性时,f −更适合用于预测反应活性位点[22]. 在本研究中,由于氯自由基、氧自由基等强亲电性自由基的存在,亲电攻击是主要的攻击方式[23]. 莫诺拉韦的分子结构如图1(a)所示,福井函数如表1所示. O21(0.1123)、N20(0.0909)、C15(0.0770)的福井函数值最大,且被福井函数负等值面紧密包围(图1(a)). 因此,推测其为亲电攻击优先位点;巴瑞替尼的分子结构如图1(b)所示,由表2可知,C21(0.0859)、N14(0.0691)、C20(0.0666)的福井函数值最大,根据福井函数f −在电子密度等值面上的填色图(图1(b))可知,C20、C21被负等值面紧密包围,相比之下N14周围负等值面不够紧密,综合推测,C21、N14、C20为优先攻击位点,在反应体系中受到·OH和含氯自由基(Cl·,ClO·和Cl2·−)等自由基攻击的可能性更大.
-
为了进一步验证上述预测的易受攻击的反应位点,本研究采用Hirshfeld电荷在Multiwfn中接着计算了简缩双描述符(CDD),以定量比较每个位点易受攻击的可能性. CDD为负值的区域易受亲电攻击,而CDD为正值的区域更容易发生亲核反应. 与福井函数类似,本研究也绘制了基于双描述符的等值面图,便于综合分析各个位点受到攻击的可能性. 由表3(a)可以发现,在莫诺拉韦中O21(−0.0529)、O23(−0.0352)、N20(−0.0332)的CDD值最负,因此初步推测这3个位点更易受到亲电攻击,发生亲电反应,根据表3(a)中莫诺拉韦基于双描述符的等值面图可知,N20、O21、C15受到负等值面的紧密包围,易受到亲电攻击,相比之下O23受到亲电攻击的可能性会小一些. 在表3(b)中,巴瑞替尼C20(−0.0439)、C21(−0.0274)、C11(−0.0267)具有最负的CDD值,推测这3个位点更容易受到亲电攻击,结合等值面图(表3(b))来看,C11、C20受到较大负等值面包围,C21也在一定程度上被负等值面包围.
结合福井函数及简缩双描述符对易受到亲电攻击位点的预测发现,两种函数对反应位点的预测吻合较好. 综合分析,在莫诺拉韦中O21、N20发生反应的可能性最大,其中C15也有发生反应的可能;在巴瑞替尼中,最有可能发生反应的位点是C20、C21,其中C11也存在发生反应的可能.
-
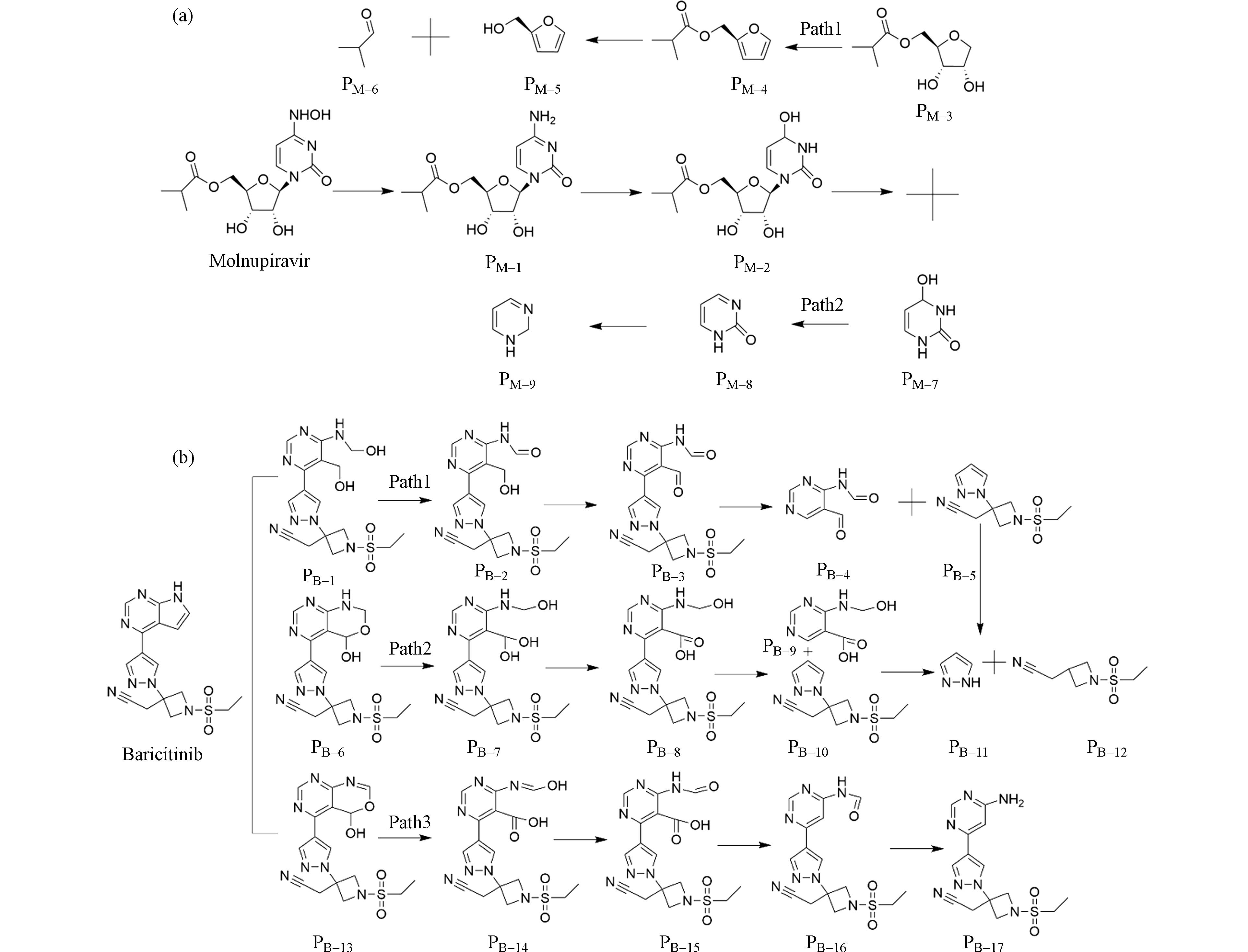
在紫外/二氯异氰尿酸盐体系下,·OH和RCSs对莫诺拉韦的降解起主导作用,根据福井函数及简缩双描述的评价结果可知:在羟胺转变为仲胺的光照条件下,O21位点首先发生脱水反应形成PM-1,PM-1在·OH 攻击下在N20位点发生取代反应转化为PM-2,随着反应的进一步进行,PM-2在N13位点发生裂解反应生成PM-3和PM-7,随后PM-3经过质子化反应脱水转化为PM-4,PM-4发生取代反应转化为PM-5、PM-6. 路径2的降解路径符合上述理论分析且与Paymode[24]的部分研究结果类似. 经研究,本文预测的莫诺拉韦降解路径(图2(a))与Jain等[25]在实验室尺度上预测的路径基本一致. 由于目前对莫诺拉韦的研究较少且多聚焦于莫诺拉韦的高效合成[26],在实验室尺度上对莫诺拉韦降解途径的研究还仍然缺乏,本研究的预测是基于机理性的,尚存在较大局限性. 巴瑞替尼的降解路径如图2(b)所示,路径1中,C20、C21首先被氢氧化物攻击导致C20-C21键断裂生成PB-1,PB-1进一步去质子化生成PB-2,PB-2在分子氧存在下进行环氧化反应生成PB-3,结合福井函数及简缩双描述符分析结果,C11也存在发生反应的可能,因此预测PB-3在自由基攻击下会发生断裂,生成PB-4、PB-5. 相似地,路径2、路径3的猜测也是基于前文分析的结果,降解路径预测的总体趋势符合Chaganti[27]的降解实验分析. 综合来看,福井函数以及简缩双描述符能够较好地反映出易发生反应的位点.
-
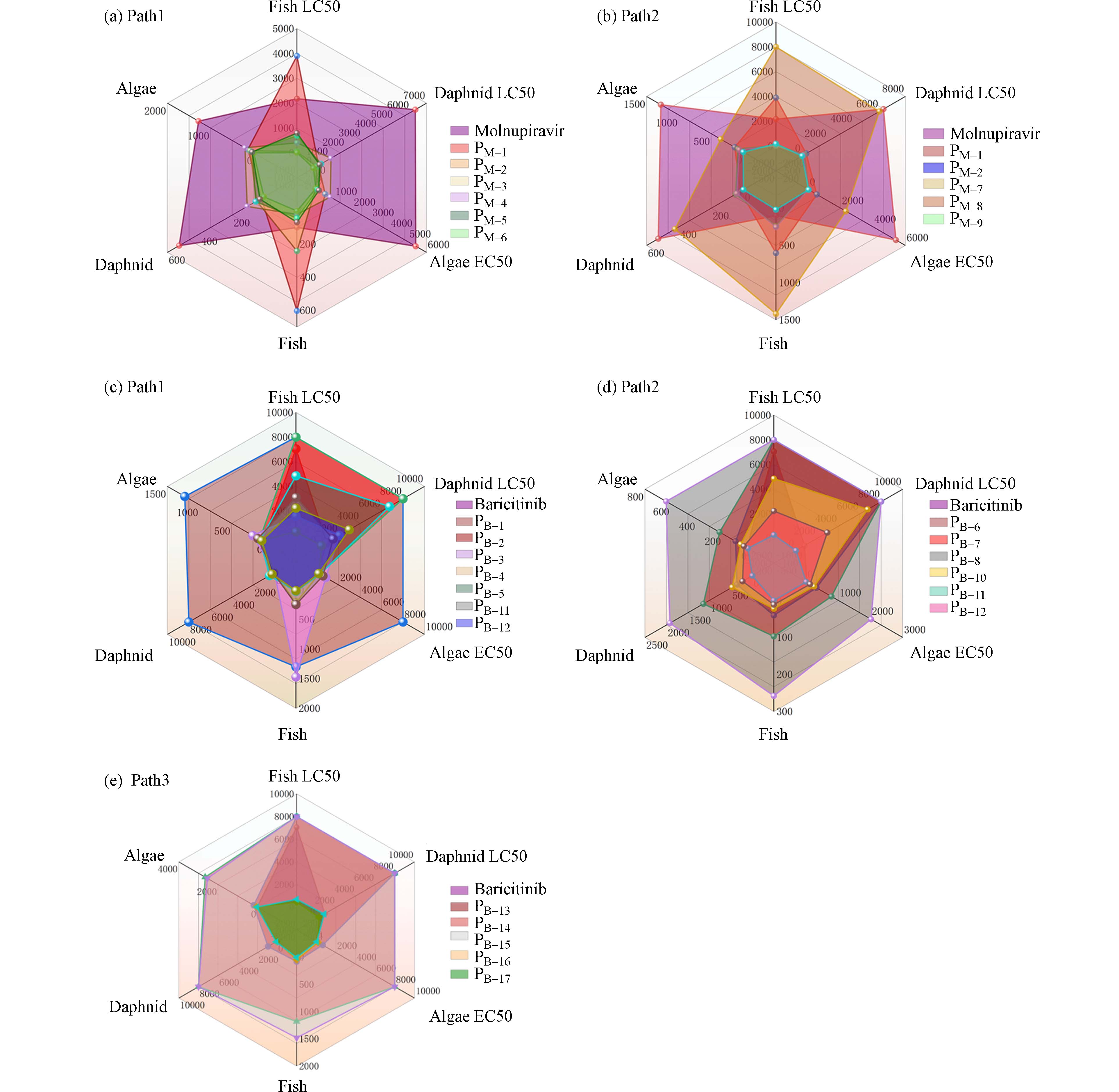
根据不同的降解路径,本文将莫诺拉韦及其产物分为2组,巴瑞替尼及其产物分为3组. 为了雷达图的直观性,本文将LC50大于10000 mg·L−1显著无害的降解产物统一取值为8000 mg·L−1来作图;巴瑞替尼降解路径2中产物PB-9处于显著无害状态,在图中并未显示. 根据欧盟制定的毒性分级标准,利用ECOSAR预测发现,莫诺拉韦对鱼类、水蚤和绿藻的LC50或EC50分别为2.18×103、6.31×103、5.42×103 mg·L−1,均处于无害状态;莫诺拉韦对鱼类、水蚤和绿藻的慢性毒性也处于无害状态. 虽然莫诺拉韦对鱼类、水蚤和绿藻没有毒性,但它可能加速抗病毒药物耐药性的建立,因此其去除是必要的,同时也需要关注降解产物的毒性变化. 如图3(a)路径1所示,PM-2对鱼类的急性毒性为有害状态,对绿藻的急性毒性达到有毒状态;PM-4对鱼类、水蚤、绿藻的急性毒性均达到有害水平,对水蚤、绿藻的慢性毒性达到有害水平,其中对鱼类的慢性毒性达到有毒水平. 路径2中PM-7对鱼类、绿藻的急性毒性分别为有害、有毒水平;PM-9对水蚤和绿藻的急慢性毒性均达到有害水平. 在所有降解路径中绿藻对该产品更为敏感,这与Jiang关于利巴韦林的研究保持一致[28]. 巴瑞替尼对鱼类、水蚤和绿藻的LC50或EC50分别为7.05×103、8.66×102、27.6 mg·L−1,对鱼类、水蚤无害,对绿藻表现出有害;对鱼类和绿藻表现出慢性毒性.
如图3(c)、3(d)、3(e)所示,在路径1中,PB-4对水蚤的慢性毒性为有害;PB-5对水蚤的急慢性毒性处于有害水平,对绿藻的急性毒性达到有毒水平;PB-11对绿藻的急性毒性处于有毒状态,对鱼类的慢性毒性达到有毒水平,对水蚤和绿藻都有害;路径2中,除PB-11外,PB-10对绿藻的急性毒性达到有害状态,对鱼类、水蚤和绿藻的慢性毒性也达到有害水平. 部分降解产物表现出比母体物质更高的毒性,类似现象已在许多研究中发现[29 − 31]. 这可能是由于在本研究中降解产物还未达到毒性降低的转折点[32]. 预测产物的毒性在降解过程中会有所增加,表明UV/NaDCC在莫诺拉韦和巴瑞替尼的降解中存在一定风险. 因此,有毒中间产物的去除可能是UV/NaDCC实际应用于抗病毒药物降解的关键.
由于莫诺拉韦、巴瑞替尼在疫情期间的大量使用,定性评价其毒性往往具有局限性. 因此,可以利用生物蓄积性、发育毒性等参数来描述莫诺拉韦和巴瑞替尼在水中毒性的大致变化. 经过T.E.S.T预测,莫诺拉韦和巴瑞替尼生物蓄积性和发育毒性预测结果如图4所示. 莫诺拉韦的生物蓄积性为0.0357和明显的发育毒性(0.83),经路径1降解后产物PM-5生物蓄积性增高(2.82)、发育毒性降至无毒(0.46),PM-6生物蓄积性未检出、发育毒性略有降低(0.82);降解路径2中,PM-9生物蓄积性未检出、发育毒性从0.83降至0.51(PM-9). 巴瑞替尼具有较强的生物蓄积性(3.53)和明显的发育毒性(0.78),在路径1、路径2中产物PB-11、PB-12生物蓄积性分别降至0.81(PB-11)和3.1(PB-12),经路径3降解后,巴瑞替尼降解产物PB-17生物蓄积性有所增加(3.91),发育毒性降至0.74. 除莫诺拉韦降解产物PM-5、巴瑞替尼降解产物PB-17生物蓄积性有所增加,其余降解产物生物蓄积性和发育毒性均有所降低. 莫诺拉韦、巴瑞替尼较高的生物蓄积性和发育毒性将导致其以较高生物活性的形式持续存在于水生环境中,对水生生态系统和生物健康带来威胁,因此在充分考虑有毒中间产物去除的前提下其降解是必要的.
-
本文通过Gaussian09和Multiwfn程序计算了莫诺拉韦和巴瑞替尼的福井函数、简缩双描述符,结果表明,f −较大的点与CDD越负的位点相吻合. 结合福井函数及简缩双描述符预测发现,莫诺拉韦中O21、N20、C15,巴瑞替尼中C20、C21、C11是UV/NaDCC中的反应活性位点. 进而通过所预测的活性位点推测了莫诺拉韦和巴瑞替尼可能的降解路径. 利用ECOSAR、T.E.S.T程序对莫诺拉韦、巴瑞替尼分子及其降解产物在水生环境中的行为做出预测,部分降解产物表现出比母体物质更高的急、慢性毒性,综合其在环境中的生物蓄积性、发育毒性来看:莫诺拉韦降解产物PM-5、巴瑞替尼降解产物PB-17生物蓄积性有所增加,其余降解产物生物蓄积性和发育毒性均有所降低. 因此环境中莫诺拉韦、巴瑞替尼的降解应重点关注其有毒中间产物的产生以及去除.
紫外/二氯异氰尿酸盐体系对水中抗病毒药物降解路径及降解产物毒性分析
Analysis of degradation pathways and toxicity of degradation products of antiviral drugs in water by UV/Dichloroisocyanurate process
-
摘要: 紫外/二氯异氰尿酸盐体系(UV/NaDCC)是一种新兴的高级氧化工艺,通过产生羟基自由基(·OH)和含氯自由基(Cl·,ClO·和Cl2·−)等活性物质降解水中持久性有机污染物. 莫诺拉韦和巴瑞替尼是用于治疗新冠肺炎等的抗病毒类药物,大量生产和使用导致其通过各环境介质排放进入天然水体,威胁水环境和水质安全. 因此,迫切需要探索高效绿色降解莫诺拉韦和巴瑞替尼的新技术. 本研究采用密度泛函理论,通过福井函数、简缩双描述符预测了UV/NaDCC体系中莫诺拉韦和巴瑞替尼易发生反应的位点及其降解路径;通过ECOSAR和T.E.S.T程序预测降解产物毒性变化. 结果表明,莫诺拉韦中O21、N20及巴瑞替尼中C20、C21更易受到UV/NaDCC产生的含氯自由基和·OH攻击. ECOSAR预测结果显示,部分降解产物表现出比母体物质更高的毒性水平,因此在UV/NaDCC实际应用于抗病毒药物的降解中应关注有毒中间产物的去除. T.E.S.T预测结果表明,多数降解产物生物蓄积性和发育毒性均有所降低. 研究成果对UV/NaDCC高级氧化技术对水中抗病毒药物的无害化去除机制和实际应用提供理论依据.Abstract: UV/Dichloroisocyanurate (UV/NaDCC) process is an emerging advanced oxidation process for degrading persistent organic pollutants in aquatic environments by efficiently generating various reactive species such as hydroxyl radicals (·OH) and reactive chlorine species (including Cl·, ClO·, and Cl2·−). Monoravir and Baricitinib are antiviral drugs used to treat COVID-19 pneumonia, which could be inevitably released into natural water via various environmental media due to their extensive production and usage, and then threaten the aquatic environment and water quality safety. Therefore, it is urgent to develop new technologies for efficiently degrading Monolavir and Baricitinib. In this study, the susceptible reaction sites and degradation pathways of Molnupiravir and Baricitinib were investigated and proposed on the basis of density functional theory, Fukui function, and Condensed dual descriptor, and the toxicity changes of the degradation products were evaluated by ECOSAR and T.E.S.T. programs. The results showed that O21 and N20 in Molnupiravir and C20 and C21 in Baricitinib were more susceptible to attack by RCSs and ·OH produced by UV/NaDCC. ECOSAR predictions showed that some degradation products exhibited higher toxicity than their parent substance, so the removal of toxic intermediates should be concerned in the application of UV/NaDCC for the degradation of antiviral medications. T.E.S.T predictions indicated that the Bioconcentration factor and Developmental Toxicity of most degradation products were reduced. The results provide a theoretical basis for the harmless removal mechanism and practical application of UV/NaDCC advanced oxidation technology to antiviral medications in water.
-
Key words:
- UV/NaDCC /
- degradation pathway /
- product toxicity /
- density functional theory /
- Molnupiravir /
- Baricitinib.
-
莫诺拉韦(Molnupiravir)和巴瑞替尼(Baricitinib)是常见的抗病毒药物[1]. 其中,莫诺拉韦具有广谱的抗RNA病毒活性,对新型冠状病毒也有显著抑制作用;巴瑞替尼是口服选择性JAK1/2 抑制剂,具有抗癌、免疫调节和抗炎活性. 自新冠肺炎疫情爆发以来,莫诺拉韦和巴瑞替尼迅速被许多国家纳入疾病治疗指南,并大量投入到新冠肺炎患者的治疗当中. 莫诺拉韦和巴瑞替尼等抗病毒药物可通过医疗应用、药物处置和不完全代谢等途径进入天然水体,导致药物在污水中的浓度升高[2]. 在水生环境中的质量浓度范围在ng·L−1—μg·L−1[3]. 这些药物的持续排放不仅会威胁生态环境健康,还会对城市生活饮用水安全构成潜在风险[4]. 因此,迫切需要探索去除水生环境中残留莫诺拉韦、巴瑞替尼的新技术.
传统的污水处理方法(如混凝、沉淀、过滤和消毒)对其去除效果有限[5]. Kuroda[6]的研究表明,传统污水处理厂对羟氯喹(Hydroxychloroquine)、瑞德西韦(Remdesivir)、利巴韦林(Ribavirin)等抗新冠肺炎药物及其代谢物的去除率小于20%. 近年来,高级氧化工艺(AOPs)因其具有将人为和生物难降解有机物转化为矿物质的潜力而越来越普遍地应用于饮用水消毒及污染物质的去除[7]. 紫外辐射具有提高自由基量子产率以及降低其选择性的能力. 因此,将紫外辐射结合氧化剂在AOPs中产生活性氧自由基已成为一种常用的催化活化方法[8]. 紫外/过氧化氢(UV/H2O2)和紫外/过硫酸盐(UV/PDS)可以有效降解药品和个人护理产品(PPCPs),具有高效、低耗的特点[9]. 然而,在极大促进污染物去除的同时,也出现了一些局限性,比如:UV/H2O2体系中H2O2难以消耗完全,因此需要一定的后续工艺来清理残留物,由此会造成成本的增加[10];而UV/PDS体系中,活性物质的产率低限制了其在水处理中的应用[11]. 紫外/氯工艺因其具有明显的节能优势和较高的吸光度、量子产率和氯残留,目前已被广泛应用于饮用水的消毒和循环水的净化[12].
一般来说,常规的氯化物包括次氯酸盐和单氯胺. Wang等[13]将传统的紫外/氯工艺(紫外/次氯酸盐(UV/HClO)、紫外/单氯胺(UV/NH2Cl))在成本效益、吸光度、自由基产率、去除新污染物效率方面与紫外/二氯异氰尿酸盐(UV/Dichloroisocyanurate, NaDCC)体系进行了对比. 结果表明,使用UV/NaDCC在纯水中实现ECs去除的成本比使用UV/HClO低4%—53%,比使用UV/NH2Cl低75%—95%,而使用UV/NaDCC在实际水中实现新污染物(ECs)去除的成本比使用UV/HClO低26%—91%,比使用UV/NH2Cl低89%—96%;与HClO和NH2Cl相比,NaDCC是一种平衡良好的化合物,具有较强的紫外吸收能力和中等的量子产率;UV/NaDCC覆盖了UV/HClO工艺中的所有自由基,且大多数自由基浓度在UV/NaDCC中显著高于其他紫外/氯工艺;在去除新污染物效率方面,UV/NaDCC在多种水基质中的降解性能优于传统的紫外/氯工艺,具有优异的降解效率. 该研究为水中污染物的去除提供了一个新的研究方向,但是在实验中验证该体系对抗病毒药物的降解往往耗时耗力,因此本研究以模型预测的方式探究了莫诺拉韦、巴瑞替尼在紫外/二氯异氰尿酸盐体系下的降解路径及中间产物的毒性.
本研究以UV/NaDCC作为降解体系,采用Gaussian和Multiwfn计算了莫诺拉韦、巴瑞替尼的福井函数及简缩双描述符,预测了其在UV/NaDCC体系下的反应位点及降解路径,采用毒性评估软件ECOSAR、T.E.S.T.对莫诺拉韦、巴瑞替尼及其降解中间体和产物的急性毒性、慢性毒性、生物蓄积性、发育毒性进行定量构效关系(QSAR)预测.
1. 材料与方法(Material and Methods)
1.1 福井函数(FuKui function)计算
本研究通过GaussView5.0构建分子结构,计算任务为频率优化加结构分析(Opt+Freq),方法选择B3LYP,基组设置为6-311G,将计算任务提交Gaussian开始计算. 基于密度泛函理论(DFT)用Multiwfn[14]计算了Molnupiravir、Baricitinib的福井函数[15],根据福井函数的数值可以推测易发生亲电(f −)、亲核(f +)或者自由基攻击(f 0)的位点[16].
1.2 简缩双描述符(Condensed dual descriptor)计算
双描述符(DD)[17]是在概念密度泛函理论框架下定义的一种实空间函数,用于显示活性位点. DD为负值的区域易受亲电攻击,DD为正值的区域易受亲核攻击,在实际分析中,一个位点上的DD越正(负),该位点与亲核试剂(亲电试剂)反应的倾向就越大[18]. 双描述符与福井函数的区别是:双描述符可以同时展现亲电与亲核反应位点而不需要分别考察f +和f −.
1.3 风险评估和毒性测量
ECOSAR和Toxicity Estimation Software Tool(T.E.S.T)是由United States Environmental Protection Agency(EPA,美国环境保护署)开发的毒性评估软件. ECOSAR作为一种有效的预测程序已成功地应用于有机污染物的生态毒性评估[19],广泛用于水生生物毒性的估计;T.E.S.T能够使用定量结构活性关系(QSAR)方法估计化学品的毒性,预测结果与实测结果保持大体一致,具有较好的准确性[20]. 本研究利用ECOSAR程序对莫诺拉韦、巴瑞替尼分子及其降解产物进行毒性评估,预测了莫诺拉韦、巴瑞替尼及其降解产物对鱼类、绿藻和水蚤的急性毒性(LC50、EC50)和慢性毒性(Chv),毒性分为剧毒、有毒、有害和无害;本文还利用T.E.S.T软件(层级分析法)对其生物蓄积性、发育毒性进行了预测.
2. 结果与讨论(Results and discussion)
2.1 福井函数分析反应位点
基于福井函数的对比分析可以预测出反应的活性位点,其中,f −(f +)越大的位点,越容易受到亲电(亲核)攻击[21],而较高的f 0值表示容易受到自由基攻击. 在AOPs领域,自由基被认为是最重要的活性物质,因此f 0被广泛使用. 然而值得注意的是,f 0同时考虑了自由基的亲电性和亲核性,当自由基在反应中主要表现出亲电性时,f −更适合用于预测反应活性位点[22]. 在本研究中,由于氯自由基、氧自由基等强亲电性自由基的存在,亲电攻击是主要的攻击方式[23]. 莫诺拉韦的分子结构如图1(a)所示,福井函数如表1所示. O21(0.1123)、N20(0.0909)、C15(0.0770)的福井函数值最大,且被福井函数负等值面紧密包围(图1(a)). 因此,推测其为亲电攻击优先位点;巴瑞替尼的分子结构如图1(b)所示,由表2可知,C21(0.0859)、N14(0.0691)、C20(0.0666)的福井函数值最大,根据福井函数f −在电子密度等值面上的填色图(图1(b))可知,C20、C21被负等值面紧密包围,相比之下N14周围负等值面不够紧密,综合推测,C21、N14、C20为优先攻击位点,在反应体系中受到·OH和含氯自由基(Cl·,ClO·和Cl2·−)等自由基攻击的可能性更大.
表 1 莫诺拉韦福井函数值Table 1. FuKui function of MolnupiravirAtom q(N) q(N+1) q(N−1) f − f + f 0 1(C) −0.0845 −0.0901 −0.0796 0.0049 0.0056 0.0052 2(C) −0.0123 −0.0192 −0.0062 0.0061 0.0069 0.0065 3(C) −0.0797 −0.0884 −0.0747 0.0050 0.0086 0.0068 4(C) 0.2153 0.1667 0.2325 0.0172 0.0487 0.0329 5(O) −0.2707 −0.3152 −0.2203 0.0503 0.0445 0.0474 6(O) −0.1362 −0.1553 −0.1212 0.0150 0.0191 0.0170 7(C) 0.0305 0.0289 0.0333 0.0028 0.0017 0.0022 8(C) 0.0600 0.0534 0.0683 0.0083 0.0066 0.0074 9(C) 0.0481 0.0424 0.0604 0.0123 0.0056 0.0090 10(C) 0.0413 0.0384 0.0530 0.0118 0.0029 0.0073 11(C) 0.1036 0.0976 0.1164 0.0128 0.0059 0.0094 12(O) −0.1684 −0.1698 −0.1522 0.0163 0.0014 0.0088 13(N) −0.0240 −0.0657 0.0317 0.0557 0.0418 0.0487 14(C) 0.0404 −0.0844 0.0803 0.0399 0.1248 0.0824 15(C) −0.0921 −0.1503 −0.0151 0.0770 0.0581 0.0676 16(C) 0.1082 0.0310 0.1277 0.0195 0.0772 0.0483 17(N) −0.2017 −0.2597 −0.1738 0.0279 0.0580 0.0429 18(C) 0.1896 0.1645 0.2086 0.0190 0.0251 0.0220 19(O) −0.2926 −0.3475 −0.2412 0.0514 0.0550 0.0532 20(N) −0.0087 −0.0665 0.0822 0.0909 0.0577 0.0743 21(O) −0.1818 −0.2411 −0.0695 0.1123 0.0593 0.0858 22(O) −0.2376 −0.2555 −0.2062 0.0315 0.0179 0.0247 23(O) −0.2492 −0.2631 −0.2002 0.0490 0.0139 0.0314 表 2 巴瑞替尼福井函数值Table 2. FuKui function of BaricitinibAtom q(N) q(N+1) q(N−1) f − f + f 0 1(N) −0.1191 −0.1405 −0.1120 0.0071 0.0215 0.0143 2(C) −0.0098 −0.0184 −0.0066 0.0032 0.0086 0.0059 3(C) 0.0732 0.0684 0.0739 0.0008 0.0048 0.0028 4(C) −0.0086 −0.0205 −0.0019 0.0067 0.0119 0.0093 5(C) 0.0685 0.0613 0.0708 0.0023 0.0072 0.0047 6(C) −0.0201 −0.0293 −0.0141 0.0060 0.0093 0.0076 7(N) −0.1977 −0.2355 −0.1635 0.0342 0.0378 0.0360 8(N) 0.0325 0.0128 0.0747 0.0422 0.0197 0.0309 9(N) −0.1399 −0.1689 −0.0904 0.0495 0.0290 0.0392 10(C) −0.0001 −0.0222 0.0422 0.0423 0.0220 0.0322 11(C) −0.0480 −0.0650 −0.0043 0.0437 0.0170 0.0303 12(C) −0.0050 −0.0521 0.0325 0.0375 0.0471 0.0423 13(C) 0.0694 0.0245 0.1278 0.0584 0.0449 0.0517 14(N) −0.1955 −0.2647 −0.1264 0.0691 0.0692 0.0692 15(C) 0.0867 0.0626 0.1198 0.0331 0.0241 0.0286 16(C) −0.0433 −0.0637 −0.0132 0.0301 0.0204 0.0253 17(C) 0.0649 0.0074 0.0995 0.0346 0.0575 0.0460 18(N) −0.1783 −0.2157 −0.1402 0.0382 0.0374 0.0378 19(N) −0.0560 −0.0788 −0.0155 0.0405 0.0228 0.0316 20(C) −0.0872 −0.1100 −0.0206 0.0666 0.0228 0.0447 21(C) −0.0011 −0.0596 0.0848 0.0859 0.0585 0.0722 22(S) 0.4869 0.4517 0.4949 0.0080 0.0351 0.0216 23(O) −0.3275 −0.3782 −0.3058 0.0217 0.0506 0.0362 24(O) −0.3377 −0.3862 −0.3203 0.0174 0.0485 0.0329 25(C) −0.0090 −0.0361 −0.0020 0.0070 0.0271 0.0171 26(C) −0.0796 −0.0874 −0.0770 0.0026 0.0078 0.0052 2.2 简缩双描述符分析
为了进一步验证上述预测的易受攻击的反应位点,本研究采用Hirshfeld电荷在Multiwfn中接着计算了简缩双描述符(CDD),以定量比较每个位点易受攻击的可能性. CDD为负值的区域易受亲电攻击,而CDD为正值的区域更容易发生亲核反应. 与福井函数类似,本研究也绘制了基于双描述符的等值面图,便于综合分析各个位点受到攻击的可能性. 由表3(a)可以发现,在莫诺拉韦中O21(−0.0529)、O23(−0.0352)、N20(−0.0332)的CDD值最负,因此初步推测这3个位点更易受到亲电攻击,发生亲电反应,根据表3(a)中莫诺拉韦基于双描述符的等值面图可知,N20、O21、C15受到负等值面的紧密包围,易受到亲电攻击,相比之下O23受到亲电攻击的可能性会小一些. 在表3(b)中,巴瑞替尼C20(−0.0439)、C21(−0.0274)、C11(−0.0267)具有最负的CDD值,推测这3个位点更容易受到亲电攻击,结合等值面图(表3(b))来看,C11、C20受到较大负等值面包围,C21也在一定程度上被负等值面包围.
表 3 莫诺拉韦和巴瑞替尼简缩双描述符及等值面图Table 3. Condensed dual descriptor and Isosurface of Molnupiravir and Baricitinib(a) 
Atom CDD Atom CDD 1(C) 0.0007 22(O) −0.0136 2(C) 0.0008 23(O) −0.0352 3(C) 0.0036 24(H) 0.0015 4(C) 0.0315 25(H) 0.0009 5(O) −0.0059 26(H) 0.0007 6(O) 0.0040 27(H) 0.0041 7(C) −0.0011 28(H) 0.0030 8(C) −0.0017 29(H) 0.0043 9(C) −0.0067 30(H) 0.0019 10(C) −0.0089 31(H) −0.0022 11(C) −0.0069 32(H) −0.0010 12(O) −0.0149 33(H) −0.0016 13(N) −0.0140 34(H) −0.0106 14(C) 0.0849 35(H) −0.008 15(C) −0.0189 36(H) −0.0075 16(C) 0.0577 37(H) 0.0239 17(N) 0.0301 38(H) 0.0031 18(C) 0.0062 39(H) −0.0036 19(O) 0.0036 40(H) −0.0082 20(N) −0.0332 41(H) −0.0034 21(O) −0.0529 42(H) −0.0068 (b) 
Atom CDD Atom CDD 1(N) 0.0144 22(S) 0.0271 2(C) 0.0054 23(O) 0.0289 3(C) 0.0040 24(O) 0.0312 4(C) 0.0051 25(C) 0.0201 5(C) 0.0049 26(C) 0.0052 6(C) 0.0033 27(H) 0.0029 7(N) 0.0037 28(H) 0.0070 8(N) −0.0225 29(H) 0.0043 9(N) −0.0205 30(H) 0.0057 10(C) −0.0203 31(H) 0.0041 11(C) −0.0267 32(H) 0.0049 12(C) 0.0096 33(H) −0.0056 13(C) −0.0135 34(H) 0.0015 14(N) 0.0001 35(H) −0.0042 15(C) −0.0089 36(H) −0.0073 16(C) −0.0097 37(H) −0.0139 17(C) 0.0229 38(H) −0.0078 18(N) −0.0008 39(H) 0.0082 19(N) −0.0178 40(H) 0.0091 20(C) −0.0439 41(H) 0.0081 21(C) −0.0274 42(H) 0.0054 结合福井函数及简缩双描述符对易受到亲电攻击位点的预测发现,两种函数对反应位点的预测吻合较好. 综合分析,在莫诺拉韦中O21、N20发生反应的可能性最大,其中C15也有发生反应的可能;在巴瑞替尼中,最有可能发生反应的位点是C20、C21,其中C11也存在发生反应的可能.
2.3 基于福井函数及简缩双描述符模拟紫外/二氯异氰尿酸盐体系下的降解产物
在紫外/二氯异氰尿酸盐体系下,·OH和RCSs对莫诺拉韦的降解起主导作用,根据福井函数及简缩双描述的评价结果可知:在羟胺转变为仲胺的光照条件下,O21位点首先发生脱水反应形成PM-1,PM-1在·OH 攻击下在N20位点发生取代反应转化为PM-2,随着反应的进一步进行,PM-2在N13位点发生裂解反应生成PM-3和PM-7,随后PM-3经过质子化反应脱水转化为PM-4,PM-4发生取代反应转化为PM-5、PM-6. 路径2的降解路径符合上述理论分析且与Paymode[24]的部分研究结果类似. 经研究,本文预测的莫诺拉韦降解路径(图2(a))与Jain等[25]在实验室尺度上预测的路径基本一致. 由于目前对莫诺拉韦的研究较少且多聚焦于莫诺拉韦的高效合成[26],在实验室尺度上对莫诺拉韦降解途径的研究还仍然缺乏,本研究的预测是基于机理性的,尚存在较大局限性. 巴瑞替尼的降解路径如图2(b)所示,路径1中,C20、C21首先被氢氧化物攻击导致C20-C21键断裂生成PB-1,PB-1进一步去质子化生成PB-2,PB-2在分子氧存在下进行环氧化反应生成PB-3,结合福井函数及简缩双描述符分析结果,C11也存在发生反应的可能,因此预测PB-3在自由基攻击下会发生断裂,生成PB-4、PB-5. 相似地,路径2、路径3的猜测也是基于前文分析的结果,降解路径预测的总体趋势符合Chaganti[27]的降解实验分析. 综合来看,福井函数以及简缩双描述符能够较好地反映出易发生反应的位点.
2.4 莫诺拉韦、巴瑞替尼降解产物毒性分析
根据不同的降解路径,本文将莫诺拉韦及其产物分为2组,巴瑞替尼及其产物分为3组. 为了雷达图的直观性,本文将LC50大于10000 mg·L−1显著无害的降解产物统一取值为8000 mg·L−1来作图;巴瑞替尼降解路径2中产物PB-9处于显著无害状态,在图中并未显示. 根据欧盟制定的毒性分级标准,利用ECOSAR预测发现,莫诺拉韦对鱼类、水蚤和绿藻的LC50或EC50分别为2.18×103、6.31×103、5.42×103 mg·L−1,均处于无害状态;莫诺拉韦对鱼类、水蚤和绿藻的慢性毒性也处于无害状态. 虽然莫诺拉韦对鱼类、水蚤和绿藻没有毒性,但它可能加速抗病毒药物耐药性的建立,因此其去除是必要的,同时也需要关注降解产物的毒性变化. 如图3(a)路径1所示,PM-2对鱼类的急性毒性为有害状态,对绿藻的急性毒性达到有毒状态;PM-4对鱼类、水蚤、绿藻的急性毒性均达到有害水平,对水蚤、绿藻的慢性毒性达到有害水平,其中对鱼类的慢性毒性达到有毒水平. 路径2中PM-7对鱼类、绿藻的急性毒性分别为有害、有毒水平;PM-9对水蚤和绿藻的急慢性毒性均达到有害水平. 在所有降解路径中绿藻对该产品更为敏感,这与Jiang关于利巴韦林的研究保持一致[28]. 巴瑞替尼对鱼类、水蚤和绿藻的LC50或EC50分别为7.05×103、8.66×102、27.6 mg·L−1,对鱼类、水蚤无害,对绿藻表现出有害;对鱼类和绿藻表现出慢性毒性.
如图3(c)、3(d)、3(e)所示,在路径1中,PB-4对水蚤的慢性毒性为有害;PB-5对水蚤的急慢性毒性处于有害水平,对绿藻的急性毒性达到有毒水平;PB-11对绿藻的急性毒性处于有毒状态,对鱼类的慢性毒性达到有毒水平,对水蚤和绿藻都有害;路径2中,除PB-11外,PB-10对绿藻的急性毒性达到有害状态,对鱼类、水蚤和绿藻的慢性毒性也达到有害水平. 部分降解产物表现出比母体物质更高的毒性,类似现象已在许多研究中发现[29 − 31]. 这可能是由于在本研究中降解产物还未达到毒性降低的转折点[32]. 预测产物的毒性在降解过程中会有所增加,表明UV/NaDCC在莫诺拉韦和巴瑞替尼的降解中存在一定风险. 因此,有毒中间产物的去除可能是UV/NaDCC实际应用于抗病毒药物降解的关键.
由于莫诺拉韦、巴瑞替尼在疫情期间的大量使用,定性评价其毒性往往具有局限性. 因此,可以利用生物蓄积性、发育毒性等参数来描述莫诺拉韦和巴瑞替尼在水中毒性的大致变化. 经过T.E.S.T预测,莫诺拉韦和巴瑞替尼生物蓄积性和发育毒性预测结果如图4所示. 莫诺拉韦的生物蓄积性为0.0357和明显的发育毒性(0.83),经路径1降解后产物PM-5生物蓄积性增高(2.82)、发育毒性降至无毒(0.46),PM-6生物蓄积性未检出、发育毒性略有降低(0.82);降解路径2中,PM-9生物蓄积性未检出、发育毒性从0.83降至0.51(PM-9). 巴瑞替尼具有较强的生物蓄积性(3.53)和明显的发育毒性(0.78),在路径1、路径2中产物PB-11、PB-12生物蓄积性分别降至0.81(PB-11)和3.1(PB-12),经路径3降解后,巴瑞替尼降解产物PB-17生物蓄积性有所增加(3.91),发育毒性降至0.74. 除莫诺拉韦降解产物PM-5、巴瑞替尼降解产物PB-17生物蓄积性有所增加,其余降解产物生物蓄积性和发育毒性均有所降低. 莫诺拉韦、巴瑞替尼较高的生物蓄积性和发育毒性将导致其以较高生物活性的形式持续存在于水生环境中,对水生生态系统和生物健康带来威胁,因此在充分考虑有毒中间产物去除的前提下其降解是必要的.
3. 结论(Conclusion)
本文通过Gaussian09和Multiwfn程序计算了莫诺拉韦和巴瑞替尼的福井函数、简缩双描述符,结果表明,f −较大的点与CDD越负的位点相吻合. 结合福井函数及简缩双描述符预测发现,莫诺拉韦中O21、N20、C15,巴瑞替尼中C20、C21、C11是UV/NaDCC中的反应活性位点. 进而通过所预测的活性位点推测了莫诺拉韦和巴瑞替尼可能的降解路径. 利用ECOSAR、T.E.S.T程序对莫诺拉韦、巴瑞替尼分子及其降解产物在水生环境中的行为做出预测,部分降解产物表现出比母体物质更高的急、慢性毒性,综合其在环境中的生物蓄积性、发育毒性来看:莫诺拉韦降解产物PM-5、巴瑞替尼降解产物PB-17生物蓄积性有所增加,其余降解产物生物蓄积性和发育毒性均有所降低. 因此环境中莫诺拉韦、巴瑞替尼的降解应重点关注其有毒中间产物的产生以及去除.
-
表 1 莫诺拉韦福井函数值
Table 1. FuKui function of Molnupiravir
Atom q(N) q(N+1) q(N−1) f − f + f 0 1(C) −0.0845 −0.0901 −0.0796 0.0049 0.0056 0.0052 2(C) −0.0123 −0.0192 −0.0062 0.0061 0.0069 0.0065 3(C) −0.0797 −0.0884 −0.0747 0.0050 0.0086 0.0068 4(C) 0.2153 0.1667 0.2325 0.0172 0.0487 0.0329 5(O) −0.2707 −0.3152 −0.2203 0.0503 0.0445 0.0474 6(O) −0.1362 −0.1553 −0.1212 0.0150 0.0191 0.0170 7(C) 0.0305 0.0289 0.0333 0.0028 0.0017 0.0022 8(C) 0.0600 0.0534 0.0683 0.0083 0.0066 0.0074 9(C) 0.0481 0.0424 0.0604 0.0123 0.0056 0.0090 10(C) 0.0413 0.0384 0.0530 0.0118 0.0029 0.0073 11(C) 0.1036 0.0976 0.1164 0.0128 0.0059 0.0094 12(O) −0.1684 −0.1698 −0.1522 0.0163 0.0014 0.0088 13(N) −0.0240 −0.0657 0.0317 0.0557 0.0418 0.0487 14(C) 0.0404 −0.0844 0.0803 0.0399 0.1248 0.0824 15(C) −0.0921 −0.1503 −0.0151 0.0770 0.0581 0.0676 16(C) 0.1082 0.0310 0.1277 0.0195 0.0772 0.0483 17(N) −0.2017 −0.2597 −0.1738 0.0279 0.0580 0.0429 18(C) 0.1896 0.1645 0.2086 0.0190 0.0251 0.0220 19(O) −0.2926 −0.3475 −0.2412 0.0514 0.0550 0.0532 20(N) −0.0087 −0.0665 0.0822 0.0909 0.0577 0.0743 21(O) −0.1818 −0.2411 −0.0695 0.1123 0.0593 0.0858 22(O) −0.2376 −0.2555 −0.2062 0.0315 0.0179 0.0247 23(O) −0.2492 −0.2631 −0.2002 0.0490 0.0139 0.0314 表 2 巴瑞替尼福井函数值
Table 2. FuKui function of Baricitinib
Atom q(N) q(N+1) q(N−1) f − f + f 0 1(N) −0.1191 −0.1405 −0.1120 0.0071 0.0215 0.0143 2(C) −0.0098 −0.0184 −0.0066 0.0032 0.0086 0.0059 3(C) 0.0732 0.0684 0.0739 0.0008 0.0048 0.0028 4(C) −0.0086 −0.0205 −0.0019 0.0067 0.0119 0.0093 5(C) 0.0685 0.0613 0.0708 0.0023 0.0072 0.0047 6(C) −0.0201 −0.0293 −0.0141 0.0060 0.0093 0.0076 7(N) −0.1977 −0.2355 −0.1635 0.0342 0.0378 0.0360 8(N) 0.0325 0.0128 0.0747 0.0422 0.0197 0.0309 9(N) −0.1399 −0.1689 −0.0904 0.0495 0.0290 0.0392 10(C) −0.0001 −0.0222 0.0422 0.0423 0.0220 0.0322 11(C) −0.0480 −0.0650 −0.0043 0.0437 0.0170 0.0303 12(C) −0.0050 −0.0521 0.0325 0.0375 0.0471 0.0423 13(C) 0.0694 0.0245 0.1278 0.0584 0.0449 0.0517 14(N) −0.1955 −0.2647 −0.1264 0.0691 0.0692 0.0692 15(C) 0.0867 0.0626 0.1198 0.0331 0.0241 0.0286 16(C) −0.0433 −0.0637 −0.0132 0.0301 0.0204 0.0253 17(C) 0.0649 0.0074 0.0995 0.0346 0.0575 0.0460 18(N) −0.1783 −0.2157 −0.1402 0.0382 0.0374 0.0378 19(N) −0.0560 −0.0788 −0.0155 0.0405 0.0228 0.0316 20(C) −0.0872 −0.1100 −0.0206 0.0666 0.0228 0.0447 21(C) −0.0011 −0.0596 0.0848 0.0859 0.0585 0.0722 22(S) 0.4869 0.4517 0.4949 0.0080 0.0351 0.0216 23(O) −0.3275 −0.3782 −0.3058 0.0217 0.0506 0.0362 24(O) −0.3377 −0.3862 −0.3203 0.0174 0.0485 0.0329 25(C) −0.0090 −0.0361 −0.0020 0.0070 0.0271 0.0171 26(C) −0.0796 −0.0874 −0.0770 0.0026 0.0078 0.0052 表 3 莫诺拉韦和巴瑞替尼简缩双描述符及等值面图
Table 3. Condensed dual descriptor and Isosurface of Molnupiravir and Baricitinib
(a) Atom CDD Atom CDD 1(C) 0.0007 22(O) −0.0136 2(C) 0.0008 23(O) −0.0352 3(C) 0.0036 24(H) 0.0015 4(C) 0.0315 25(H) 0.0009 5(O) −0.0059 26(H) 0.0007 6(O) 0.0040 27(H) 0.0041 7(C) −0.0011 28(H) 0.0030 8(C) −0.0017 29(H) 0.0043 9(C) −0.0067 30(H) 0.0019 10(C) −0.0089 31(H) −0.0022 11(C) −0.0069 32(H) −0.0010 12(O) −0.0149 33(H) −0.0016 13(N) −0.0140 34(H) −0.0106 14(C) 0.0849 35(H) −0.008 15(C) −0.0189 36(H) −0.0075 16(C) 0.0577 37(H) 0.0239 17(N) 0.0301 38(H) 0.0031 18(C) 0.0062 39(H) −0.0036 19(O) 0.0036 40(H) −0.0082 20(N) −0.0332 41(H) −0.0034 21(O) −0.0529 42(H) −0.0068 (b) Atom CDD Atom CDD 1(N) 0.0144 22(S) 0.0271 2(C) 0.0054 23(O) 0.0289 3(C) 0.0040 24(O) 0.0312 4(C) 0.0051 25(C) 0.0201 5(C) 0.0049 26(C) 0.0052 6(C) 0.0033 27(H) 0.0029 7(N) 0.0037 28(H) 0.0070 8(N) −0.0225 29(H) 0.0043 9(N) −0.0205 30(H) 0.0057 10(C) −0.0203 31(H) 0.0041 11(C) −0.0267 32(H) 0.0049 12(C) 0.0096 33(H) −0.0056 13(C) −0.0135 34(H) 0.0015 14(N) 0.0001 35(H) −0.0042 15(C) −0.0089 36(H) −0.0073 16(C) −0.0097 37(H) −0.0139 17(C) 0.0229 38(H) −0.0078 18(N) −0.0008 39(H) 0.0082 19(N) −0.0178 40(H) 0.0091 20(C) −0.0439 41(H) 0.0081 21(C) −0.0274 42(H) 0.0054 -
[1] LAMONTAGNE F, STEGEMANN M, AGARWAL A, et al. A living WHO guideline on drugs to prevent covid-19[J]. BMJ, 2021, 372: n526. [2] HUANG T H, GUO J J, LU G. Ultraviolet-coupled advanced oxidation processes for anti-COVID-19 drugs treatment: Degradation mechanisms, transformation products and toxicity evolution[J]. Chemosphere, 2022, 303(Pt 1): 134968. [3] KUMAR M, KURODA K, DHANGAR K, et al. Potential emergence of antiviral-resistant pandemic viruses via environmental drug exposure of animal reservoirs[J]. Environmental Science & Technology, 2020, 54(14): 8503-8505. [4] 王晓虹, 何桂琳, 田立平, 等. 抗病毒药物在水体中的污染现状与去除技术进展[J]. 工业水处理, 2022, 42(3): 47-54. WANG X H, HE G L, TIAN L P, et al. Pollution status and removal technology progress of antiviral drugs in water[J]. Industrial Water Treatment, 2022, 42(3): 47-54 (in Chinese).
[5] YANG X, SUN J L, FU W J, et al. PPCP degradation by UV/chlorine treatment and its impact on DBP formation potential in real waters[J]. Water Research, 2016, 98: 309-318. doi: 10.1016/j.watres.2016.04.011 [6] KURODA K, LI C, DHANGAR K, et al. Predicted occurrence, ecotoxicological risk and environmentally acquired resistance of antiviral drugs associated with COVID-19 in environmental waters[J]. Science of the Total Environment, 2021, 776: 145740. doi: 10.1016/j.scitotenv.2021.145740 [7] AO X W, ELORANTA J, HUANG C H, et al. Peracetic acid-based advanced oxidation processes for decontamination and disinfection of water: A review[J]. Water Research, 2021, 188: 116479. doi: 10.1016/j.watres.2020.116479 [8] LI R X, WANG J Q, WU H, et al. Periodate activation for degradation of organic contaminants: Processes, performance and mechanism[J]. Separation and Purification Technology, 2022, 292: 120928. doi: 10.1016/j.seppur.2022.120928 [9] GUO K H, WU Z H, YAN S W, et al. Comparison of the UV/chlorine and UV/H2O2 processes in the degradation of PPCPs in simulated drinking water and wastewater: Kinetics, radical mechanism and energy requirements[J]. Water Research, 2018, 147: 184-194. doi: 10.1016/j.watres.2018.08.048 [10] SHARMA A, AHMAD J, FLORA S J S. Application of advanced oxidation processes and toxicity assessment of transformation products[J]. Environmental Research, 2018, 167: 223-233. doi: 10.1016/j.envres.2018.07.010 [11] MIKLOS D B, WANG W L, LINDEN K G, et al. Comparison of UV-AOPs (UV/H2O2, UV/PDS and UV/Chlorine) for TOrC removal from municipal wastewater effluent and optical surrogate model evaluation[J]. Chemical Engineering Journal, 2019, 362: 537-547. doi: 10.1016/j.cej.2019.01.041 [12] ZHANG Z, CHUANG Y H, SZCZUKA A, et al. Pilot-scale evaluation of oxidant speciation, 1, 4-dioxane degradation and disinfection byproduct formation during UV/hydrogen peroxide, UV/free chlorine and UV/chloramines advanced oxidation process treatment for potable reuse[J]. Water Research, 2019, 164: 114939. doi: 10.1016/j.watres.2019.114939 [13] WANG J Q, ZHENG M, DU E D, et al. A novel source of radicals from UV/dichloroisocyanurate for surpassing abatement of emerging contaminants versus conventional UV/chlor(am)ine processes[J]. Environmental Science & Technology, 2023, 57(47): 18452-18461. [14] LU T, CHEN F W. Multiwfn: A multifunctional wavefunction analyzer[J]. Journal of Computational Chemistry, 2012, 33(5): 580-592. doi: 10.1002/jcc.22885 [15] LU T, CHEN Q. Realization of Conceptual Density Functional Theory and Information-Theoretic Approach in Multiwfn Program[M]//Conceptual Density Functional Theory. Wiley, 2022: 631-647. [16] ZHANG C, TIAN S H, QIN F Z, et al. Catalyst-free activation of permanganate under visible light irradiation for sulfamethazine degradation: Experiments and theoretical calculation[J]. Water Research, 2021, 194: 116915. doi: 10.1016/j.watres.2021.116915 [17] MORELL C, GRAND A, TORO-LABBÉ A. New dual descriptor for chemical reactivity[J]. The Journal of Physical Chemistry. A, 2005, 109(1): 205-212. doi: 10.1021/jp046577a [18] 罗婷. 基于密度泛函理论的高级氧化体系降解磺胺类抗生素的机理研究[D]. 广州: 华南理工大学, 2020. LUO T. Research on degradation mechanism of sulfonamides treated with advanced oxidation processes based on density functional theory[D]. Guangzhou: South China University of Technology, 2020 (in Chinese).
[19] GAO Y P, JI Y M, LI G Y, et al. Theoretical investigation on the kinetics and mechanisms of hydroxyl radical-induced transformation of parabens and its consequences for toxicity: Influence of alkyl-chain length[J]. Water Research, 2016, 91: 77-85. doi: 10.1016/j.watres.2015.12.056 [20] SUN Y H, LI M, HADIZADEH M H, et al. OH/O3-initiated transformation of primidone in AOPs based on the theoretical calculations: Mechanisms, kinetics, and eco-toxicity assessments[J]. Journal of Environmental Chemical Engineering, 2023, 11(1): 109167. doi: 10.1016/j.jece.2022.109167 [21] LI Y Y, YANG Y, LEI J M, et al. The degradation pathways of carbamazepine in advanced oxidation process: A mini review coupled with DFT calculation[J]. Science of the Total Environment, 2021, 779: 146498. doi: 10.1016/j.scitotenv.2021.146498 [22] LIANG J L, ZHEN P, GAN P F, et al. DFT calculation of nonperiodic small molecular systems to predict the reaction mechanism of advanced oxidation processes: Challenges and perspectives[J]. ACS ES&T Engineering, 2023,DOI:10.1021/acsestengg.3c00204. [23] PARSAEE F, SENARATHNA M C, KANNANGARA P B, et al. Radical philicity and its role in selective organic transformations[J]. Nature Reviews Chemistry, 2021, 5(7): 486-499. doi: 10.1038/s41570-021-00284-3 [24] PAYMODE D J, VASUDEVAN N, AHMAD S, et al. Toward a practical, two-step process for molnupiravir: Direct hydroxamination of cytidine followed by selective esterification[J]. Organic Process Research & Development, 2021, 25(8): 1822-1830. [25] JAIN S, GIRI S, SHARMA N, et al. LC and LC-HRMS studies on stability behavior of molnupiravir an anti-COVID 19 drug[J]. Journal of Liquid Chromatography & Related Technologies, 2021, 44(15/16): 750-759. [26] LIU Z, YANG J, LIU F W. New routes to antiviral molnupiravir against SARS-CoV-2 infection[J]. Chinese Journal of Organic Chemistry, 2022, 42(9): 2988. doi: 10.6023/cjoc202203044 [27] CHAGANTI S, DHIMAN V, MADHYANAPU GOLLA V, et al. Forced degradation study of baricitinib and structural characterization of its degradation impurities by high-resolution mass spectrometry and nuclear magnetic resonance spectroscopy[J]. Rapid Communications in Mass Spectrometry, 2023, 37(18): e9605. doi: 10.1002/rcm.9605 [28] JIANG J C, AN Z X, LI M X, et al. Comparison of ribavirin degradation in the UV/H2O2 and UV/PDS systems: Reaction mechanism, operational parameter and toxicity evaluation[J]. Journal of Environmental Chemical Engineering, 2023, 11(1): 109193. doi: 10.1016/j.jece.2022.109193 [29] ZHANG X K, GUO J J, HUANG Y, et al. Toxicity evolution and control for the UV/H2O2 degradation of nitrogen-containing heterocyclic compounds: SDZ and PMM[J]. Chemosphere, 2023, 338: 139541. doi: 10.1016/j.chemosphere.2023.139541 [30] FENG L, SONG W W, OTURAN N, et al. Electrochemical oxidation of Naproxen in aqueous matrices: Elucidating the intermediates’ eco-toxicity, by assessing its degradation pathways via experimental and density functional theory (DFT) approaches[J]. Chemical Engineering Journal, 2023, 451: 138483. doi: 10.1016/j.cej.2022.138483 [31] 龚卓炫, 何欣, 乔显亮. 铁活化过硫酸盐降解抗生素的动力学、降解产物和毒性评估[J]. 生态毒理学报, 2023, 18(4): 34-44. GONG Z X, HE X, QIAO X L. Kinetics, degradation products and toxicity assessment of iron-activated persulfate degradation of antibiotics[J]. Asian Journal of Ecotoxicology, 2023, 18(4): 34-44 (in Chinese).
[32] JI Q Y, SUN D Y, YANG S G, et al. Oxidation degradation of tri(dichloropropyl) phosphate by UV/H2O2 system: Degradation pathways and risk assessment of intermediates[J]. Journal of Environmental Chemical Engineering, 2020, 8(6): 104513. doi: 10.1016/j.jece.2020.104513 期刊类型引用(0)
其他类型引用(1)
-





 下载:
下载: