-
阿特拉津(2-Chloro-4-ethylamino-6-isopropylamino-s-triazine,ATZ)是一种通过阻断叶绿体中质体醌结合蛋白和抑制光合作用来防治阔叶和禾草类杂草的选择性除草剂,土壤对其吸附性较低,因此极易向地表水、深层土壤和地下中迁移[1]. 低浓度的阿特拉津显著降低作物的株高、根长、根干重,高浓度的阿特拉津甚至会导致植物死亡[2];阿特拉津影响雄性斑马鱼的神经发育和神经功能[3],已成为一种污染源并对动植物和人类健康造成不可忽视的风险[4-5]. 由于阿特拉津严重的危害及影响,2004年在欧盟被禁止使用,但由于它价格低廉而在世界其他地区被广泛使用,仍是国际上销售的主要除草剂之一.
目前,环境中阿特拉津的去除方法主要有吸附法[6]、光催化法[7]、高级氧化法[8]、植物修复法[9-10]、生物法[11]等,其中基于微生物降解的生物修复法不仅效率高,而且几乎不损害生态环境,并且可将ATZ转化为无毒或低毒的物质.很多研究者已筛选出能将阿特拉津作为唯一氮源或碳源的微生物,如细菌Pseudomonas sp. strain AKN5[12]、Klebsiella sp. FH-1[13]、Citricoccus sp. strain TT3[14]、Arthrobacter sp. 30、Pseudomonas sp. AD39[15]和真菌Pleuroyus ostreatus INCQS 40310[16]等. 这些菌株对阿特拉津具有耐受性并能降解高浓度的阿特拉津,Enterobacter sp.LY-2[17]对浓度为100 mg·kg−1的污染土壤具有修复效果,14 d后阿特拉津浓度降低为9.9 mg·kg−1;产脲节杆菌(Arthrobacter ureafaciens)CS3,培养2 d 可将50 mg·L−1的阿特拉津完全降解[18]. 虽然目前报道的去除阿特拉津的微生物种类较多,但仍然存在停滞期长,降解耗时较长,耐受性差等不足。而且大部分菌株来源于中国北部寒冷地区,东北多为黑土,呈中性或偏碱性、有机质及氮磷含量丰富,而长三角地区雨量充沛、气温较高,土壤通透性差、呈弱酸性、有机质及氮磷含量较低,这些菌株可能不适应长三角的土壤环境,因此有必要寻找适合长三角地区的高效阿特拉津降解微生物资源.
本研究从江苏省5个不同地区的农田土壤中筛选分离能适应长三角地区阿特拉津污染土壤的菌株,研究ATZ初始浓度、温度和pH对菌株繁殖和降解效果的影响,判断菌株的适用环境范围,并采用HPLC-MS测定ATZ的降解产物,推断可能存在的降解转化途径.
-
阿特拉津(>97%)(C18H14ClN5,分子量214.69,密度为1.2 g·cm−1、沸点为200 ℃、熔点为174 ℃,难溶于水)、甲醇、乙醇、葡萄糖、C6H9Na3O9、C9H11NO、C6H7NO3S、C10H9N等. 无机盐培养基(MSM):Mg2SO4·7H2O 0.1 g·L−1、K2HPO4 1 g·L−1、KH2PO4 1 g·L−1、FeSO4·7H2O 0.025 g·L−1、CaCl2 0.025 g·L−1,适量阿特拉津;富集培养基(LB) :牛肉浸膏5 g·L−1、胰蛋白胨10 g·L−1、NaCl 5 g·L−1,加15 g·L−1琼脂即为固体培养基.
-
取江苏省5个长期喷洒阿特拉津的农田表层土壤(0—10 cm)进行降解菌株的驯化. 将土壤样品(5 g)分别加入150 mL MSM培养基中(ATZ浓度为50 mg·L−1),培养条件设置为 30 ℃、150 r·min−1. 驯化7 d后将上清液转至新的MSM培养基中,重复3次适应过程. 最后,将梯度稀释的培养液涂布于LB固体培养基,待菌落长成后,将其进行多次划线分离和纯化,并于无机盐培养基验证降解效果. 最终,筛选出一株能够降解阿特拉津的菌株D2,并采用斜面培养基和甘油保存.
-
将菌株接种于固体培养基,2—3 d后观察长出菌落的形态(形状、色泽、透明程度等);采用扫描电镜(Quanta FEG 250)观察菌株 D2放大50000倍后的表面形态.
-
革兰氏染色试验、水解产酸、柠檬酸盐利用、V-P、过氧化氢酶等指标,具体测定方法依据《污染控制微生物实验》[19],并参照《常见细菌系统鉴定手册》[20]与类似细菌进行比较.
-
将菌株在固体培养基中划线分离,待菌落长成后进行16S rDNA基因鉴定,由上海天霖生物科技有限公司完成. 将D2的 16Sr DNA 基因序列在BLAST系统中与基因库进行比对分析,并使用MEGA 7.0软件绘制系统发育树.
-
采用紫外可见分光光度计(INESA-N4)测定菌株生长不同时间段的OD600以推测菌株的生长情况,以原始溶液为空白对照绘制D2的生长曲线,并用 SGompertz模型[21](式(1))和Slogistic模型[22](式(3))拟合D2的生长曲线,并得到菌株最大比生长速率(μ1、μ2):
式中,t为时间(h),Nt1为时间t时菌株的数量,N0为初始菌株数量,Xc为达到最大升值速率的时间(h),μ1为菌株的最大比生长速率(h−1),a1为菌株最大生长量.
式中,t为时间(h),Nt2为时间t时菌株的数量,X0为达到最大升值速率的时间(h),μ2为菌株的最大比生长速率(h−1),a2为菌株最大生长量.
-
为了探究环境因素对菌株降解阿特拉津的影响,将菌悬液按照体积比为2%的量接种到含阿特拉津的MSM培养基中,研究不同初始浓度(10、20、50、100、150、200 mg·L−1)、不同pH(5.0、7.0、9.0)、不同温度(10、20、30、40 ℃)下菌株对阿特拉津的降解情况,以转速 150 r·min−1 避光培养,溶液经0.22 μm滤膜后测定阿特拉津的浓度.
-
阿特拉津的浓度采用岛津高效液相色谱仪(LC-20AT,Japan)测定. 检测条件:流动相为V(甲醇):V(水)=60:40,流速为1.0 mL·min−1,波长为220 nm. 降解产物定性分析采用带有高效液相色谱仪UltiMate 3000(Thermo Fisher Scientific,USA)和高分辨质谱仪5600 QTOF(AB SCIEX,Framingham,USA)的超高压液相质谱仪HPLC-MS,色谱柱(ACQUITY UPLC HSS T3 1.8 μm ,2.1 mm×100 mm),以水(含有2 mmol·L−1乙酸铵)和乙腈进行梯度洗脱. 条件:轰击能量,30 eV;雾化气压(GS1):60 Psi,辅助气压,60 Psi;气帘气压,35 Psi;温度,650 ℃;喷雾电压,5000 V(正离子模式)或−4000 V(负离子模式).
-
采用以阿特拉津(50 mg·L−1)为碳源和氮源的无机盐培养基对5个种植玉米的农田土壤经过驯化、分离,筛选出8株生长良好且菌落形态不同的菌株,最终选择一株降解效能最好的菌株,将其命名为D2.
菌株D2的菌落形态观察如图1(a),菌落较小,边缘整齐,浅黄色;革兰氏染色结果表明,D2为革兰氏阳性菌 (图1b ). 扫描电镜(×5000)下观察的菌株形态如图1(c)所示,D2呈杆状. 生理生化试验结果显示(表1),硝酸盐还原及过氧化氢酶试验均呈阳性,其余结果均为阴性.
将PCR扩增后得到的基因片段进行测序,测序结果在 NCBI 上经 BLAST 分析目标序列与同源序列,并绘制系统发育树(图2),对比发现ATZ降解菌株D2与土壤芽孢杆菌(Solibacillus)的核苷酸序列相似率高达99.58%,因此菌株D2经鉴定为土壤芽孢杆菌.
-
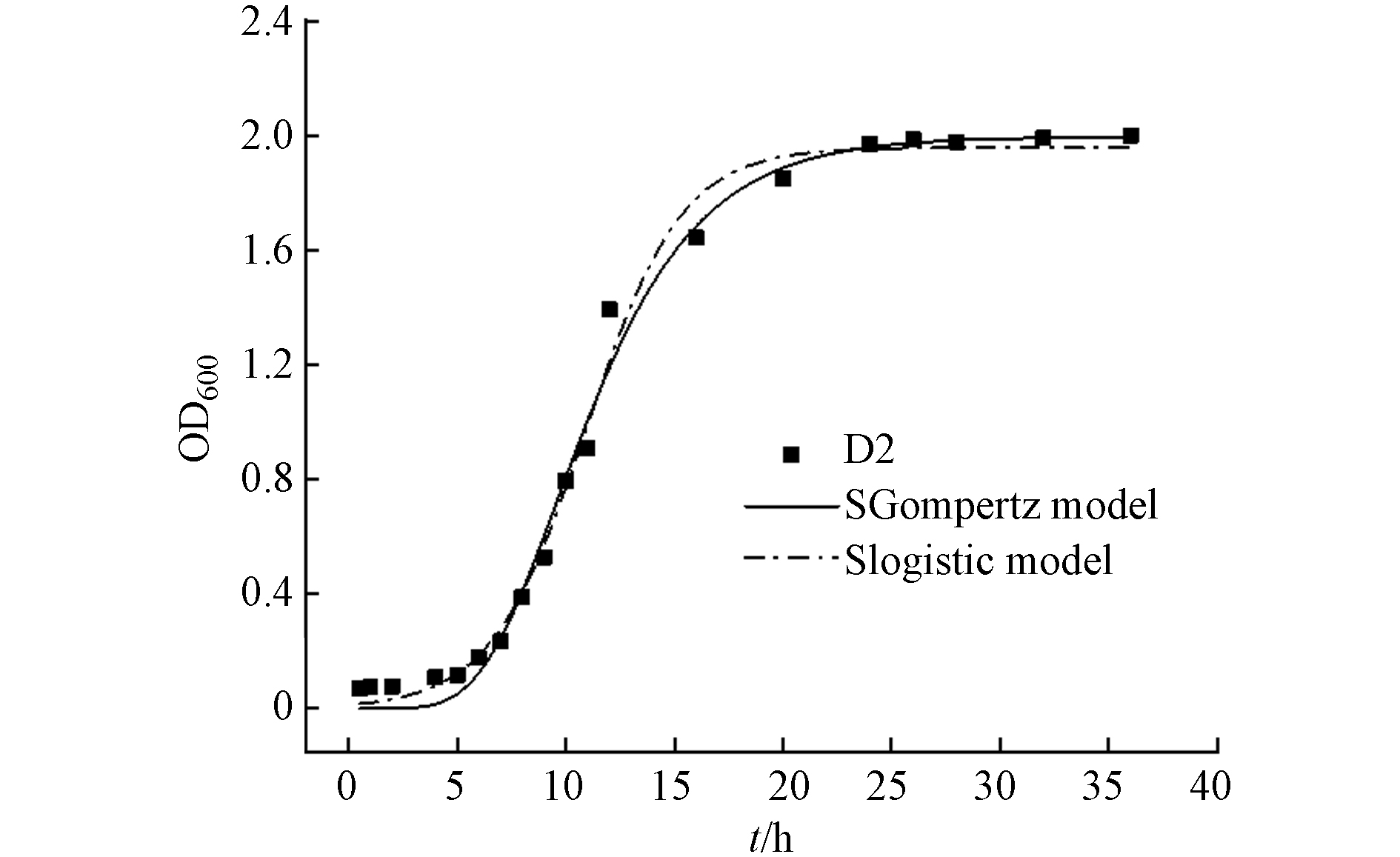
菌株D2的生长曲线见图3. 在LB培养基中,D2经过5 h的适应期后进入菌株代谢旺盛的对数期;12 h后OD600达到1.0,此时的菌株增长速率最快;24 h后达到生长繁殖的峰值,此后OD600不再增长,菌株数量相对趋于稳定.
采用SGompertz模型[23]和Slogistic模型拟合菌株的生长曲线(表2,图3). 结果显示,两种模型均可较好地拟合D2的生长曲线,拟合度(R2)分别为0.991和0.992,在第10 h左右D2到达最大比生长速率,随后比生长速率逐渐降低.
-
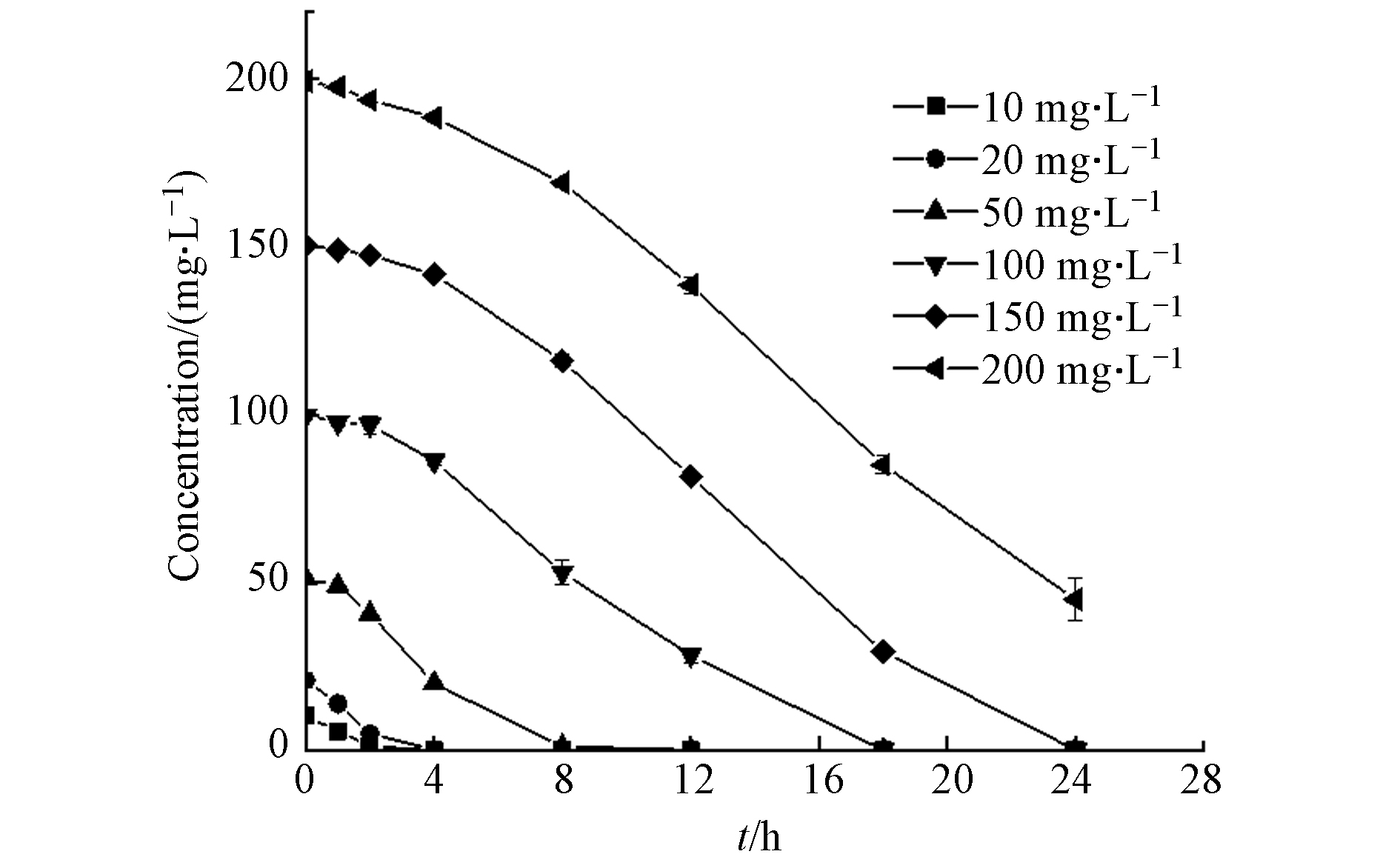
污染物浓度对菌株的影响是评价菌株生长及降解能力的标准之一[24],D2在不同初始浓度下对ATZ的降解效果如图4所示. 当浓度为200 mg·L−1时,D2对ATZ仍具有较高的去除能力,24 h的降解率为77.43%;初始浓度越低,D2的去除效率越高,将初始浓度为150、100、50、20、10 mg·L−1的ATZ完全降解分别需要24、18、8、4、3 h.长三角洲地区农田土壤中ATZ浓度的平均值为5.7 ng·L−1,检出率高达57.7%[25],菌株D2对ATZ的耐受性远高于环境中的平均浓度,因此它是一株在实际污染环境中具有应用价值的降解菌株.
-
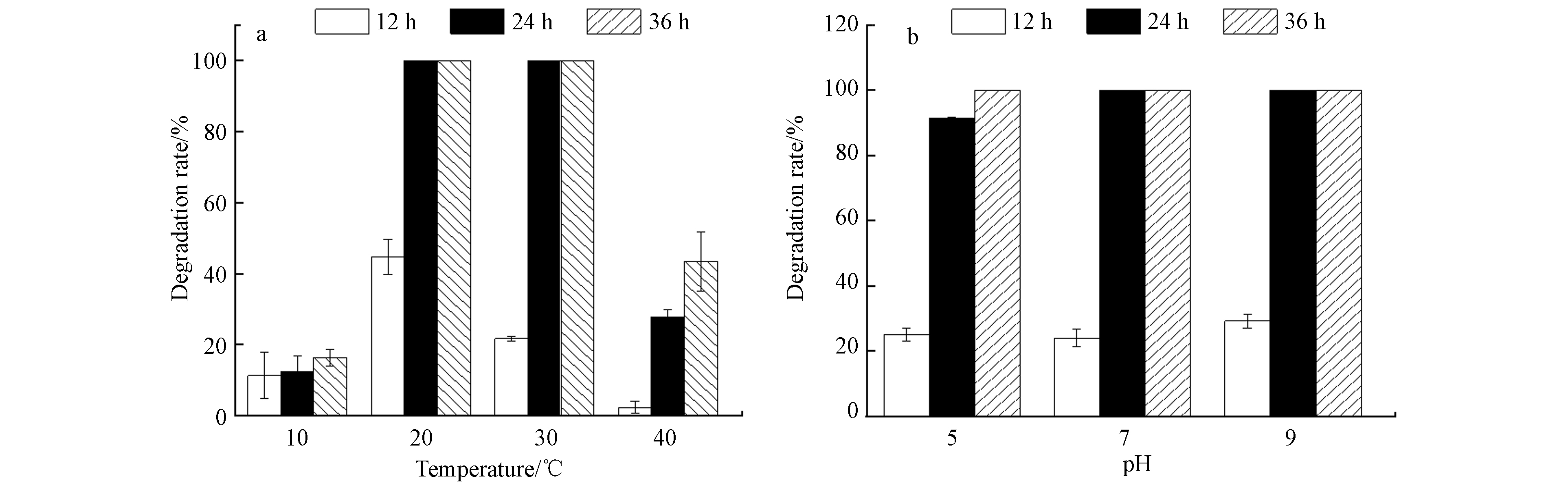
温度和pH会通过影响细菌的生长而影响其降解能力[26-28],不同温度(10、20、30、40 ℃)条件下,D2对初始浓度为100 mg·L−1 ATZ的降解情况见图5a. 当温度为20—30 ℃时,D2在24 h内将ATZ完全降解;但在10 ℃和40 ℃下培养36 h时,D2对ATZ的降解率仅为16.33%和43.48%,说明低温和高温都会抑制菌株的代谢作用,这与菌株的酶活性密切相关[29]. 另外,培养基的酸碱度也会影响细菌酶的合成和催化活性[9],细菌表面的电荷分布随着pH的改变而改变 [26]. 不同pH条件下,D2对初始浓度为100 mg·L−1(培养温度为30 ℃)ATZ的降解率如图5b所示. 当pH为5.0、7.0和9.0 时,经过24 h,D2对ATZ的降解率分别为到91.42%、100%、100%. 据以往研究报道,pH为5.0和9.0时,阿特拉津降解菌(Enterobacter sp.)的降解率低于70%[27],菌株L-6 [28] 仅能适应碱性环境,当pH=6时,降解率为45.6%,因此菌株D2对pH具有较高的适应性.
-
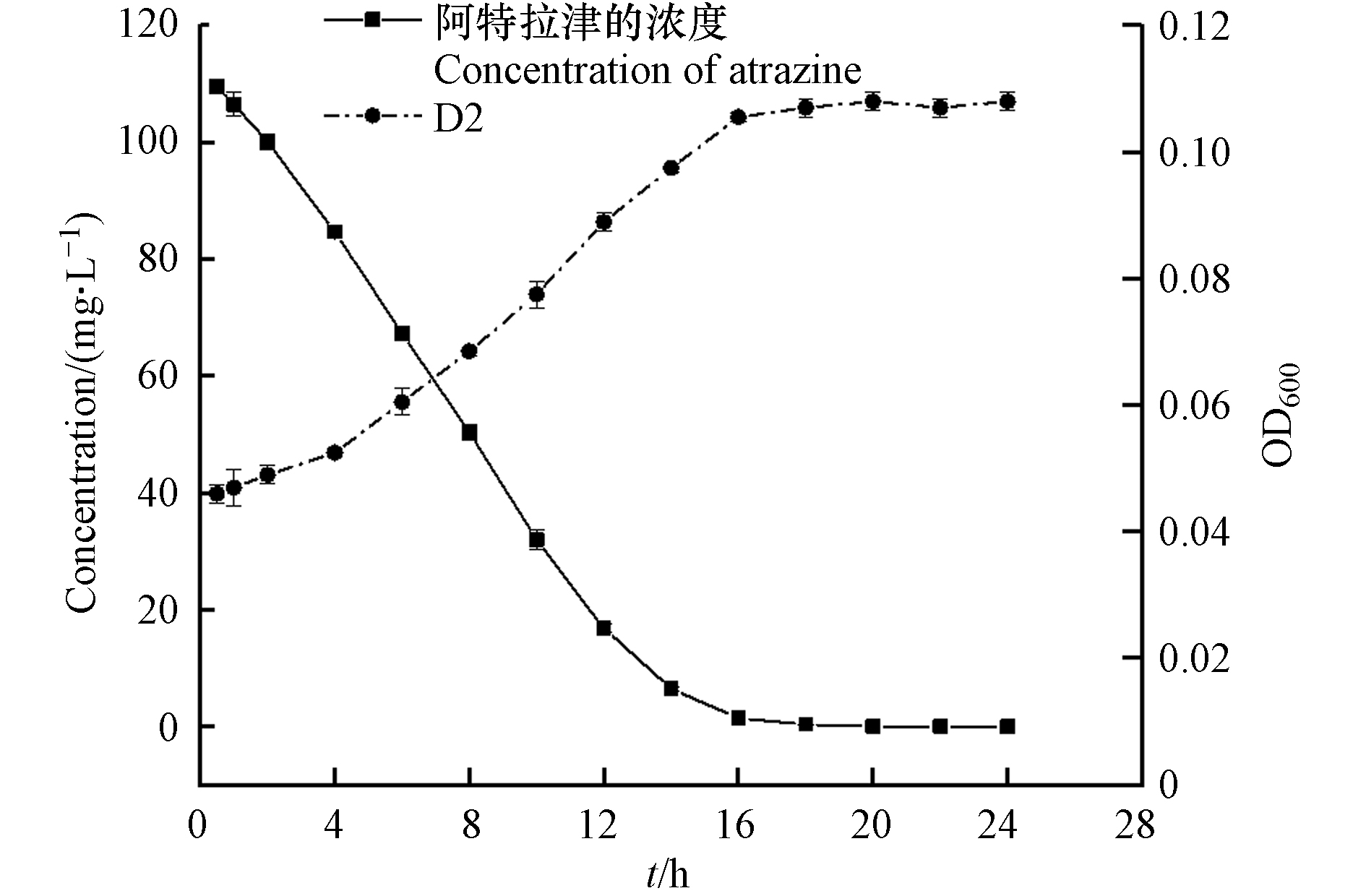
在最佳降解条件下(2%的菌悬液接种量、温度为20 ℃、pH为9.0),将菌株D2接种至初始浓度为100 mg·L−1的ATZ无机盐培养基中,生长及降解动态曲线如图6所示. 菌株D2的生长与ATZ浓度呈较强的负相关(表3,r=−0.983,P<0.01),随着菌株的生长,阿特拉津的浓度急剧降低. 培养初期D2存在短暂的适应期,OD600从0.046增至0.052;培养4 h后进入对数生长期,OD600达到0.097,此时随着菌株的大量生长ATZ被快速降解;在培养14 h以后菌株进入静止期,此时菌株数量达到最大值,其OD600为0.107,ATZ浓度降为6.41 mg·L−1,降解率达到94.33%,在18 h时ATZ被完全去除. 实验结果表明,阿特拉津浓度的降低与D2的数量密切相关,证明菌株D2具有阿特拉津去除能力.
-
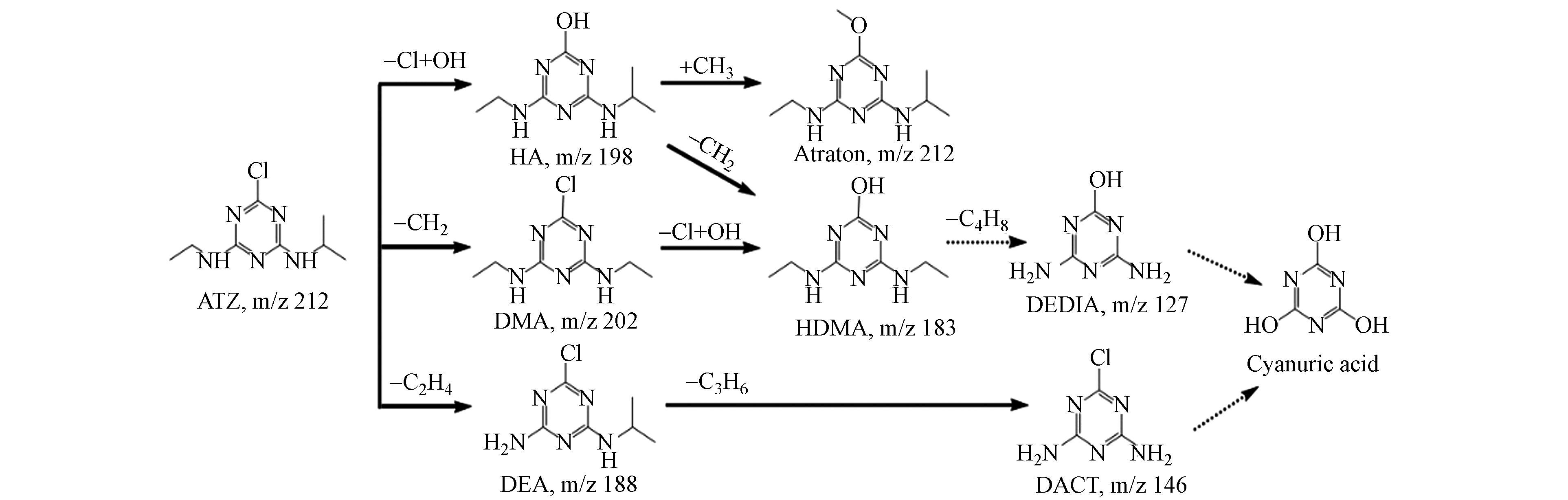
菌株D2于含ATZ的无机盐培养基进行培养,取12 h时的样品进行分析,采用高效液相色谱-质谱联用仪(HPLC-MS)测定其降解产物,共检测出6种可能的代谢物(图7 b—g),质荷比(m/z)分别为146.00、184.12、198.13、202.08、212.15、188.07,与已知标准化合物和报道的阿特拉津代谢物的比较,这6种代谢物分别被确定为脱乙基脱异丙基阿特拉津(DACT)、羟基西玛津(HDMA)、羟基阿特拉津(HA)、西玛津(DMA)、阿特拉通(Atraton)和脱乙基阿特拉津(DEA).
根据产物的结构组成和报道过的阿特拉津降解途径提出菌株D2降解阿特拉津的可能途径,结果如图8所示.
途径Ⅰ:首先羟基取代氯,形成脱氯羟基化的阿特拉津(HA),然后甲基取代HA的氢生成阿特拉通(Atraton),HA也可能通过脱甲基化反应转化为羟基西玛津(HDMA).途径Ⅱ:阿特拉津的异丙基脱甲基化生成西玛津(DMA),然后DMA的氯被一个羟基取代,转化为羟基西玛津(HDMA).途径Ⅲ:阿特拉津经N-脱烷基反应转化为脱乙基阿特拉津(DEA),随后DEA异丙基化为脱乙基脱异丙基阿特拉津(DACT). Liu等[30]在研究土壤中阿特拉津的降解途径时得到了相似的结果,阿特拉津可通过光解、水解及微生物降解转化为羟基阿特拉津、阿特拉通、扑灭津、脱乙基阿特拉津、羟基西玛津、西玛津、去乙基异丙基化阿特拉津等. 而Zhang 等[31]和郭火生等[32]还发现羟基阿特拉津可以被进一步水解为三聚氰酸.通过以上总结推测,阿特拉津的降解中间产物还会通过脱甲基化、脱烷基化和水解等产生三聚氰胺二酰胺(DEDIA)、去乙基异丙基化阿特拉津(DACT)和三聚氰酸等副产物.
-
将本研究中的D2与已报道过具有阿特拉津降解能力的菌株进行对比,结果汇总如表4所示. 目前已经筛选出很多能以阿特拉津为唯一碳源或氮源生长的菌株,并适用于阿特拉津浓度范围为8—100 mg·L−1的废水,但本研究中的新菌株D2具有更优异的降解性能. 已研究的菌株Acinetobacter lwoffii DNS32 [32]在 20 ℃和 35 ℃时,对阿特拉津的降解率约为 30%—35%,最佳生长pH为7—8,在酸性及pH高于8的条件下,菌株的生长及降解能力受到抑制;菌株C2[33]在20 ℃、30 ℃、40 ℃下培养5 d后,降解率分别为86.7%、97.6%和21.9%;菌株SB5[34]生长的温度范围是25—37 ℃,在pH=8的条件下完全降解ATZ需36 h. 本研究分离出的菌株D2在20 —30 ℃、pH为5.0 — 9.0的范围内,18 h以内即可将100 mg·L−1的阿特拉津完全降解,远远快于培养时间为24 —264 h的其他菌株,且具有更强的pH和温度的适应性,在40 ℃的高温下仍具有一定的活性,降解率高于40%,在10 ℃的条件下,D2对ATZ的降解率仍有近20%. 另外,已报道的菌株大部分来源于北部寒冷地区,而菌株D2来源于江苏省农田土壤,更能适应夏季高温、有机质及氮磷含量低、弱酸性的土壤环境,可成为长三角地区阿特拉津污染土壤和废水修复的微生物资源.
-
(1)从江苏省5个不同区域的农田土壤中筛选出一株以阿特拉津为唯一碳源和氮源生长的菌株D2,属于土壤芽孢杆菌(Solibacillus),该菌株能适应长三角地区环境,并有效降解水体和土壤中的阿特拉津.
(2)在pH 5.0—9.0、温度20—30 ℃的条件下,菌株具有良好的降解能力. 最适条件下,D2能在18 h内将100 mg·L−1的ATZ完全降解,具有较高的耐受性及降解效率.
(3)D2主要通过脱氯羟基化、加氢脱烷基化、甲基化、脱烷基化和水解等将ATZ转化为羟基阿特拉津(HA)、阿特拉通(Atraton)、脱乙基阿特拉津(DEA)、西玛津(DMA)、羟基西玛津(HDMA)和脱乙基脱异丙基阿特拉津(DACT).
一株高效阿特拉津降解菌株的筛选及其降解能力和机理
Screening and identification of an atrazine-degrading strain and its degradation capacity and mechanism on atrazine
-
摘要: 在江苏省某玉米农田土壤中筛选出以阿特拉津(ATZ)为唯一碳源和氮源的菌株D2,经16S rDNA基因序列分析,将其鉴定为土壤芽孢杆菌(Solibacillus),菌株D2的生长曲线符合SGompertz模型和Slogistic模型. 不同ATZ初始浓度、pH和培养温度条件下对ATZ的降解实验表明,菌株D2对ATZ具有极高的耐受性(>200 mg·L−1),在温度为20—30 ℃、pH值为5—9的条件下降解率均能达100%,可将100 mg·L−1的ATZ在18 h内完全去除,ATZ去除量与D2菌株的数量呈显著的负相关(r=−0.983,P<0.01). 对ATZ的降解中间产物测定表明,菌株D2可通过脱氯羟基化、加氢脱烷基化、甲基化、脱烷基化和水解等过程将阿特拉津转化为羟基阿特拉津(HA)、阿特拉通(atraton)、脱乙基阿特拉津(DEA)、西玛津(DMA)、羟基西玛津(HDMA)和脱乙基脱异丙基阿特拉津(DACT). 因此,D2是一株高效降解菌株,环境适应能力高于大部分已报道菌株,能够广泛应用于ATZ污染废水和污染土壤修复等领域.Abstract: Strain D2, using atrazine(ATZ) as the only carbon source and nitrogen source, was isolated from a maize field in Jiangsu Province. It was preliminarily identified as Solibacillus by 16S rDNA gene sequence analysis, and its growth curve both fit SGompertz model and Slogistic model. Experiments demonstrated that under different initial ATZ concentration, pH and temperature, strain D2 could tolerate high concentration of ATZ (>200 mg·L−1), wider pH and temperature, compared with reported strains. Strain D2 degraded ATZ from 100 mg·L−1 to 0 mg·L−1 within 18 h at 20—30 ℃, pH 5—9, and there was a significant negative correlation between the amount of ATZ removal and the population of D2 strain (r=−0.983, P<0.01). Analysis of the degradation intermediates of ATZ revealed that strain D2 transformed atrazine into hydroxy atrazine (HA), atraton, simazine (DMA), deethyl atrazine (DEA), hydroxy simazine (HDMA) and desethyl-desisopropyl atrazine(DACT)through dechlorination hydroxylation, hydrodealkylation, methylation, dealkylation and hydrolysis. Therefore, strain D2 is a strong degrading strain with better environmental adaptability than most reported strains, and could be used widely in the remediation of ATZ wastewater and contaminated soil.
-
Key words:
- atrazine /
- strain /
- degradation characteristics /
- degradation pathway /
- Yangtze River Delta
-
煤矸石是采煤和洗煤过程中排放的固体废物,是在成煤过程中与煤层伴生的一种含碳量较低、比煤坚硬的黑灰色岩石[1],可通过堆积、风化、淋溶等多种作用对周边大气、土壤和水体造成不同程度的污染[2- 3]。长期以来,煤矸石被用于低洼地区的垃圾填埋,形成了大量露天堆积物,造成植被破坏、水土流失等环境污染问题。煤矸石长时间的堆积以及治理措施经验的不足,造成门头沟区尾矿库附近的矸石山存在严重的重金属污染,尤其是汞(Hg)[4]。汞是一种毒性很强的重金属元素,在自然界中呈现多种赋存形态,如金属汞、无机结合态汞和有机结合态汞。无机汞的毒性较弱但进入环境后将经历一系列的转化为甲基汞,甲基汞是毒性最强的汞化合物[5]。为有效解决汞污染,联合国环境规划部于2013年制定了国际汞公约《水俣公约》;我国政府于2011年颁布的《重金属污染综合防治“十二五”规划》(国函〔2011〕13号)中,将汞列为重点管理重金属,并制定了汞污染排放量额指标,随后于2013年正式启动973汞污染特性以及环境和减排技术项目[6]。
国内外学者对煤矸石利用方面的研究[7-23]较多,如建材(包括制作水泥、混凝土,制砖,作路基材料,井下填充,土地复垦等)、高值化利用(包括合成陶瓷,制备氧化铝,提取镓、锂等稀有稀土元素)、吸附剂作用、碳回收等,但在环境污染方面的研究较少。对于重金属总量、赋存形态、淋溶等研究[24-29]主要集中于矿区中煤的研究和周边土壤重金属污染评价;对煤矸石中汞的研究主要讨论不同的加热速率、停留时间、气氛条件(N2、O2、CO2)、氧气体积分数等影响因素对煤矸石中汞的热释放[30-32],并表明煤矸石中汞释放的主要因素为温度。然而,北京门头沟区煤矸石中汞的空间分布、迁移富集规律及潜在环境风险却没有被系统地研究过。本次研究区域位于门头沟区的西南和中东部矿集区,具有百年多的煤炭开采历史[33],该地拥有煤和非金属矿产等多种资源,大规模的开采曾带动了当地区域的发展,同时也改变和破坏了当地的地质环境,产生了一系列的环境问题。在煤炭开采过程中遗留下大量煤矸石裸露堆积在地表已成为门头沟矿区的主要污染源,这些露天堆放的煤矸石经过风化和淋滤等作用,成为矿区水土污染的重要来源,对北京市门头沟矿区周边环境造成了严重影响。因此,研究北京市门头沟煤矸石堆放过程中汞的环境效应有着重要的理论和现实意义。
本文以北京市门头沟典型矿区堆积的煤矸石为研究对象,探讨煤矸石中汞的含量及影响因子,并采用改进的BCR连续提取法提取汞的赋存形态,探究在酸雨条件下初始pH、粒径、固液比等因素对汞的溶出特征的影响,为重金属汞的污染监测、矿区的进一步治理及生态修复提供理论依据。
1. 材料与方法(Materials and methods)
1.1 研究区概况
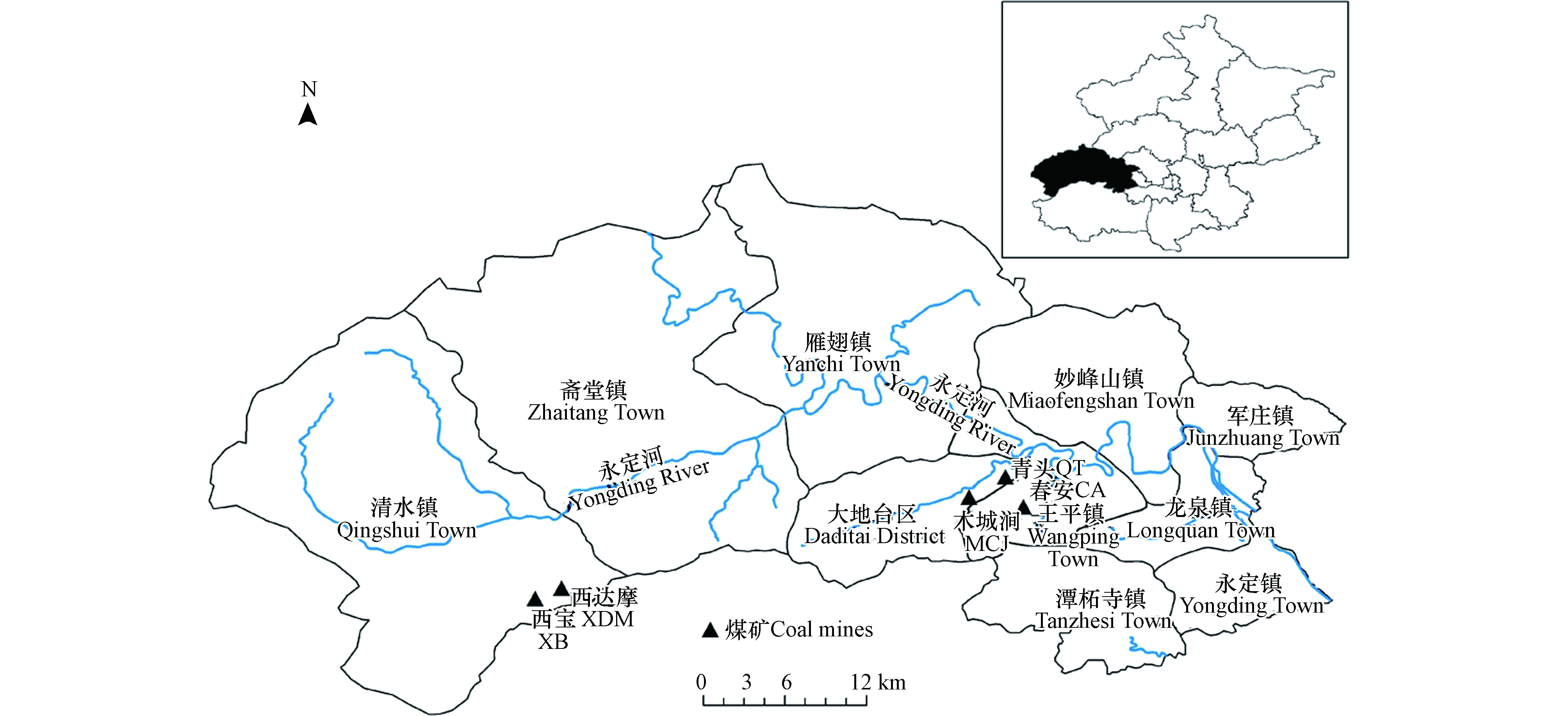
门头沟区蕴藏着丰富的煤炭资源,煤的储藏面积近700 km2, 占全区总面积的一半, 是中国五大无烟煤产地之一, 曾经是首都的能源基地[34]。但随着门头沟“首都西部生态涵养区”定位的确定, 各类矿山在2020年9月已经全部关闭, 在此过程中产生了大量的矿业废弃地。其中, 煤矿废弃地是主要的废弃地类型,煤矿废弃类型包括煤矸石压占、塌陷坑、废弃房屋压占等[35],大部分的煤矸石堆停止排矸后就地露天堆放,其中的重金属经过长年淋溶逐渐释放到周边环境中,造成重金属污染。研究区位于北京城区正西偏南,山地面积占98.5%,是北京唯一的纯山区。该地区气候为温带季风气候,冬季干燥寒冷, 最低温度−15 ℃,7—8月份温度最高, 在35 ℃以上。年均降水量600—700 mm, 多集中7—8月份,无霜期170—200 d左右[36]。本文选择清水镇、大地台区、王平镇3个镇的5个矿区进行研究,分别为西达摩(简称“XDM”)、西宝(简称“XB”)、青头(简称“QT”)、春安(简称“CA”)和木城涧(简称“MCJ”)(见图1)。
1.2 样品采集与前处理
2019年4月沿永定河上游向依次对清水镇、大地台区、王平镇等地的矿区矸石山采集45个自然风化煤矸石固态样品,选用5点采样法采集表层0—20 cm矸石,其中XDM矿区5个(1—5),XB矿区9个(5—14),MCJ矿区4个(15—18),CA矿区9个(19—27),QT矿区18个(28—45)样品,分别保存在聚乙烯塑封袋中,依次编号。去除样品中根系、石块和虫体等杂物,于干净通风处自然风干,切割磨碎后分别过20目、100目和200目尼龙筛,密封于自封袋中保存待测。
1.3 实验方法
风化煤矸石总汞用王水水浴消解-冷原子荧光测定[37]。其主要步骤为:称取0.5 g过100目筛的样品,加入10 mL(1+1)王水,加塞后摇匀,于沸水浴中消解2 h,取出冷却,立即加入10 mL的保存液,用稀释液稀释至刻度,摇匀后放置,取上清液待测,同时做空白实验。每组实验带有平行样品,且每次实验均重复3次,实验结果的不确定性低于5%。pH值用雷磁PHS-3C测量,Eh值测试配夹ORP计复合电极。
煤矸石中汞(Hg)采用AFS-930双道原子荧光光度计(北京吉天)测定;采用扫描电镜(SEM)(SU8100型冷场扫描电镜,日本日立)测定浸出前后矸石样的表面结构;pH的测定参照王萍[38]等的实验方法(固液比为1∶2.5);总有机碳(TOC)含量采用重铬酸钾氧化-分光光度法(HJ615—2011)测定;含水率按照国家《煤和岩石物理力学测定方法》(GB/T 23561)测定。
煤矸石形态采用欧共体标准物质局提出的BCR连续提取法测定[39],其提取步骤为:
(1)弱酸提取态(F1):准确称取0.5000 g 200目矸石样置于50mL离心管中,加入25 mL 0.11 mol·L−1醋酸溶液,设定温度(22±5)℃,放于摇床上220 r·min−1振荡16 h,再3000 r·min−1离心20 min获得上清液,待测,其残渣供下一步提取。
(2)可还原态(F2):将上一步的残渣物加入25 mL 0.5 mol·L−1的盐酸羟胺溶液,用HNO3调至pH为2,(22±5)℃再以220 r·min−1的转速振荡16 h,同上离心洗涤,取上清液供下一步提取。
(3)可氧化态(F3):将F2的残渣物加入5 mL H2O,摇匀,加入10 mL 30% H2O2。离心管加盖在室温下反应1 h,间歇振荡,然后去盖在85 ℃水浴中继续加热1h,直到试管中H2O2体积减少到1—2 mL,再向其中加入10 mL H2O2,在85 ℃水浴中加热1 h,直到H2O2蒸发近干。待冷却后,向其中加入25 mL醋酸铵溶液(1 mol·L−1,pH=2)。同前两步,再次振荡16 h后转移上清液
(4)残渣态(F4):其提取步骤参考土壤汞的消解步骤。
1.4 数据处理与分析
重金属含量描述性统计分析与Person相关性分析由SPSS 24.0实现;样点分布图与Hg的空间分布图由ArcGIS 10.3.1实现;折线图等由Excel 2013和Origin 2017实现。所有实验设置空白对照和平行试验,回收率86%—110%,将BCR提取的4种Hg形态含量之和与煤矸中Hg总量相比,其回收率在80%—115%之间。
1.5 污染评价方法
1.5.1 富集因子法
富集因子(enrichment factor,EF)可定量表示元素富集程度并识别来源,计算方法如下:
EF=(Ci/Cref)sample(Ci/Cref)backgrpund 式中,Ci为重金属i的含量(mg·kg−1);Cref为参比元素的含量(mg·kg−1)。
EF分级标准为:EF≤2,无或轻微富集;2<EF≤5,中度富集;5<EF≤20,显著富集;20<EF≤40,强烈富集;EF>40,极强富集[40]。研究表明,Sc的分布稳定,变异系数小[41],因此将Sc作为参比元素。
1.5.2 地质积累指数法
地质累积指数法(geoindex,Igeo)是由海德堡大学沉积物研究所MÜLLER[42]在1969年提出,不仅考虑了自然地质过程造成的背景值影响,也充分注意了人为活动对重金属污染的影响,是区分人为活动影响的重要参数,计算方法如下:
Igeo=log2CnkBn 式中,Cn表示样品中测量到的重金属含量(mg·kg−1),Bn表示重金属的背景值(mg·kg−1),引入因子k(k=1.5)是为了减小背景值可能变化的影响。Igeo分级标准为:Igeo≤0,无污染;0<Igeo≤1,轻度污染;1<Igeo≤2,中度污染;2<Igeo<3,中度-重度污染;3≤Igeo<4,重度污染;4≤Igeo<5,重度-极度污染;Igeo≥5,极度污染。
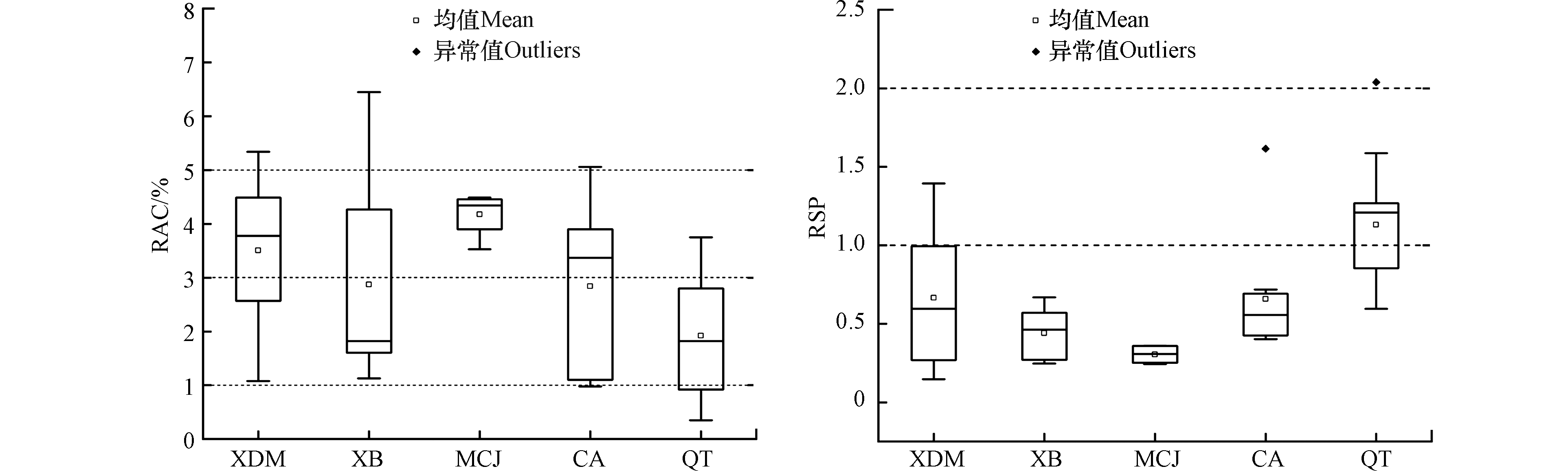
根据重金属赋存形态,将弱酸溶态称为生物可利用态,可还原态和可氧化态称为潜在生物可利用态,残渣态称为生物不可利用态[43]。基于煤矸石中Hg的赋存形态,采用风险编码法(risk assessment coding method,RAC)和次生相和原生相分布比值法(ration of secondary phase and primary phase,RSP)对Hg进行有效性评价。
1.5.3 风险编码法
风险编码法主要通过计算弱酸态含量(F1)占总量的比例来评价环境风险[44],其计算公式如下:
RAC=CF1CT×100% 式中,RAC为F1占总量的质量分数,%;CF1为F1弱酸溶态重金属含量(mg·kg−1);CT为BCR四态含量之和(mg·kg−1)。根据RAC大小,将风险程度分为5个等级:RAC<1%(无风险), 1%≤RAC<10%(低风险),10%≤RAC<30%(中等风险),30%≤RAC<50%(高风险),RAC≥50%(极高风险)。
1.5.4 次生相和原生相比值法
次生相和原生相比值法(RSP)是通过计算原生矿物的风化产物和外来次生产物的比值来评价重金属对煤矸石环境的污染程度[45-47],其计算公式下:
RSP=MsecMprim 式中,RSP为次生相和原生相的比值,Msec为次生相的重金属含量,Mprim为原生相重金属的含量,本文以BCR前三态含量之和为次生相重金属含量(mg·kg−1),原生相重金属含量为残渣态(mg·kg−1),其风险等级为:RSP<1(无污染),1<RSP<2(轻度污染),2<RSP<3(中度污染),RSP>3(重度污染)。
2. 结果与讨论(Results and discussion)
2.1 煤矸石重金属的含量分析
2.1.1 煤矸石样品的理化特性
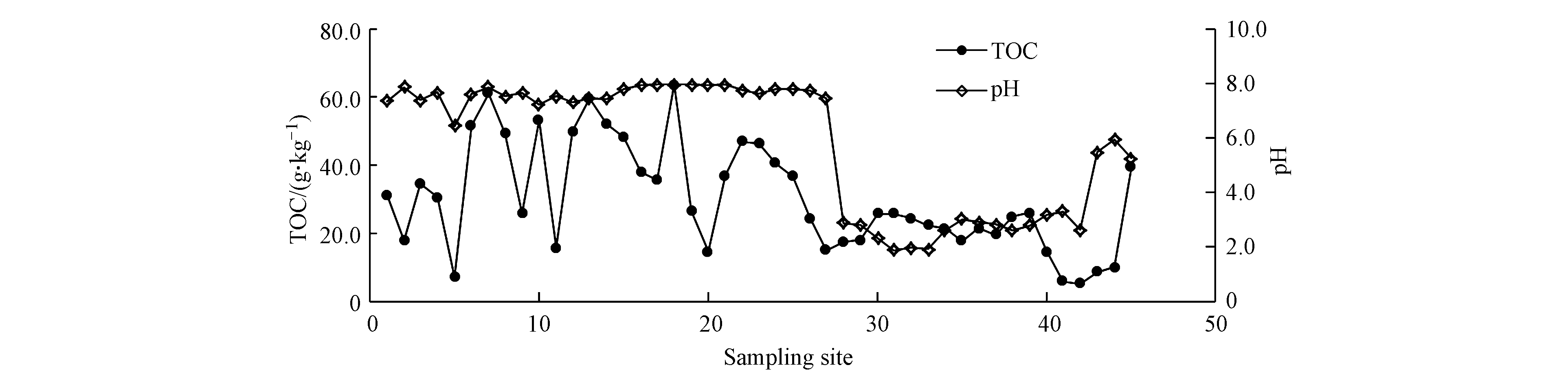
研究区煤矸石的pH和TOC含量如图2所示,除QT煤矿外,其余4个矿区pH值变化范围为6.42—8.05,均值为7.63,属于中性偏碱范围;QT煤矿周边堆积的矸石pH均值为3.13,在1.88—5.94之间,呈现出较强的酸性,这是由于QT煤矿在多年自然恢复作用下暴露在大气中,经风化淋溶作用使得矸石表面酸化,从而呈现出酸性[48]。矸石TOC含量变化范围为5.19—63.72 g·kg−1,均值为30.33 g·kg−1,最大值出现在18号采样点,该点位于矸石山堆积下方河流附近,周边生物残体、凋落物[49]及耕地土壤施肥等因素导致有机质含量较高。
2.1.2 煤矸石中Hg的全量分析
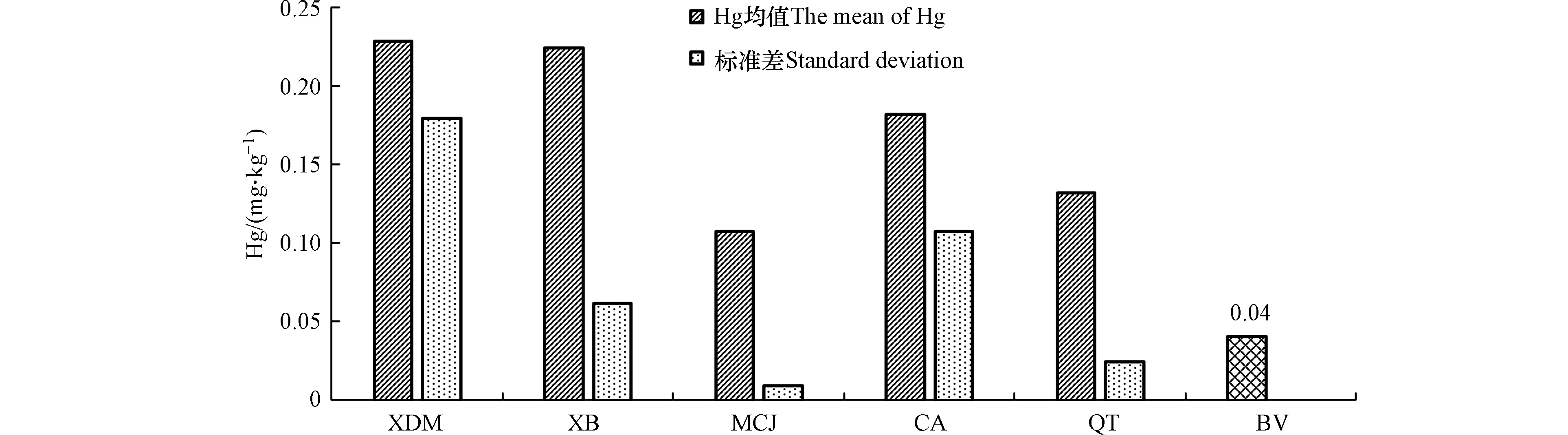
研究区45个样品中Hg含量的描述性统计结果如表1、图3所示。由表1,Hg的含量为0.085—0.519 mg·kg−1,均值为0.169 mg·kg−1,标准差为0.089 mg·kg−1,变异系数为52.09%,属于高变异(50%—100%),空间异质性强,各矿区均值从大到小依次为XDM>XB>CA>QT>MCJ。Hg的平均值含量超过了北京市土壤背景值[50],超标倍数为4.225,表明Hg对该研究区有一定的污染,各矿区单因子超标倍数均值从大到小依次为XB>XDM>CA>QT>MCJ。富集系数和地累积指数的大小能体现出研究区的污染程度[51-52],其中Hg的平均富集系数和地累积指数分别为4.53和1.35,属于中度污染,各矿区Hg的平均EF依次为XB>XDM>QT>CA>MCJ,平均Igeo依次为XDM>CA>QT>XB>MCJ。
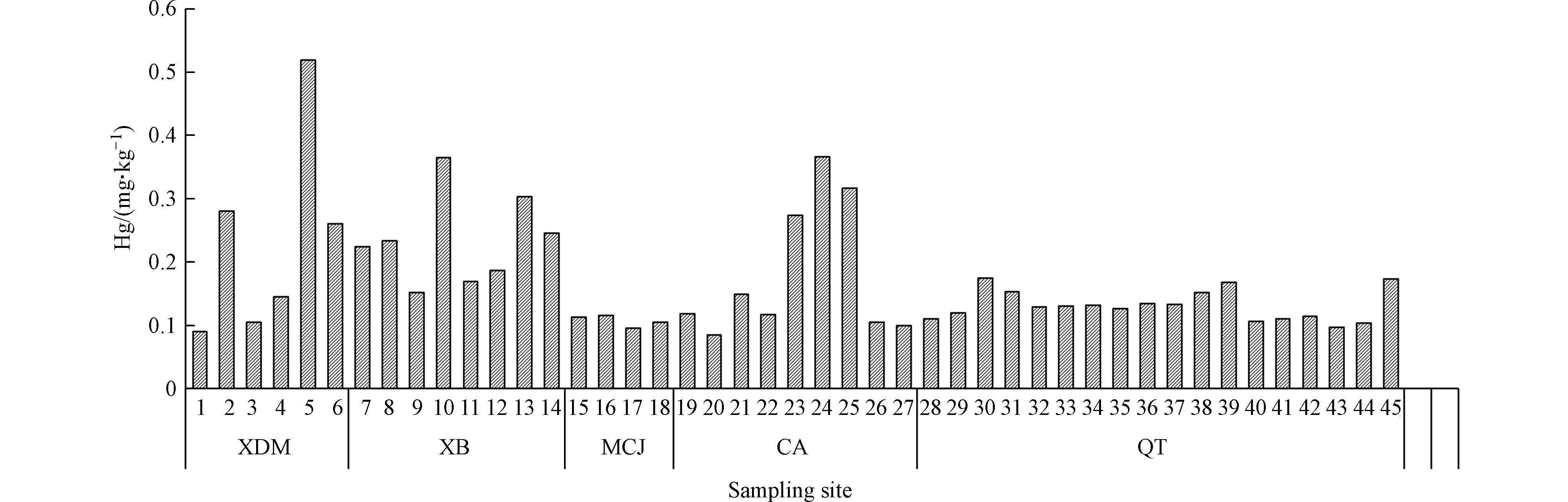
表 1 煤矸石中汞含量描述性统计分析(mg·kg−1)Table 1. Descriptive statistical analysis of Hg content in coal gangue (mg · kg−1)重金属元Heavy metal elements 最小值Minimum 最大值Maximum 平均值Mean 标准差Standard deviation 偏度Skewness 峰度Kurtosis 变异系数Coefficient of variation EF Igeo Hg 0.085 0.519 0.169 0.089 5.17 5.55 52.09% 4.53 1.35 根据各样点汞元素的含量,对汞的分布做柱状图,结果如图4所示,Hg含量分布较为均匀的是MCJ矿区和QT矿区,含量不高;同样可见,5、10、13、24、25号的5个样点含量较高,这几个点主要分布在XDM、XB和CA矿区,呈现局部污染态势,这与Li等[53-54]的研究一致;k-s检验表明,MCJ和CA矿区的均值存在显著性差异(P<0.05)。在空间位置上,研究区Hg含量高的区域主要集中在矿区边缘,并以该区域向周围扩散,这可能是由于大气沉降、矿山开采、冶炼、运输等活动产生的固体废物在矿区边缘堆积,经过雨水的冲刷和风力等物理条件的作用向四周扩散而成。
2.1.3 煤矸石中Hg与各理化指标间的相关性分析
煤矸石的理化性质(总有机碳含量、含水率、pH、Eh)会影响重金属的总量和迁移转化能力,因此,总有机碳、含水率、pH、Eh是重金属含量分析时的重要指标。由研究区Hg的含量与pH、Eh、TOC和矸石含水率(
ωH2O ωH2O ωH2O ωH2O 表 2 煤矸石中重金属含量与各理化指标间的相关系数Table 2. Correlation coefficients between Hg content in coal gangue and various physical and chemical indicatorsHg TOC pH Eh ωH2O Hg 1 TOC 0.251 1 pH 0.256 .531** 1 Eh −.326* −.513** −.972** 1 ωH2O .440** .401** −0.147 0.135 1 **. 在 0.01 级别(双尾),相关性显著;*. 在 0.05 级别(双尾),相关性显著。 **Significance level P < 0.01; * Significance level P < 0.05. 2.2 煤矸石中Hg的赋存形态及影响因素研究
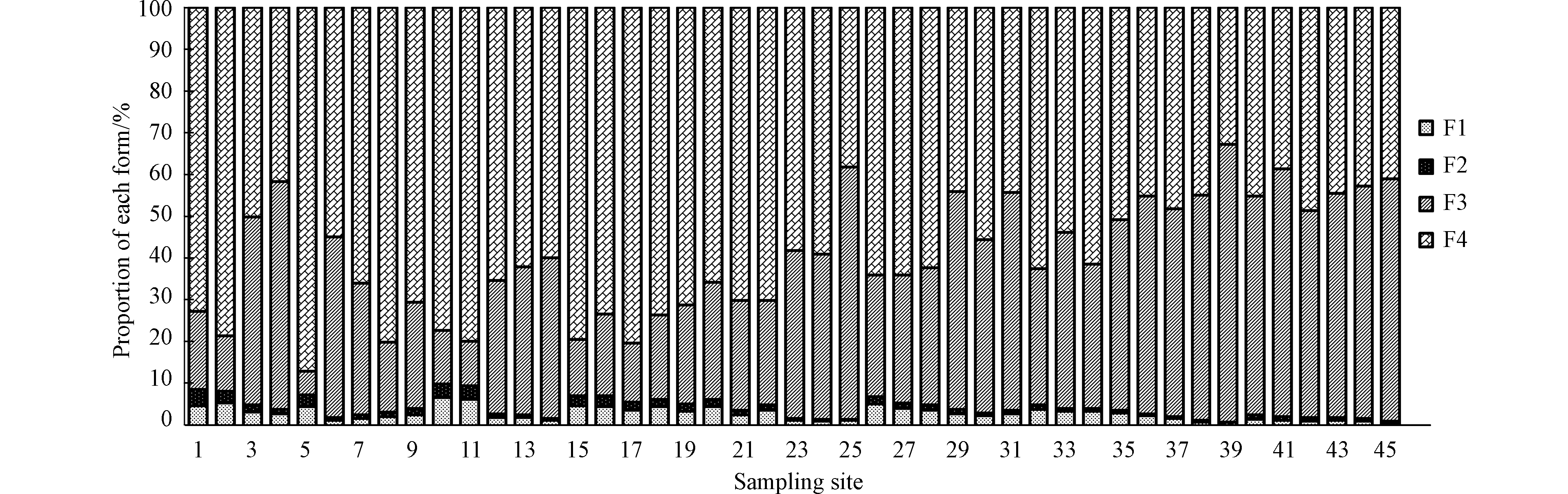
为了解研究区Hg的污染状况,采用改进BCR连续提取法对研究区表层煤矸石Hg的赋存形态进行分析,其提取形态分为4种:弱酸提取态(F1)、可还原态(F2)、可氧化态(F3)和残渣态(F4)。研究区煤矸石中Hg元素的F1占0.35%—6.45%,F2占0.28%—3.92%,F3占5.71%—66.46%,F4占32.91%—87.20%,Hg的主要形态为F3和F4,各形态之和占总量的83.53%—97.71%。由图5不难看出,Hg的赋存形态特征表现为F4(59.66%)>F3(36.37%)>F1(2.68%)>F2(1.29%),对环境的危害较小[26]。由此可见,煤矸石中Hg元素的化学性质较为稳定,可给性极弱,难以被生物吸收利用[56],但F3占比较高,易与矸石中有机物络合,导致Hg2+释放并被植物吸收利用[57],造成周边环境污染。
表3中F2与F3呈显著负相关(P<0.01),当环境处于缺氧状态时,F2被释放,当环境处于氧化条件时,F3被释放;且F2与TOC、pH呈现显著正相关(P<0.01),表明在某种条件下,TOC、pH能影响F2的含量;F3与F4呈显著正相关(P<0.01);F4与全量Hg呈显著正相关(P<0.01),表明全量Hg是影响F4的主要因素,Hg的潜在活性随全量的增加而增加;F4与TOC呈显著正相关,表明F4受TOC影响较大,易与有机质结合,但从有机质中解吸Hg需要克服较大的活化能。
表 3 煤矸石中Hg的不同形态与pH、TOC的相关性关系Table 3. Correlation between different forms of Hg in coal gangue and pH and TOCF1 F2 F3 F4 Hg TOC pH F1 1 F2 0.242 1 F3 −0.225 −.403** 1 F4 0.111 −0.023 .653** 1 Hg 0.015 0.133 0.203 .437** 1 TOC 0.206 .419** 0.055 .475** 0.29 1 pH 0.098 .702** −0.258 0.157 0.266 .531** 1 *. 在 0.05 级别(双尾),相关性显著;**. 在 0.01 级别(双尾),相关性显著。 **Significance level P < 0.01; * Significance level P < 0.05 2.3 煤矸石中Hg污染风险评价
煤矸石中重金属元素迁移能力的强弱与其化学形态密切相关[58]。由2.1.2节得知,该研究区Hg富集严重。故选择Hg元素为研究对象,探究其潜在风险。不同矿区Hg形态的潜在风险结果如图6所示。比较RAC可看出,5个矿区中有39个样品的RAC值在1%—10%,为低风险程度,占总量的86.67%,其中MCJ矿区分布较为均匀,每个样品保持在4.18%左右,相对其他矿区,环境风险相对较高;其中10号样点Hg的RAC值最大,为6.45%;总体而言,各矿区中Hg的平均RAC值由强到弱分别为MCJ>XDM>CA>XB>QT,属于低风险。比较RSP计算结果,不同矿区也存在着差异,基本属于无污染,其中QT矿区Hg的平均RSP值最大,为1.13,属于轻度污染,表明QT矿区的污染程度相对较高,可还原态Hg和可氧化态Hg极大可能会迁移到环境中;总体而言,各矿区Hg的平均RSP值由大到小依次为QT>XDM>CA>XB>MCJ,轻度污染的占比为31.11%,其余均为无污染;整体而言,研究区Hg的潜在风险程度较低,但仍存在风险,需引起重视。
2.4 煤矸石中Hg的浸出特征
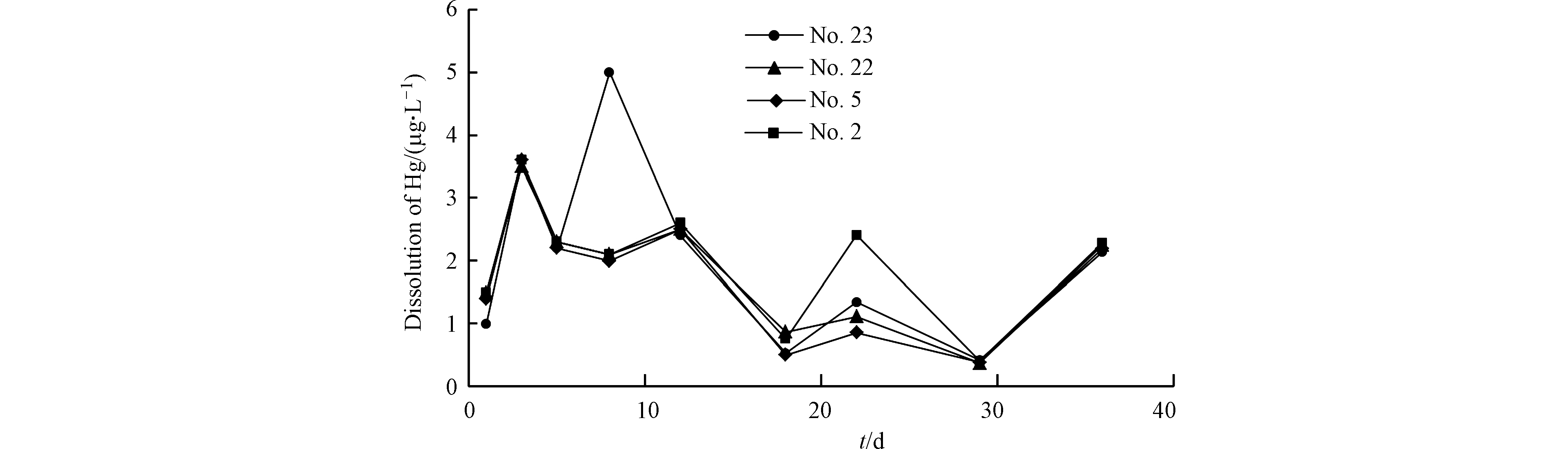
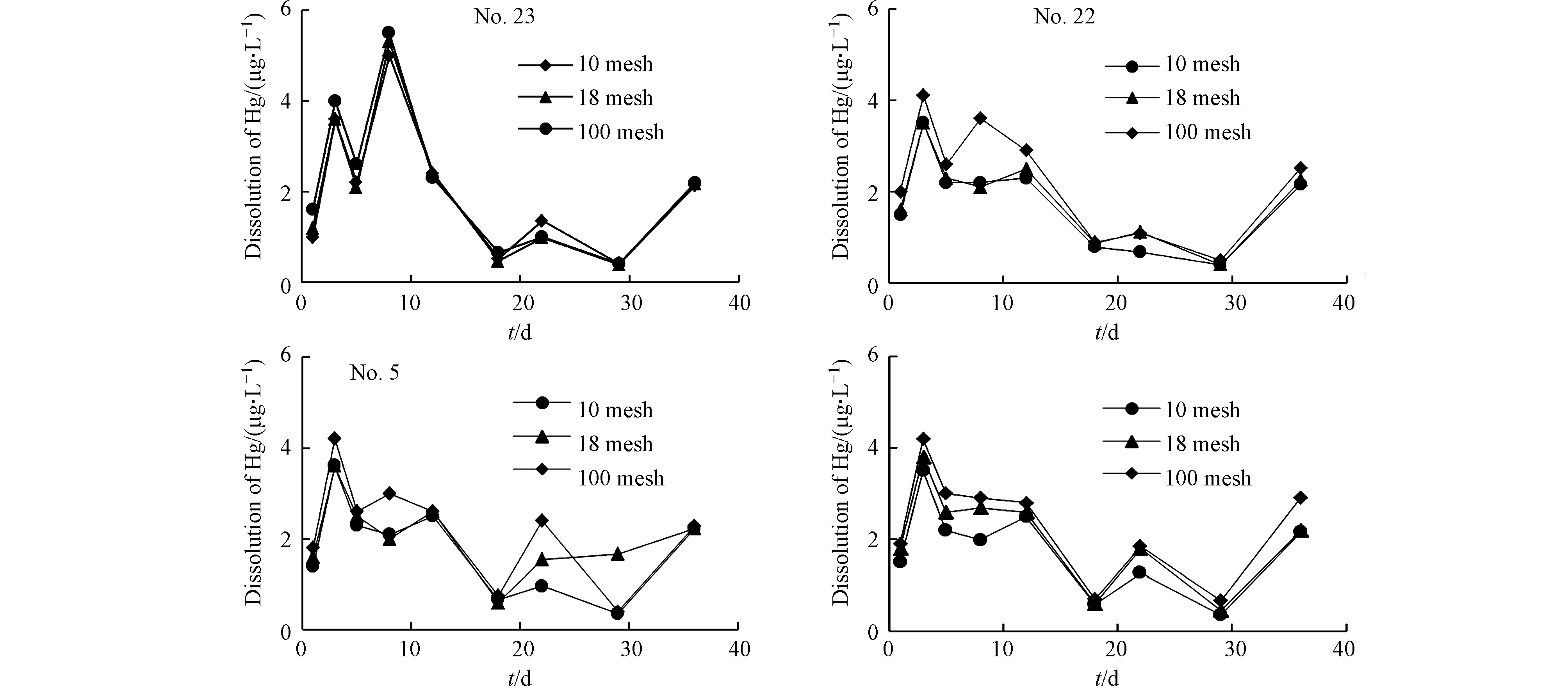
选取XDM和CA两个矿区中总汞含量较高的4个煤矸石样(23号、22号、5号、2号)对其进行淋溶试验。从淋溶时间、初始pH、粒径、固液比的4个方面看其溶出特征并进行原因分析。将初始淋溶液pH设定为5.60和3.11[59-60]置于500 mL锥形瓶中与矸石样混合浸泡,浸泡后对样品进行早中晚3次均匀搅拌,用封口膜将锥形瓶封口,对不同初始pH、粒径和固液比的样品进行同样操作。在第1、3、5、8、12、18、22、29、36 d分别取样5 mL测试Hg的浓度(王水消解法),保持室内温度为20℃左右,每组实验设置3组平行样。
2.4.1 浸泡时间对煤矸石中Hg的溶出影响
门头沟地区多年连续降雨时间最长为18 d,但降雨过程持续时间短且雨量较小,而地势较低处的煤矸石受雨水的浸泡时间较长,故选择浸泡测验时间为1、3、5、8、12、18、22、29、36 d。由图7可看出,4个样品在30 d前整体均有两个明显的峰值,在第3 天首次出现溶出高峰,溶出量在3.6 μg·L−1左右,出现原因主要为弱酸提取态Hg在酸性条件下极易发生溶解释放到上清液中,加之搅拌时间间隔较长造成缺氧环境使得可还原态Hg被溶解释放。在第22 天迎来第二次高峰,且第一次峰值约为第二次的2.54倍,可能出现的原因是该样品中Hg在搅拌过程中伴随着O2的进入被氧化释放所得。
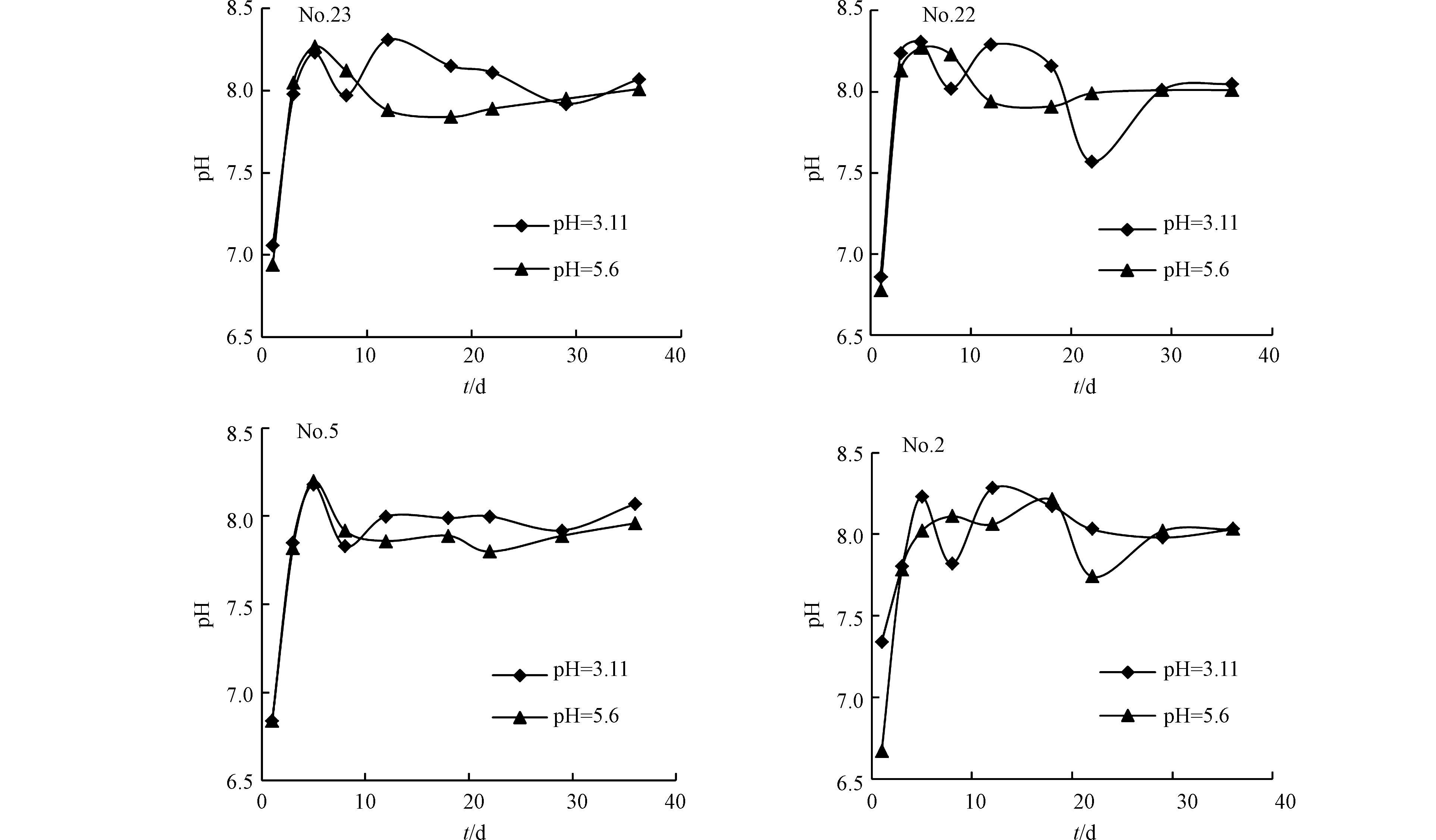
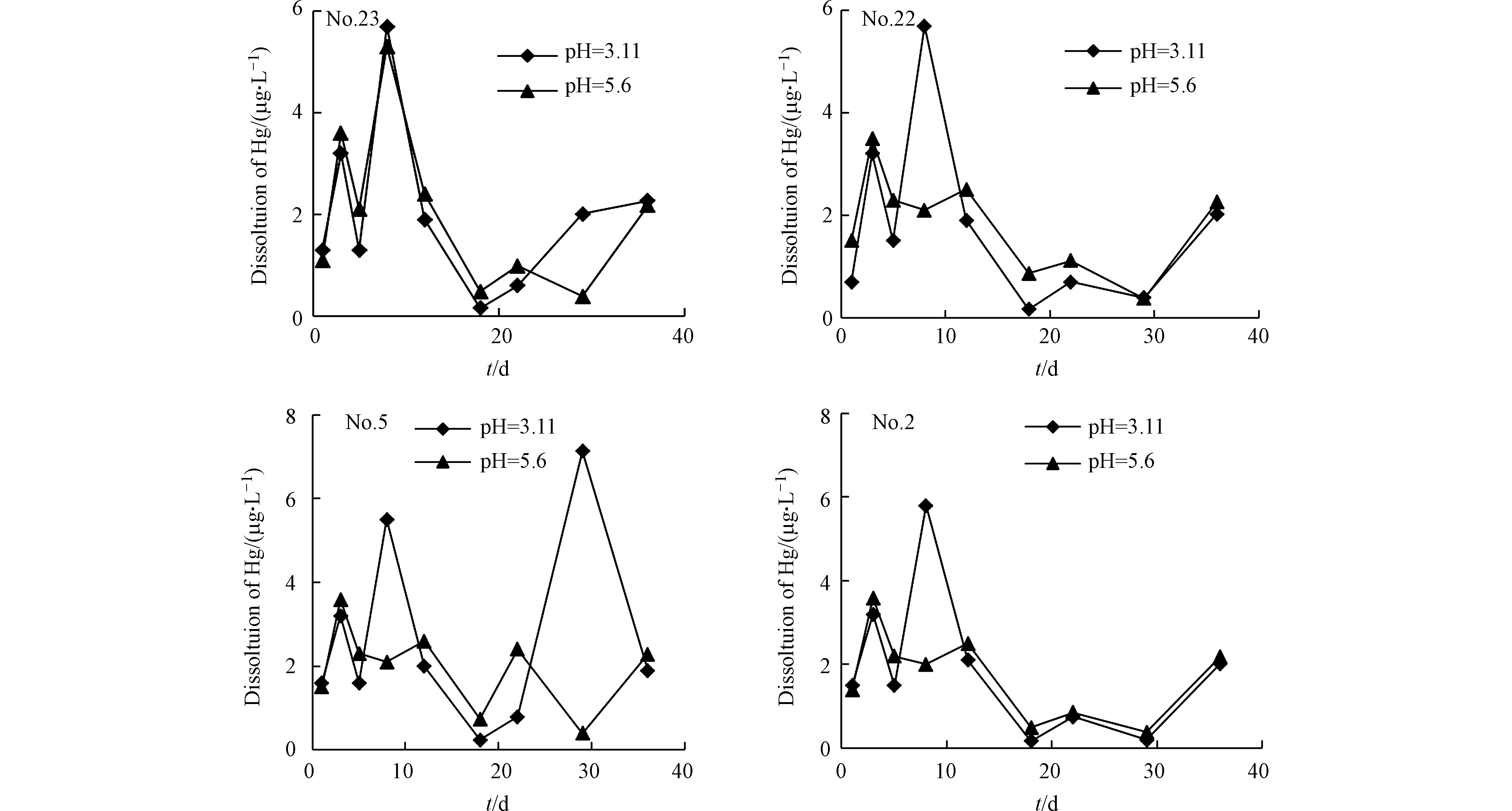
2.4.2 pH对煤矸石中Hg的溶出影响
pH值是评价环境污染程度的重要指标之一,它影响着元素的价态和形态。该地区多年降水的平均pH值和最低pH值分别为5.6和3.11,因此选择初始pH值为3.11和5.6的浸泡液。研究区处于山地,汽车较少,主要影响因子为煤中的硫元素,故初始pH采用硫酸∶硝酸=3∶1的混合液,并用去离子水稀释配制。由图8可知,无论初始pH为多少,均在第5天浸出液的pH值达到第一个峰值,之后便维持在8.0左右,为弱碱性,此时的氧化还原电位也达到稳定状态,说明矿物在该体系中的溶解和沉淀达到一个动态平衡的状态,这与LIN等[62]的研究一致。当初始pH 3.11时,4个样品的起伏较大,当初始pH 5.6时,走势均为先增再减,且样品后期均有pH下降的现象,这是因为煤矸石中本身带有消耗碱性矿物,长期浸泡会导致pH有短暂时期的降低[63]。
由图9可知,第1天,初始pH3.11时Hg的溶出量较初始pH5.6时多,这是由于在酸性条件下,Hg的溶出量大于沉淀量[62-63],溶出量主要为F1,F1与矸石结合最不稳定,最易溶解到渗滤液中,且酸性条件可以极大地增加环境中Hg和有机物的迁移转化;而在第8—12天Hg的溶出量出现了第二个高峰期,这是因为在实验过程中搅拌次数不多,造成锥形瓶中出现缺氧状态,从而使得F2被释放出来,溶解到渗滤液中[62]。因而在酸性条件下,Hg更易析出溶解并释放到环境中,影响周边环境。
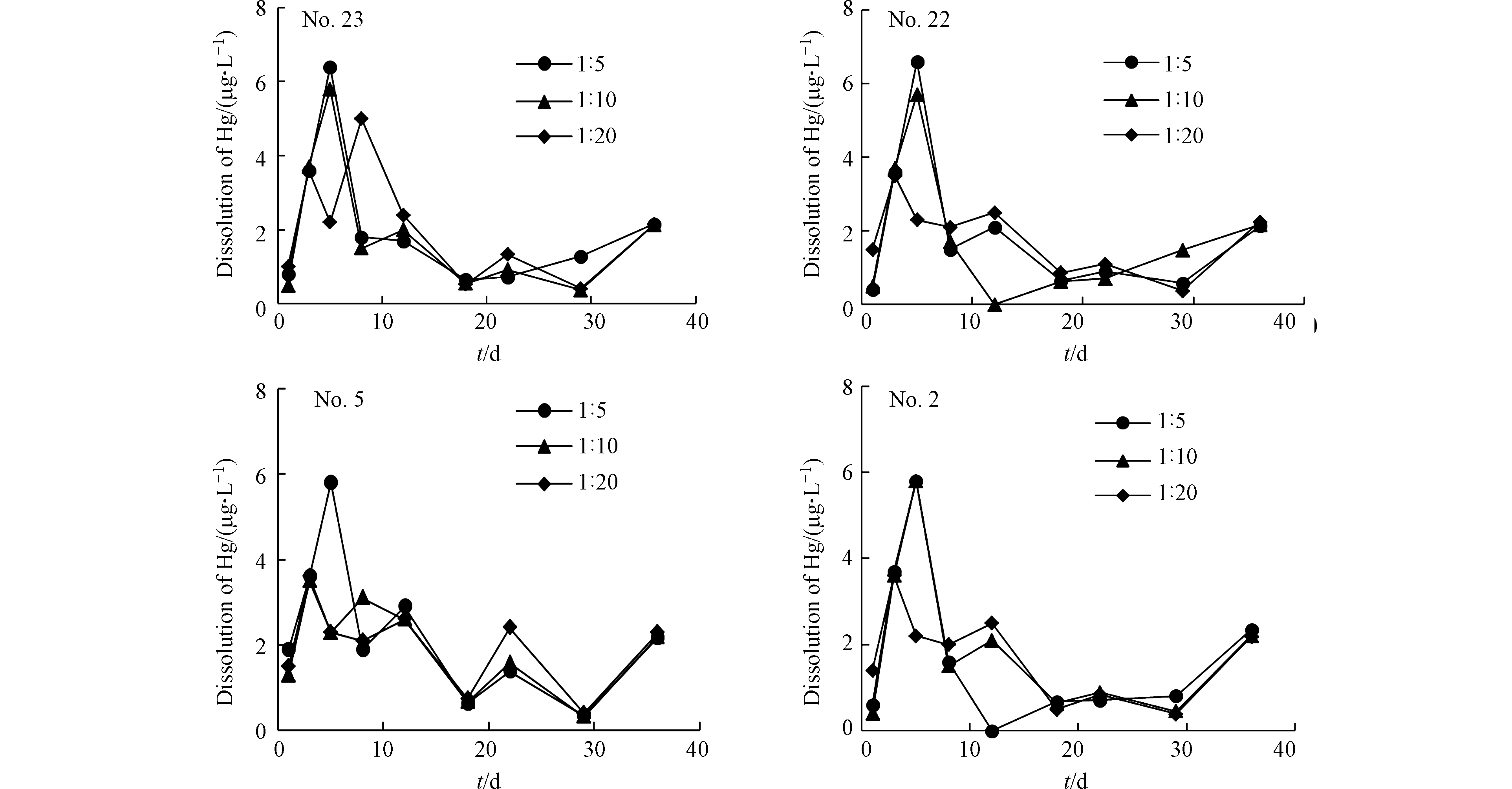
2.4.3 粒径对煤矸石中Hg的溶出影响
煤矸石的粒径对重金属的溶解量有较大影响。因而分别选择80 g 10目、18目和100目(相当于粗粒度、中粒度、细粒度)的矸石样置于500 mL的锥形瓶中,分别加入pH为5.6、固液比为1∶5的淋溶液,将样品与去离子水混合均匀后浸泡,并进行充分搅拌。结果见图10,可以看出不同粒径的煤矸石中Hg的溶出量有所差异,4个样品在第3天和第8天有两次溶出峰值。整体而言,3个粒径中Hg的溶出量随浸泡时间的变化不大,但显而易见的是,100目的细颗粒相对粗颗粒而言,溶出量最大,这是由于细颗粒的比表面积大,与液体接触的表面积大,反应速率加快,因而溶解快[64];后期由于溶解出来的Hg向溶液中扩散,一部分被基质吸附,故而溶出量减小。
2.4.4 固液比对煤矸石中Hg的溶出影响
按照煤矸石质量与浸泡液体积比(固液比)为1∶5、1∶10、1∶20,准确称取100目煤矸石样品80 g、40 g、20 g分别置于3个500 mL的锥形瓶中,加入400 mL去离子水,将样品与去离子水混合均匀后浸泡,并进行充分搅拌。由图11可知,固液比为1∶5时Hg的溶出量最大,说明固液比对煤矸石中Hg的溶解释放有重要影响,固液比越大,浸泡液中Hg的溶出量越高。
2.4.5 SEM分析
为研究酸雨侵蚀和重金属析出对样品表面形貌的影响,对原始样品、浸泡中样品和浸泡后样品进行电镜扫描分析,由图12a可观察到,原始样品粒形整体规则、光滑,且表层附有高亮颗粒;淋溶中期(图12b)受酸溶液浸泡侵蚀使得样品发生化学损伤,表面凹凸不平,并出现微孔;在淋溶后期(12c),煤矸石出现了严重的腐蚀现象,表面大颗粒较少,碎裂状较多,煤矸石内部间隙在淋溶后期酸雨作用下得到进一步的发育和扩展,进而煤矸石内部在淋溶后期得到了严重损伤,可能导致煤矸石物理性质发生变化[65],且表面高亮颗粒较多,可能是淋溶过程中析出的重金属物质[64]。
2.4.6 环境影响分析
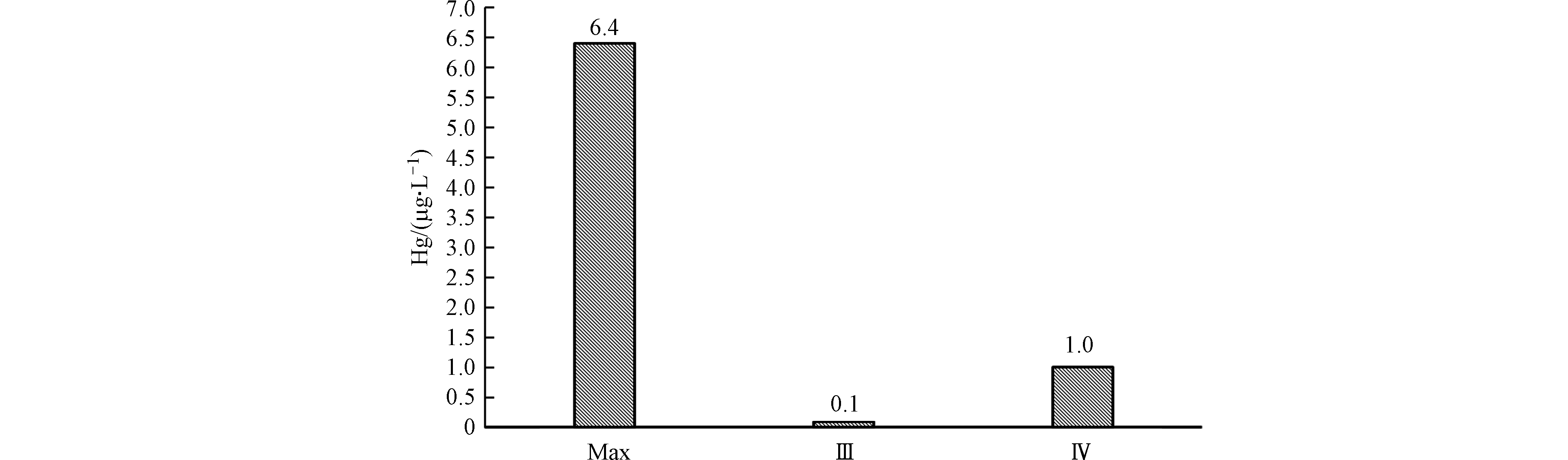
汞元素的溶出最大量为6.4 μg·L−1,为22号样点在固液比为1∶5、粒径为100目、初始pH为3.11时所达到,占该样品总量的1.75%,占比较小。该研究区毗邻永定河,因此对永定河地表水的保护也极其重要,如图13所示,淋溶液中Hg的最大溶出量与GB3838—2002《地表水环境质量标准基本项目标准限制》中Ⅲ类、Ⅳ类水质指标数值相比较得出,Hg元素严重超出国家标准,为Ⅳ类水质的6.4倍,且此次淋溶时间较短,可见煤矸石在短期淋溶下的重金属释放较为明显,加之各地区煤矸石中Hg元素的含量差异较大,Hg 的污染具有长期累积性[66]。因此煤矸石中Hg元素通过地表径流和地下渗流对永定河污染的可能性较大,且不容忽视。
3. 结论(Conclusion)
(1)研究区中Hg的平均含量均超过北京市土壤背景值,其单因子指数、富集因子指数和地累积指数分别为4.225、4.39和1.32,属于中度污染,Hg为强变异水平,XDM矿区变异水平最大,表明XDM矿区受人类活动影响强烈。
(2)该研究区煤矸石中Hg以残渣态Hg为主,对环境危害较小;可氧化态Hg次之,具有二次污染的风险。
(3)风险编码法和次生相与原生相分布比值法均表明各矿区中Hg的污染程度较低,污染风险较小。
(4)静态淋溶试验和SEM扫描电镜分析结果表明,淋溶初期Hg的溶解量较大;酸度越强,初期Hg的溶出量越大;固液比越大,溶出量越高;淋溶时间在5 d达到最大,而后减小;粒径越大,溶出越小。
(5)该研究区中Hg的最大溶出量为Ⅳ类水质的6.4倍,严重超出国家标准,故煤矸石中Hg元素在自然条件下淋溶,通过地表径流和地下渗流对永定河污染的可能性较大。
综合评价结果表明XDM矿区为风险最高矿区,处于中低风险区;MCJ为风险最低矿区,基本属于无污染区;应优先控制对象,并采取相关措施进行改善。综合赋存形态与静态溶出结果,Hg元素中可氧化态与残渣态占比较高,且溶出最大量已超过国家Ⅳ类水质标准,应对矸石山周边已经污染的土壤进行原位或异位修复,并且采取土壤固化技术,防止重金属造成更大面积的地表水与地下水污染。
-
表 1 菌株D2的生理生化特性
Table 1. Physiological characteristic of strain D2
特征 Characteristic 结果 Results 革兰氏染色 + 淀粉水解 − 吲哚 − 甲基红 − V-P产生 − 柠檬酸盐利用 − 硝酸盐还原 + 过氧化氢酶 + 注:“+”为阳性;“−”为阴性. Note:“+” means positive and “−” means negative. 表 2 菌株生长曲线的SGompertz模型和Slogistic模型拟合参数对比
Table 2. Comparative list of bacteria growth due to SGompertz and Slogistic model
名称 Name SGompertz model Slogistic model R2 a1 K1 XC/h μ1/h−1 R2 a2 K2 X0/h μ2/h−1 D2 0.991 1.995 0.279 9.647 0.214 0.992 1.958 0.454 10.950 0.222 表 3 浓度与OD600的相关性分析
Table 3. Correlation analysis between concentration and OD600
平均值 Average value 标准差 Standard deviation 浓度 Concentration OD600 浓度 41.093 44.110 1 OD600 0.080 0.026 −0.983** 1 注:* P<0.05,** P<0.01. 表 4 已报道菌株对阿特拉津的降解效果的比较
Table 4. Comparison of atrazine degradation by strains has been reported
菌种 Strains 浓度/(mg·L−1)Concentation 温度/℃Temperature pH 地区Region 降解率/% Degradation 时间/h Time 文献来源Literature sources Klebsiella sp. FH1 50 25 9 吉林 81.5% 264 [13] LY-2 100 25 — 35 6 — 9 哈尔滨 98.7% 48 [17] CS3 50 30 7 河北 100% 48 [18] Arthrobacter sp. ZXY-2 50 30 — 35 8 — 9 哈尔滨 100% 6 [35] Arthrobacter sp. DNS10 100 30 7.5 哈尔滨 99.41% 24 [36] Paenarthrobacter sp. W11 100 30 7 吉林 97.1% 60 [37] Paenarthrobacter sp. W24 100 30 7 吉林 94.2% 72 [38] Arthrobacter sp. C2 100 30 7 — 9 吉林 100% 72 [33] Pseudomonas sp. 20 30 / 巴西 99% 24 [39] Achromobacter sp. 20 30 / 巴西 39% 48 [39] Pencillium sp. yz11-22N2 8 28 7 / 91.2% 120 [40] D2 100 20 — 30 5 — 9 江苏 100% 18 本研究 -
[1] ALVES de OLIVEIRA L, JARBAS HONORIO de M, GRECCO K L, et al. Atrazine movement in corn cultivated soil using HYDRUS-2D: A comparison between real and simulated data [J]. Journal of Environmental Management, 2019, 248: 109311. doi: 10.1016/j.jenvman.2019.109311 [2] CHOWDHURY I F, DORAN G S, STODART B J, et al. Trifluralin and atrazine sensitivity to selected cereal and legume crops [J]. Agronomy, 2020, 10(4): 587. doi: 10.3390/agronomy10040587 [3] HORZMANN K A, LIN L F, TASLAKJIAN B, et al. Embryonic atrazine exposure and later in life behavioral and brain transcriptomic, epigenetic, and pathological alterations in adult male zebrafish [J]. Cell Biology and Toxicology, 2021, 37(3): 421-439. doi: 10.1007/s10565-020-09548-y [4] 万年升, 顾继东, 段舜山. 阿特拉津生态毒性与生物降解的研究 [J]. 环境科学学报, 2006, 26(4): 552-560. doi: 10.3321/j.issn:0253-2468.2006.04.003 WAN N S, GU J D, DUAN S S. Eco-toxicity and biodegradation of atrazine in the environment [J]. Acta Scientiae Circumstantiae, 2006, 26(4): 552-560(in Chinese). doi: 10.3321/j.issn:0253-2468.2006.04.003
[5] CHEN Q L, YANG B S, WANG H, et al. Soil microbial community toxic response to atrazine and its residues under atrazine and lead contamination [J]. Environmental Science and Pollution Research International, 2015, 22(2): 996-1007. doi: 10.1007/s11356-014-3369-7 [6] 孙涛, 杨再磊, 蒋靖佰伦, 等. 生物炭对土壤中阿特拉津吸附特征的影响 [J]. 环境化学, 2021, 40(3): 687-695. doi: 10.7524/j.issn.0254-6108.2019102207 SUN T, YANG Z L, JIANG J, et al. Effect of biochar on the adsorption characteristics of atrazine in soil [J]. Environmental Chemistry, 2021, 40(3): 687-695(in Chinese). doi: 10.7524/j.issn.0254-6108.2019102207
[7] MAHLALELA L C, CASADO C, MARUGÁN J, et al. Photocatalytic degradation of atrazine in aqueous solution using hyperbranched polyethyleneimine templated morphologies of BiVO4 fused with Bi2O3 [J]. Journal of Environmental Chemical Engineering, 2020, 8(5): 104215. doi: 10.1016/j.jece.2020.104215 [8] MCBEATH S T, GRAHAM N J D. Simultaneous electrochemical oxidation and ferrate generation for the treatment of atrazine: A novel process for water treatment applications [J]. Journal of Hazardous Materials, 2021, 411: 125167. doi: 10.1016/j.jhazmat.2021.125167 [9] JAMES A, SINGH D K. Atrazine detoxification by intracellular crude enzyme extracts derived from epiphytic root bacteria associated with emergent hydrophytes [J]. Journal of Environmental Science and Health, Part B, 2021, 56(6): 577-586. doi: 10.1080/03601234.2021.1922043 [10] JIANG Z, JIANG D, ZHOU Q H, et al. Enhancing the atrazine tolerance of Pennisetum americanum (L. ) K. Schum by inoculating with indole-3-acetic acid producing strain Pseudomonas chlororaphis PAS18 [J]. Ecotoxicology and Environmental Safety, 2020, 202: 110854. doi: 10.1016/j.ecoenv.2020.110854 [11] GÓNGORA-ECHEVERRÍA V R, GARCÍA-ESCALANTE R, ROJAS-HERRERA R, et al. Pesticide bioremediation in liquid media using a microbial consortium and bacteria-pure strains isolated from a biomixture used in agricultural areas [J]. Ecotoxicology and Environmental Safety, 2020, 200: 110734. doi: 10.1016/j.ecoenv.2020.110734 [12] SHARMA A, KALYANI P, TRIVEDI V D, et al. Nitrogen-dependent induction of atrazine degradation pathway in Pseudomonas sp. strain AKN5 [J]. FEMS Microbiology Letters, 2019, 366(1): 277. [13] ZHANG J P, LIANG S, WANG X H, et al. Biodegradation of atrazine by the novel Klebsiella variicola strain FH-1 [J]. BioMed Research International, 2019, 2019: 4756579. [14] YANG X Y, WEI H Y, ZHU C X, et al. Biodegradation of atrazine by the novel Citricoccus sp. strain TT3 [J]. Ecotoxicology and Environmental Safety, 2018, 147: 144-150. doi: 10.1016/j.ecoenv.2017.08.046 [15] 朱希坤, 王雪涵, 姚金城, 等. Arthrobacter sp. AD30和Pseudomonas sp. AD39在阿特拉津工业废水生物处理及污染土壤生物修复中的应用 [J]. 环境化学, 2010, 29(4): 609-613. ZHU X K, WANG X H, YAO J C, et al. Biotreatment of atrazine-containing industrial wastewater and bioremediation of atrazine-contaminated soil by Arthrobacter sp. strain AD30 and Pseudomonas sp. strain AD39 [J]. Environmental Chemistry, 2010, 29(4): 609-613(in Chinese).
[16] de OLIVEIRA LOPES R, PEREIRA P M, PEREIRA A R B, et al. Atrazine, desethylatrazine (DEA) and desisopropylatrazine (DIA) degradation by Pleurotus ostreatus INCQS 40310 [J]. Biocatalysis and Biotransformation, 2020, 38(6): 415-430. doi: 10.1080/10242422.2020.1754805 [17] 李阳阳, 张金波, 沙君雪, 等. 阿特拉津降解菌LY-2的分离鉴定及其对污染土壤的修复 [J]. 农业生物技术学报, 2018, 26(6): 987-994. LI Y Y, ZHANG J B, SHA J X, et al. Isolation and identification of atrazine-degrading bacterial strain LY-2 and its bioremediation to contaminated soil [J]. Journal of Agricultural Biotechnology, 2018, 26(6): 987-994(in Chinese).
[18] 杨晓燕, 李艳苓, 魏环宇, 等. 阿特拉津降解菌CS3的分离鉴定及其降解特性的研究 [J]. 农业环境科学学报, 2018, 37(6): 1149-1158. doi: 10.11654/jaes.2017-1453 YANG X Y, LI Y L, WEI H Y, et al. Isolation, identification, and characterization of atrazine-degrading bacterial strain CS3 [J]. Journal of Agro-Environment Science, 2018, 37(6): 1149-1158(in Chinese). doi: 10.11654/jaes.2017-1453
[19] 马放, 任南琪, 杨基先. 污染控制微生物学实验[M]. 哈尔滨: 哈尔滨工业大学出版社, 2002: 1 − 280 . MA F, REN N Q, YANG J X. Contamination control microbiology experiment [M]. Harbin: Harbin Institute of Technology Press, 2002: 1 − 280(in Chinese) .
[20] 东秀珠, 蔡妙英. 常见细菌系统鉴定手册[M]. 北京: 科学出版社, 2001: 1 − 419 . DONG X Z, CAI M Y. Handbook of systematic identification of common bacteria [M]. Beijing: Science Press, 2001: 1 − 419(in Chinese).
[21] BUCHANAN R L, WHITING R C, DAMERT W C. When is simple good enough: A comparison of the Gompertz, Baranyi, and three-phase linear models for fitting bacterial growth curves [J]. Food Microbiology, 1997, 14(4): 313-326. doi: 10.1006/fmic.1997.0125 [22] 孙鹏, 孙源. 3种方法对中肋骨条藻Logistic生长模型拟合的比较研究 [J]. 江苏农业科学, 2019, 47(2): 229-232. SUN P, SUN Y. Comparative study on three methods for Logistic growth model fitting of Skeletonema costatum [J]. Jiangsu Agricultural Sciences, 2019, 47(2): 229-232(in Chinese).
[23] PASALARI H, ESRAFILI A, REZAEE A, et al. Electrochemical oxidation pretreatment for enhanced methane potential from landfill leachate in anaerobic co-digestion process: Performance, Gompertz model, and energy assessment [J]. Chemical Engineering Journal, 2021, 422: 130046. doi: 10.1016/j.cej.2021.130046 [24] 谢柄柯, 张玉, 王晓伟, 等. 菌株Desulfovibrio sp. CMX的DNRA性能和影响因素 [J]. 环境科学, 2016, 37(10): 3955-3962. XIE B K, ZHANG Y, WANG X W, et al. Performance and influencing factors of dissimilatory nitrate reduction to ammonium process by the strain Desulfovibrio sp. CMX [J]. Environmental Science, 2016, 37(10): 3955-3962(in Chinese).
[25] 陈世宇. 不同土壤中阿特拉津降解特征、降解基因分布及细菌群落演替规律[D]. 杭州: 浙江大学, 2021: 1 − 76 . CHEN S Y. Atrazine degradation characteristic, degradation genes distribution and bacterial community succession in different soils[D]. Hangzhou: Zhejiang University, 2021: 1 − 76(in Chinese) .
[26] 程吟文, 谷成刚, 王静婷, 等. 多溴联苯醚微生物降解过程与机理的研究进展 [J]. 环境化学, 2015, 34(4): 637-648. doi: 10.7524/j.issn.0254-6108.2015.04.2014031407 CHENG Y W, GU C G, WANG J T, et al. Recent advances in mechanism and processes of microbial degradation of polybrominated diphenyl ethers [J]. Environmental Chemistry, 2015, 34(4): 637-648(in Chinese). doi: 10.7524/j.issn.0254-6108.2015.04.2014031407
[27] 刘丹丹, 梁彦秋, 刘长风, 等. 莠去津高效降解菌的鉴定和降解特性分析 [J]. 农药, 2016, 55(5): 340-342,355. doi: 10.16820/j.cnki.1006-0413.2016.05.009 LIU D D, LIANG Y Q, LIU C F, et al. Identification and characterization of an atrazine degrading bacterium [J]. Agrochemicals, 2016, 55(5): 340-342,355(in Chinese). doi: 10.16820/j.cnki.1006-0413.2016.05.009
[28] 李绍峰, 朱静, 李铁晶. 阿特拉津降解菌株的分离、鉴定及降解特性研究 [J]. 环境科学, 2012, 33(9): 3214-3219. LI S F, ZHU J, LI T J. Isolation, identification and characterization of an atrazine degrading bacterium [J]. Environmental Science, 2012, 33(9): 3214-3219(in Chinese).
[29] 赵婉琳, 叶静, 张娜, 等. 褐藻胶降解菌的筛选、鉴定及产酶条件优化 [J]. 微生物学报, 2019, 59(1): 169-180. ZHAO W L, YE J, ZHANG N, et al. Screening, identification and fermentation optimization of an alginate-degrading strain [J]. Acta Microbiologica Sinica, 2019, 59(1): 169-180(in Chinese).
[30] LIU J T, ZHOU J H, GUO Q N, et al. Physiochemical assessment of environmental behaviors of herbicide atrazine in soils associated with its degradation and bioavailability to weeds [J]. Chemosphere, 2021, 262: 127830. doi: 10.1016/j.chemosphere.2020.127830 [31] ZHANG Y, JIANG Z, CAO B, et al. Metabolic ability and gene characteristics of Arthrobacter sp. strain DNS10, the sole atrazine-degrading strain in a consortium isolated from black soil [J]. International Biodeterioration & Biodegradation, 2011, 65(8): 1140-1144. [32] 郭火生, 王志刚, 孟冬芳, 等. 阿特拉津降解菌株DNS32的降解特性及分类鉴定与降解途径研究 [J]. 微生物学通报, 2012, 39(9): 1234-1241. GUO H S, WANG Z G, MENG D F, et al. Degradation characteristics and identification and the degradation pathway of the atrazine-degrading strain DNS32 [J]. Microbiology China, 2012, 39(9): 1234-1241(in Chinese).
[33] CAO D T, HE S H, LI X, et al. Characterization, genome functional analysis, and detoxification of atrazine by Arthrobacter sp. C2 [J]. Chemosphere, 2021, 264: 128514. doi: 10.1016/j.chemosphere.2020.128514 [34] 陆长鸣, 李想, 徐明恺, 等. 一株高效广谱莠去津降解菌SB5的生长和降解特性[J]. 应用生态学报, 2022, 33 (1): 229-238 . LU C M, LI X, XU M K, et al. Growth and degradation characteristics of an efficient and broad-spectrum atrazine-degrading strainSB5[J]. Chinese Journal of Applied Ecology, 2022, 33 (1): 229-238(in Chinese) .
[35] ZHAO X Y, WANG L, MA F, et al. Characterisation of an efficient atrazine-degrading bacterium, Arthrobacter sp. ZXY-2: An attempt to lay the foundation for potential bioaugmentation applications [J]. Biotechnology for Biofuels, 2018, 11: 113-123. doi: 10.1186/s13068-018-1113-0 [36] 张庆媛, 葛世杰, 姜昭, 等. 高效阿特拉津降解菌株DNS10降解条件优化 [J]. 环境工程学报, 2013, 7(3): 1169-1174. ZHANG Q Y, GE S J, JIANG Z, et al. Optimization for degradation conditions of atrazine-degrading strain DNS10 [J]. Chinese Journal of Environmental Engineering, 2013, 7(3): 1169-1174(in Chinese).
[37] CHEN S M, LI Y Y, FAN Z W, et al. Soil bacterial community dynamics following bioaugmentation with Paenarthrobacter sp. W11 in atrazine-contaminated soil [J]. Chemosphere, 2021, 282: 130976. doi: 10.1016/j.chemosphere.2021.130976 [38] 范作伟, 李阳阳, 陈帅民, 等. 阿特拉津降解菌W24的分离鉴定和土壤修复效果研究 [J]. 玉米科学, 2021, 29(5): 172-177. FAN Z W, LI Y Y, CHEN S M, et al. Isolation and identification of atrazine-degrading bacterial strain W24 and its remediation effect on soil [J]. Journal of Maize Sciences, 2021, 29(5): 172-177(in Chinese).
[39] TONELLI FERNANDES A F, BRAZ V S, BAUERMEISTER A, et al. Degradation of atrazine by Pseudomonas sp. and Achromobacter sp. isolated from Brazilian agricultural soil [J]. International Biodeterioration & Biodegradation, 2018, 130: 17-22. [40] YU J P, HE H J, YANG W L, et al. Magnetic bionanoparticles of Penicillium sp. yz11-22N2 doped with Fe3O4 and encapsulated within PVA-SA gel beads for atrazine removal [J]. Bioresource Technology, 2018, 260: 196-203. doi: 10.1016/j.biortech.2018.03.103 -




 下载:
下载: