-
化学工业的发展在给人们生活带来便利的同时,也造成了环境介质中大量的化学品残留[1]。据统计,当前化学文摘(Chemical Abstract Service, CAS)数据库收录的化学品已达到1.25亿种[2]。暴露在环境中的化学品不仅给生态系统造成威胁,也会通过各种途径进入人体进而损害人体健康。如Stehle 和 Schulz [3]指出,杀虫剂的广泛使用造成大型无脊椎动物数量削减30%。通过食物链富集,暴露在环境中的双氯芬酸已造成印第安秃鹰数量的削减[4]。因此,控制环境介质中化学品的含量对降低其对生态系统乃至人类的危害至关重要。
识别环境介质中的化学品是对其进行削减的前提,液相色谱串联高分辨质谱(liquid chromatography coupled to mass spectrometry, LC/MS))是有机化学品检测与识别的最常用手段[2, 5]。由于环境介质较为复杂,环境样品的质谱数据极为复杂,对其进行有效的解析需要一定的策略[6]。非靶向筛查在当前环境样品的液相色谱质谱检测数据中广泛使用[7-8]。非靶向筛查是指在没有标准品及样品先验信息的前提下,仅根据质谱数据识别样品中未知化合物信息的流程及方法[9]。非靶向筛查的流程包括峰提取、去冗余、特征峰筛选、结构注释与鉴定等步骤[10]。化学信息学在每个步骤的数据处理过程中扮演重要的角色。如不同的数据处理步骤会涉及不同的算法[11]。不同的数据处理软件在同一步骤的数据处理过程中使用不同的算法[12]。对于同一步骤,不同的算法在处理数据时会产生一定的差异[13]。因此,对非靶向筛查过程中化学信息学的阐述有利于科研工作者更好的使用不同的非靶向筛查数据处理软件,从而产生更可信数据分析结果。
基于此,针对基于液相色谱串联质谱数据的非靶向筛查流程,本文综述了如图1的分析流程,以及该流程中每个数据处理步骤所涉及的化学信息学知识,对不同非靶向数据处理软件在该过程中使用的算法及该数据处理过程中算法的发展及优劣进行了综述和比较。在此基础上,对非靶向数据处理过程中所使用的化学信息学知识的未来发展方向进行了展望。
-
在LC/MS原始数据中确定二维信号的边界、中心和强度称为峰提取[14]。峰提取是LC/MS数据处理的第一步,对后续的数据处理质量好坏有至关重要的影响。峰提取算法经历了从局部最大法、递归阈值法[15]、分箱法[11]、centWave算法[14]到ADAP算法[13]的发展,这些峰提取算法已在不同时期的非靶向数据处理软件中使用。
局部最大法和递归阈值法类似,均是对用户定义的m/z分箱范围内光谱数据进行离子色谱提取(Extracted ion chromatogram,XIC)。其主要步骤在于(1)在光谱数据维度提取色谱峰;(2)基于谱峰的强度值过滤掉强度较小的谱峰;(3)将不同分箱内的谱峰连接起来。两种峰提取算法的不同之处在于,局部最大法将光谱强度最大值视为最大的谱峰,而递归阈值法将与噪音的尖峰宽度差别最大的峰宽定义为最大值进行数据处理。最后,对提取的峰在色谱维度和质谱(m/z)维度对强度较小的噪音进行过滤,进而产生峰形较好的色谱峰[15]。局部最大法和递归阈值法最初被用于mzMine2软件中。这两种算法也被纳入第二代mzMine 2软件的峰提取过程中[16]局部最大法和递归阈值法计算速度较快,对大数据集可以很快的计算出峰提取结果。但是,环境样品基质复杂,目标化合物的色谱峰可能由于基质的干扰,并不能达到优美的峰形,这使得局部最大法的峰脊线的确定比较困难。同样,递归阈值法中其峰确定化合物峰和假阳性峰的阈值也比较难。centWave算法较好的解决了上述两个问题,对环境样品由于基质复杂导致的峰形较差过程中,假阳性峰的鉴别效果有所提高。
分箱法是指根据高分辨质谱的分辨率,在质谱维度根据合适的质量单位(如0.1 m/z)进行分箱,在每个分箱内,取信号最强m/z的强度作为该分箱内的强度值,然后将不同时间点该分箱的强度值相连即为提取的色谱峰[11]。随后,对提取的色谱峰进行模拟高斯峰匹配过滤,从而改善峰形,进一步的,利用信噪比过滤的方式将噪音峰过滤以减少假阳性结果。通过大量的研究结果表明,信噪比10是比较合适的过滤值。分箱法在最初基于R语言程序操作版本的XCMS中被使用。利用该算法,Smith等[11]对476个血浆样品进行了峰提取,每个样品中提取的峰平均数目为3899个。在后续发展的基于网络平台的XCMS中,分箱法的峰提取算法也被嵌入该非靶向数据处理流程中[17]。虽然分箱法在对质谱数据处理时速度较快,然而合适的分箱值可能难以获得。如果分箱值过小,色谱峰将在不同的箱之间存在,这会导致在每个箱中检出的色谱峰部分分型损失。如果分箱值过大,每个箱内可能存在不同的色谱峰叠加,这会使得强度较小的色谱峰被噪音掩盖[14, 18-19]。
centWave算法的原理在于,具有化合物检出的质谱数据区域其强度更高(较噪音区域的强度信号),可以先找出该轻度更高的区域进行峰检出。在此基础上,采用连续小波变化进行峰匹配和过滤。这种过滤方式的好处在于可以产生预样品质谱数据峰宽等参数相匹配的模式峰,得到可信度更高的峰提取结果[14]。当前centWave算法在各类非靶向筛查数据处理软件中广泛使用。
基于LC/MS的峰提取ADAP算法是在基于气相色谱质谱ADAP算法基础上发展起来的,主要用于解决centWave峰提取假阳性结果较多的问题[13, 20]。ADAP算法首先将质谱第一次扫描得到的m/z按强度大小依次排列,随后对第二次、第三次扫描中的m/z值也依次排列。进而对不同扫描次序中出现的同一m/z在时间维度上将其相连,形成色谱峰。ADAP峰提取算法打破了如分箱算法及centWave算法需要认为设定封箱大小或m/z大小的做法,从而避免了认为设定该参数可能造成的数据处理混乱[13, 21-22]。Myers等 [13]指出,利用ADAP峰提取算法得到的色谱峰与基于XCMS和mzMine 2得到的色谱峰重合度为83%,然而XCMS和mzMine 的2两种软件的峰提取算法提取的色谱峰重合度仅仅为68%,这表明ADAP可以在一定程度上减少假阳性峰的检出。
在峰提取的过程中,平衡假阳性和假阴性峰的提取结果至关重要。为了更好的覆盖所有可能的色谱峰,相应的峰提取参数门槛应该被尽可能设置的更小[23],然而这可能会导致更多的噪音峰被检出,提高假阳性的峰提取结果。相反,又会增加假阴性峰的数目。当前已有不同的尝试去解决这个问题。如Ju等 [24]利用XCMS, MZmine 2 和 SIEVE软件同时对样品数据进行峰提取,通过叠加不同数据处理软件的峰提取结果,从而较大提高的提取的色谱峰数目(1619个、1103个 XCMS、1500个MZmine2、387个SIEVE)。Hu等[23]通过将XCMS, MZmine2及MSDIAL等软件提取的色谱峰数据分为三类:(1)好的一级峰,好的二级谱图;(2)差的一级峰,好的二级谱图;(3)差的一级峰,好的二级谱图;(4)差的一级峰,差的二级谱图或无二级谱图。进而将第三类峰也纳入后续的化合物注释及结构鉴定,从而提高了化合物注释鉴定的结果。
-
一般来说,化合物在液相色谱串联质谱检测过程中可能发生源内裂解、二聚体及多聚体的出现,以及同位素峰及加和物的出现,并非提取的每个色谱峰对应一个化合物信息[25-26]。因此,对峰提取过程中的所有色谱峰进一步去冗余可以减少后续数据分析的复杂程度,增加数据分析的可信度[27]。当前已有不同的算法或软件用于去除质谱中的冗余数据。Zeng等[28]提出离子融合的概念,用以将同位素峰、加和物、中性丢失峰聚到一起,从而达到理想的一个质谱特征峰指代一个化合物信息。离子融合的原理在于同位素、加和物及中性丢失离子与前体离子间具有一定m/z差异的关联。如同位素峰一般是由于C12或者C13造成的m/z为1.0034 Da的差异,加和物峰一般出现的是常见类型的加和物所造成的m/z差异。根据这种关系构建算法,将血浆样品的正离子模式下,峰从609聚合到106个,负离子模式下的峰从1084聚合到169个,大大减少了冗余结果。DeFelice等[29]利用峰形相似性和这些离子对间的质量偏差进行皮尔逊和峰高相似性计算从而聚类同位素峰、加和物峰,并去除仪器等空白噪音。在此基础上开发了MS-FLO软件,可以减少7.8%的冗余信息。Senan等[30]利用同位素峰及加和峰之间峰形及m/z差值之间的相似性,构建相似性网络,从而聚类同位素及加和物峰信息,并开发了CliqueMS软件,从而减少冗余峰信息。根据同位素峰、源内裂解峰和加和物(表1)峰形间的类似性(峰顶时间、左右半峰宽处的时间、以上3个时间之和),通过30种标准品判断相似性大小从而聚类环境样品中冗余峰的信息。利用这种方法,对尼罗河水样进行检测,从而实现对每个样品中平均46%冗余峰信息的扣除[31]。利用这些同位素峰、源内裂解化合物及加和物间峰形的类似性,其他一些软件或算法也已经大量被开发或使用[32-36]。
-
扣除冗余的色谱峰信息后,一般来说仍有大量的色谱峰信息。这些信息可能并不全是我们需要关注的色谱峰信息,因此合适的特征峰筛选手段十分必要[36-37]。
根据统计学知识进行检验是常用的特征峰筛选手段之一。这种差异可以归类为时间前后的差异及空间上的差异。利用时间上前后的差异,Gornik等[38]通过比较生物转化前后样品质谱检测数据提取到的峰强度差异(变化达到10倍以上,P值小于0.05)视为可能的转化产物峰,从而鉴定出10种舍曲林的生物转化产物。采用类似的方法,Weizel等 [39]识别出污水生物处理过程中糖皮质激素的41种转化产物。Purschke 等 [40]采用PCA及分组PCA分析了污水处理过程污染物变化趋势,并筛选出特征污染物氮-甲基吡咯烷酮。利用空间上点的差异,Hohrenk等[41]采用主成分分析(PCA)及多元曲线分辨交替最小二乘法(MCR-ALS)对污水厂进水、出水及下游河流中的特征污染物进筛选,发现经过臭氧处理后,24种污染物被削减至检测限以下。
根据同系物之间的质量亏损值是发现同系物特征峰的有效手段。根据全氟化合物间同系物结构-CF2结构的共性差异,利用质量亏损开发的算法识别出了一系列新的全氟化合物[42-44]。根据这一特征,Koelmel等[45]开发了全流程自动化识别全氟化合物的软件,提高了全氟化合物的识别通量,减少了其识别的工作量和时间。
根据分子结构类似性的分子网络发现类似化合物是一种常用手段。Fu等[46]通过抗生素类化合物的特征碎片对食品中的抗生素类化合物及其产物进行筛选,从而发现6种抗生素类化合物。Zhan等 [47]通过已知神经毒气类化合物特征碎片离子筛选实际样品中的神经毒气类化合物,从而发现一系列新的神经毒气类化合物。Esposito等[48]根据结构类似的化合物具有类似的二级谱图,进而根据二级谱图类似原则构建分子网络,发现了新的藻毒素类化合物[49]。根据类似的原理,Le Dare等[50]将分子网络用于转化产物的鉴定,从而筛选出两种奎硫平在人体内的代谢产物。
根据毒性值来识别是基于毒性效应特征峰的筛选手段[51]。Pochiraju等[52]利用污水水样的极性程度差别进行分馏,针对分馏出各部分水样的不同毒性效应进行关键致毒化合物的识别,最终识别出污水中具有雌激素效应的化合物(邻苯二甲酸二异丁酯、邻苯二甲酸二乙酯和苯甲酮)。
-
对筛选出的特征峰进一步的注释和结构鉴定对下一步的生物学或毒理学意义解释至关重要。化合物结构注释需要将样品的质谱数据与数据库中的谱图进行比对打分,这其中涉及到的打分算法对匹配结果的可靠性至关重要[53-55]。另外,谱库中一般无法涵盖所有感兴趣化合物的二级谱图,二级谱图预测软件在一定程度上解决上述问题[56]。另外上述流程目前已经被整合到不同的数据处理软件中,各个数据处理软件算法及所选择的谱库的差异也会影响最后的分析结果。
化合物注释是指将样品谱图信息与谱库中已知化合物信息进行匹配,进而确定化合物的结构[37]。匹配样品质谱数据和谱库质谱数据的过程需要合适的匹配算法,当前DP(dot-product)算法使用最为广泛[54]。DP算法是指将质谱数据的m/z数值和峰强度数值看做二维向量,从而对样品质谱数据和谱库中对应的质谱数据进行余弦相似性计算,计算所得的余弦值大小即可反映两谱图类似性大小。进而,根据规定的余弦类似性阈值确定谱库中该谱图对应的化合物是否是样品谱图对应的化合物[53]。利用该算法,目前已有各种不同的非靶向处理软件或平台实现了自动化的谱图匹配、打分,最终筛选出了最可能的对应结构的化合物,从而达到样品谱图数据自动注释的功能[57-61]。如MS-DIAL在对原始样品的质谱数据进行峰提取、去冗余等步骤后,将样品质谱数据的MS和MS/MS信息与载入的谱图库进行对比,进而根据余弦类似性大小判断识别出的结构的可信性[62- 63]。在对市政污水厂污水样品进行分析时,Qian等[64]利用MS-DIAL对样品数据进行处理和结构注释,从而识别出568种化合物。
然而,由于当前质谱谱图数据库中所含的化合物数据有限,难以完全注释样品质谱数据中的特征峰,谱图库中不存在的化合物结构注释困难。利用二级谱图预测软件预测二级谱图从而对化合物结构进行注释或验证,或者直接利用二级谱图预测软件预测的谱图数据扩充谱图库进而进行化合物结构注释可以解决上述难题[65]。利用化合物在质谱仪中碎裂规则进行二级谱图预测的软件如Metfrag[66]和利用穷举法进行二级谱图的软件如Mass Frontier[67]均已有所应用。为了扩大对环境样品中全氟化合物及其类似物的发现,Getzinger等[68]利用CFM-ID二级谱图预测软件[69]扩充了全氟化合物的谱图库。并在DP算法的基础上构建了自动匹配及注释的流程,从而大大增加了环境样品中全氟化合物的识别数目。对化合物结构进行注释后,进一步的结构鉴定是确定所注释结构可信度的基础。目前, Schymanski等 [70]提出的化合物结构鉴定可信度标准已被广泛接受。
-
由于非靶向筛查的流程较长,不同的算法与软件共同存在,这会造成科研工作者在非靶向筛查质谱数据解析时面临困惑。如Hohrenk, Itzel, Baetz, Tuerk, Vosough 和Schmidt [12]比较了MZmine2, enviMass, Compound Discoverer和XCMS等4种常用的非靶向数据处理软件对同一批数据处理之间的一致性,发现对同一批样品,4个软件间共同识别出的的峰仅占10%,不同软件的数据处理一致性问题较大。作者指出各个软件之间算法的不同是造成之一差异的主要原因。Li等[70]从定量准确度、定性覆盖度等角度比较了MS-Dial, MZmine 2, XCMS, MarkerView及Compound Discoverer等5个软件的数据处理结果,发现5个软件在非靶向定量结果上与靶向定量的结果差异较大,其中MZmine 2是非靶向定量准确度最高的软件。因此针对不同非靶向数据处理软件和算法间的参数优化以及结合不同软件在数据处理方面的优势可能对解决数据处理一致性问题或更好的展示非靶处理结果有用[71]。
非靶向数据处理常用软件的参数优化方面,目前已有一些解决策略。Libiseller等[72]开发了基于XCMS非靶向筛查不同步骤参数优化的软件IPO。根据样品中天然同位素C13和C12相对丰度的比值关系构建优化方程,利用梯度下降法对XCMS非靶向数据处理过程中的参数如最小宽、最大峰宽等参数进行优化,从而将真阳性结果提高了146%—361%,减少了3%—8%的假阳性结果。Eliasson等 [73]通过将样品稀释成一定的浓度梯度进行检测,从而根据真实的峰在不同样品间存在这种浓度梯度信息,而仪器或背景噪音则不存在这种梯度信息,从而设计了相应的算法对XCMS数据处理过程中的参数进行优化。为了缩减计算机处理时间,该算法被进一步优化,从而使得数据处理的可信度提高了19.4%(标准混合物样品)和54.7%(人类尿液样品)[74]。McLean等[75]通过构建机器学习算法,利用梯度下降法不断优化XCMS及mzMine2在数据处理过程中的参数,从而得到最好的非靶向数据处理参数组合,减少假阳性,并是的检出的峰数目最多。
目前也有一些研究将不同软件及算法的优势进行结合,进而提高数据分析质量。为了提供给用户以最好的软件及算法的组合使用策略,Helmus等[10]将目前开源软件中的算法汇总,开发出了patRoon的R语言软件包,从而提高了数据处理的可信度。
-
本文详细介绍了化学信息学在非靶向筛查数据处理流程中的应用,重点阐述了该过程中涉及到的算法、软件、谱库等工具。基于目前产物识别方面存在的问题,提出以下展望:
(1) 不同算法和软件之间数据处理一致性问题。应进一步加强对不同软件、算法对质谱数据处理结果的比较、数据一致性的探讨。从而使得不同软件处理的质谱数据可以进行比较。
(2) 结构注释时谱库过小。应基于标准品和二级谱图预测得到的谱图进一步扩充谱图库,从而增加化合物结构注释的数目。
(3) 加强机器学习算法在非靶向数据处理过程中的应用。如利用机器学习算法更好的解决峰提取过程中假阳性与覆盖度低的问题。
化学信息学在液相色谱高分辨质谱联用的化学品非靶向筛查中的应用
Cheminformatics in untargeted screening of liquid chromatography coupled to mass spectrometry data
-
摘要: 化学工业的发展使得环境介质中未知化合物数目巨大,对其进行识别是认识其环境风险进而开发削减策略的关键。液相色谱串联高分辨质谱是化合物识别的常用技术,该技术采集的数据一般较复杂,需要适当的数据解析手段方能呈现复杂环境样品中的化合物信息,化学信息学在高分辨质谱非靶向筛查中的发展为化合物结构解析提供了可能。本文综述了化学信息学在非靶向筛查中的应用。基于非靶向筛查流程中的峰提取、去冗余、特征峰筛选、注释与结构确定步骤,从涉及的算法、软件、化合物数据库、谱图数据库等进行了阐述。在此基础上,对算法和软件工具的参数优化和数据处理一致性进行了阐述。本综述为更好的进行高分辨质谱数据非靶向处理提供了支撑。Abstract: The development of the chemical industry has resulted in the exposure of a huge number of unknown compounds in environment matrices. Identifying the chemicals is the key of assessing environmental risks of them and further attenuating them in environmental matrices. Liquid chromatography coupled to mass spectrometry (LC/MS) is a common technique of identifying compounds. However, the data collected by LC/MS is generally complex, which requires appropriate data analysis to reveal the information unrevealed in environmental samples. The development of analytical chemistry in untargeted screening of high-resolution mass spectrometry provides the possibility for compound structure identification. In this paper, we reviewed the application of analytical chemistry in untargeted screening, focused on the algorithm, software, compound database, spectrometry database and other aspects of the workflow such as process of peak extraction, de-redundancy, prioritization, annotation and structure determination. In addition, the parameter optimization and data processing consistency of algorithms and software tools are discussed. This review provides a better support of untargeted processing of high-resolution mass spectrometry data.
-
城市固体废弃物 (MSW) 的时变性会影响填埋体的稳定 [1-3]。MSW组分复杂,随着填埋龄期增加,大量有机物和纤维状物质产生降解,其抗剪强度也会随之改变。而抗剪强度的降低是造成分层垃圾填埋体滑移的主要原因[4-6]。填埋体的滑移失稳破坏会导致大量填埋垃圾和渗滤液滑出场外,造成严重的环境污染及财产损失[7]。
抗剪强度是MSW重要的力学性能之一,其变化规律与填埋龄期密切相关。随着龄期的增加,MSW的内摩擦角会增大,而粘聚力逐渐降低直至为0[8-10],这会导致填埋体沿衬垫发生滑移破坏。因此,土-膜界面的剪切特性得到了学者们的广泛研究[11-15]。PUNETHA等[13]通过直剪实验研究了光面及糙面HDPE土工膜和各类土颗粒物界面抗剪强度。BACAS等[14]对8种土工合成材料和18种不同界面进行了直剪实验,分析了界面剪切强度特性。LI等[15]研究了冻融循环作用下,密实粘土衬垫与HDPE土工膜界面剪切特性。在稳定性分析方面,ZIENKIEWIEZ等[16]首次将强度折减法引入到有限元边坡分析中。UGAI等[17]、郑颖人等[18]和陈雪珍等[19]将有限元分析的安全系数应用在工程中,推动有限元强度折减法的发展。
学者们通常以实验确定各层垃圾土的抗剪强度,对分层填埋场进行稳定性分析[4,6]。但目前对分层填埋场的稳定性分析都集中在以粘土作为中间衬垫,HDPE土工膜因为其耐久性好、化学性质稳定、柔韧性佳等特性而被视为替代粘土的理想材料[20],同时又缺乏将土工膜作为中间衬垫的应用研究。因此,本研究以HDPE土工膜代替粘土作为中间衬垫,进行土-膜界面剪切实验,利用PLAXIS有限元软件模拟分层填埋体的滑移过程及其整体稳定性变化,并分析HDPE土工膜作为中间衬垫的可行性,以期为土工膜应用于填埋场作为中间衬垫提供参考。
1. 材料与方法
1.1 实验材料
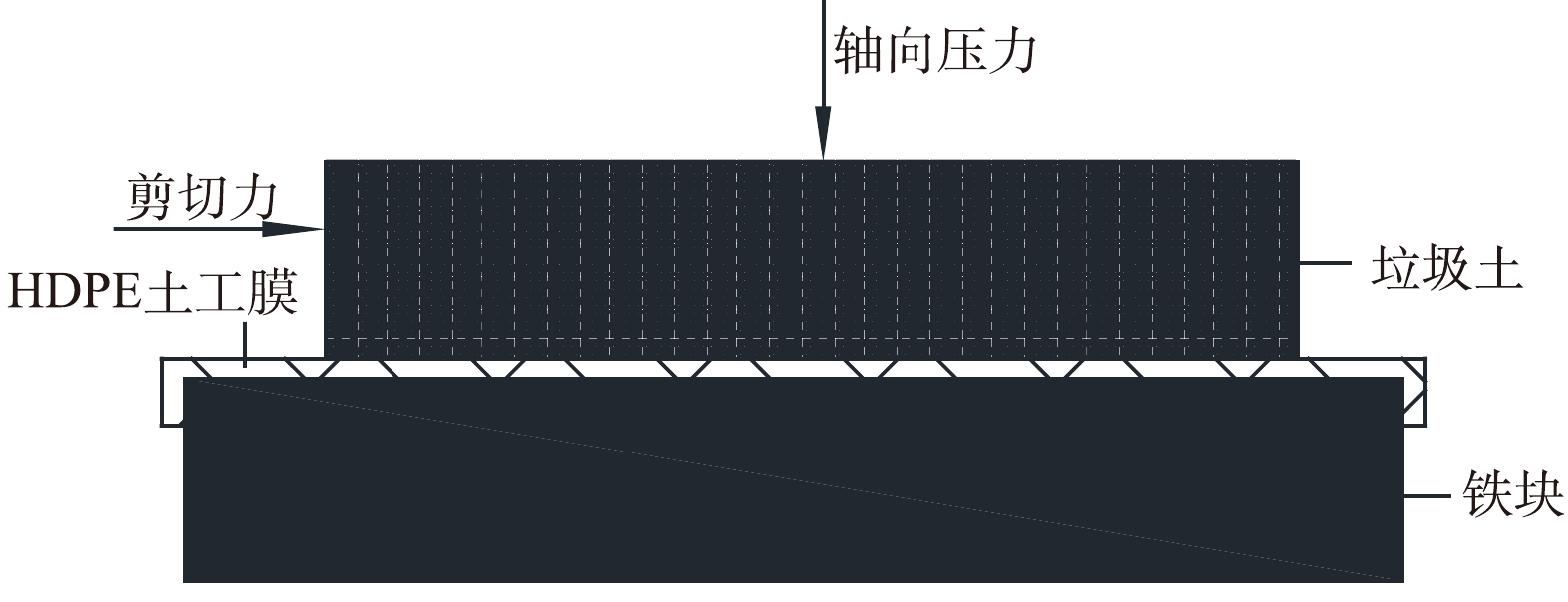
直剪实验的材料取自某苏南平原型垃圾填埋场,总计填埋库容量为476.5×104 m3。实验土样分别取自该场地的改扩建工程、二期工程、续建工程、续建二期工程。其物理指标如表1所示。选用3种不同规格的HDPE土工膜,分别为喷着式、柱点式和光面式。其中,柱点式表面排布规则的小凸点;喷着式表面具有不规则纹理,粗糙程度比柱点式低;光面式表面则是光滑的平面。3种土工膜厚度均为1.5 mm,密度大于0.94 g·cm-3,炭黑含量在2%~3%。HDPE土工膜的物理参数如表2所示。
表 1 MSW试样物理力学指标Table 1. Physical and mechanical indices of refuse MSW sample样品编号 填埋龄期/a 含水率 孔隙比 粘聚力/kPa 内摩擦角/ (°) Z1 1.5~2.0 45%~76% 2.33 23.3 9.8 Z2 4.5~5.0 1.65 23.8 17.5 Z3 8.0~9.0 1.96 16.1 26.0 Z4 10.0~13.0 2.62 2.8 34.2 表 2 屈服应变下HDPE土工膜的物理参数Table 2. Physical parameter of HDPE geomembrane under yield strain类型 厚度/mm 拉伸强度/(kN·m−1) 屈服应变 拉伸刚度/(kN·m−1) 柱点式 1.50 17.80 16.00% 111.25 光面 1.50 20.51 14.00% 146.50 喷着式 1.50 17.51 14.00% 125.07 1.2 实验装置
实验仪器与试样布置如图1所示,采用大尺寸直剪仪(THE-1000,天水红山实验机有限公司),上下剪切盒尺寸为500 mm×500 mm×410 mm,根据《土工合成材料测试规程》 (SL 235-2012) [21]进行实验 。
1.3 实验方法
将试样按龄期分为4组,每组选择50、100、150、200 kPa竖向荷载进行压缩,压缩至少4 h,剪应变不小于20%,剪切位移设置为100 mm,剪切速率为2 mm·min-1。以土-膜界面峰值强度表征土-膜界面强度[21]。依据莫尔-库伦破坏准则,得到不同龄期MSW试样与HDPE土工膜的界面抗剪强度参数关系。
2. 分层填埋体数值模拟
2.1 几何模型建立
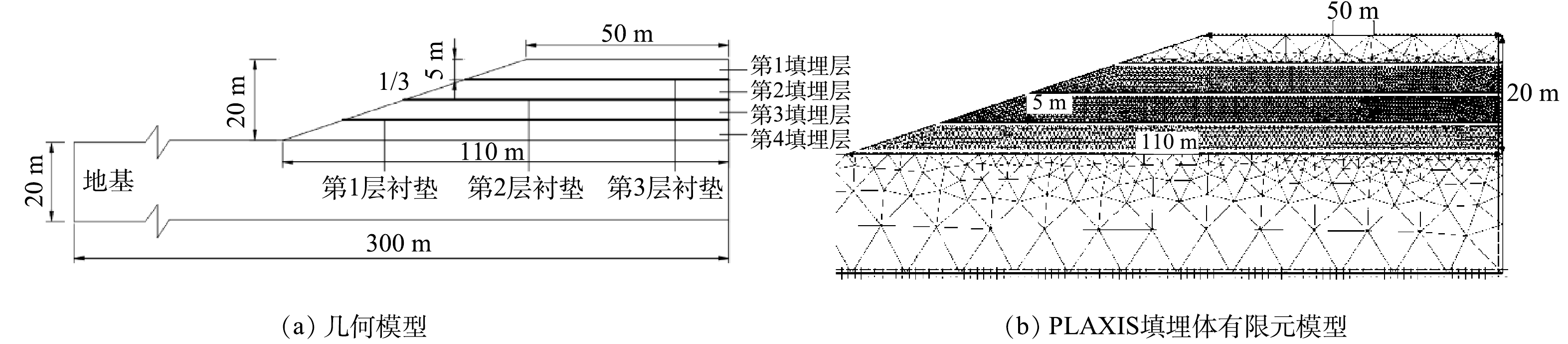
本研究参考某苏南平原型垃圾填埋场,建立的数值模型如图2所示。图2 (a) 从下至上为第1至第4填埋层;填埋体下底边长110 m、上底边长50 m、高20 m、斜坡度为1∶3,边界地基土深20 m、长300 m[22],分层处布设HDPE土工膜衬垫[23],有限元网格划分如图2 (b) 所示;不设HDPE土工膜的填埋体采用粘土封层,厚度为300 mm [22]。
2.2 计算参数确定
基于Mohr-Coulomb准则,不考虑渗流影响,土-膜界面相互作用按照强度折减计算。土体物理参数取值见表3。土体抗剪强度参数按照表1赋予每一填埋层。 HDPE土工膜厚度和拉伸刚度按照表2分别赋予每层衬垫。由于垃圾土无剪胀性,不考虑剪胀效应,剪胀角取值为0[24]。
表 3 各层土材料的参数Table 3. Table of material parameters of soil layers土层名称 压缩模量/MPa 泊松比 容重/(kN·m−3) 粘聚力/kPa 内摩擦角/ (°) 地基土层砂土状强风化花岗岩 30 0.25 19.5 35.0 28.5 粘土覆盖层 23 0.25 21.0 12.0 25.0 MSW第1填埋层 2 0.40 10.5 23.3 9.8 MSW第2填埋层 23.8 17.5 MSW第3填埋层 16.1 26.0 MSW第4填埋层 2.8 34.2 3. 结果与讨论
3.1 龄期对土-膜界面强度的影响
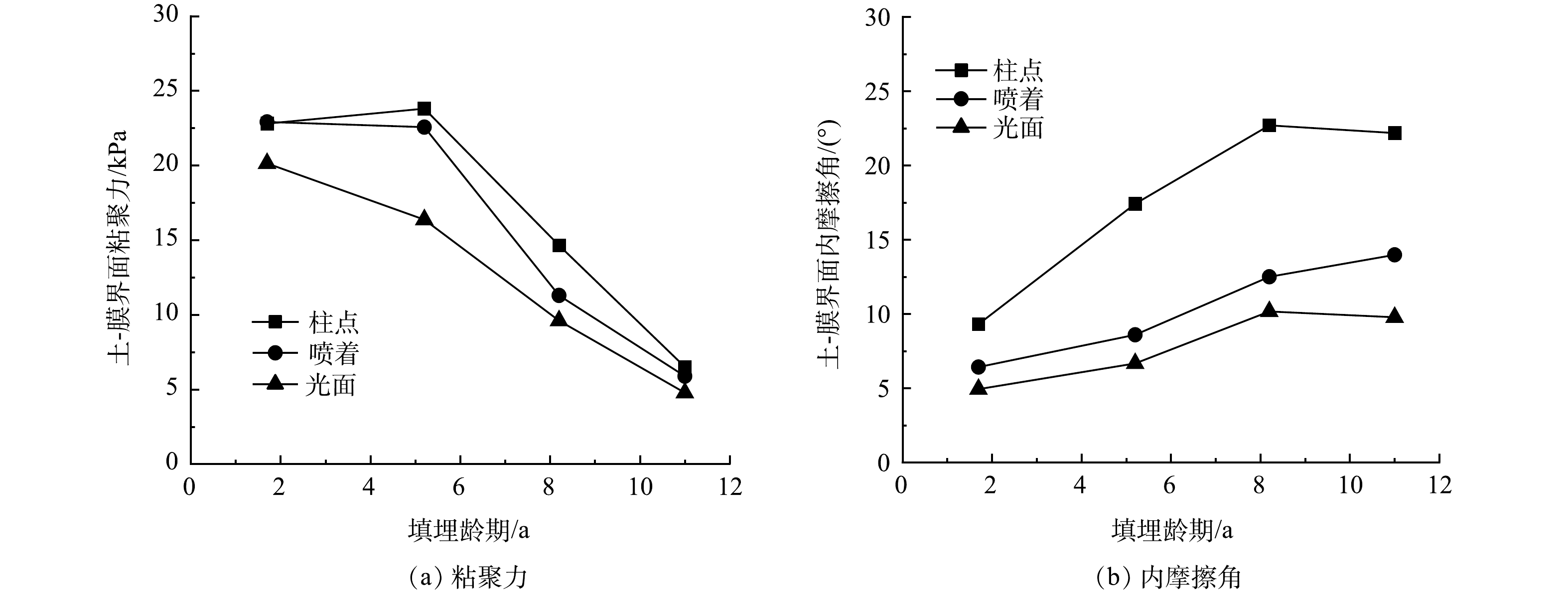
界面剪切实验结果如图3所示。由图3 (a) 可知,11 a内土-膜界面粘聚力呈现下降趋势,但在小于6 a时,土-膜界面粘聚力降低不明显。其原因可能是,在龄期较短时,MSW中有机质组分迅速降解,纤维物质降解缓慢导致其质量占比增加,因此,在剪切过程中能持续提供粘聚效果[9]。随着龄期增长,生物降解充分导致纤维物质含量降低,废渣等组分中颗粒含量增高[9],增大了土-膜界面的摩擦,因而,MSW失去粘聚力,土-膜界面内摩擦角呈现上升趋势,如图3 (b) 所示。垃圾土与HDPE土工膜界面剪切实验表明,相同的竖向荷载作用下,由于土工膜表面粗糙程度不同,随着法向应力的增大,表面越粗糙的的土工膜与MSW之间的摩擦越大,界面抗剪能力越好。3种不同土工膜按照剪切试验结果,抗剪强度表现为:柱点>喷着>光面。本实验研究了龄期影响下MSW与HDPE土工膜之间的界面抗剪强度变化特性,但未考虑土工膜本身随龄期变化产生的劣化影响。尽管HDPE土工膜相较于MSW受到龄期影响要小,但其水力性能会随时间下降并且产生而外的拉伸应变[25],此种影响仍不可忽略。因此,本次实验得到的数据结果较保守,龄期对HDPE土工膜材质的影响还有待研究。
3.2 土工膜作为中间衬垫的填埋体滑移分析
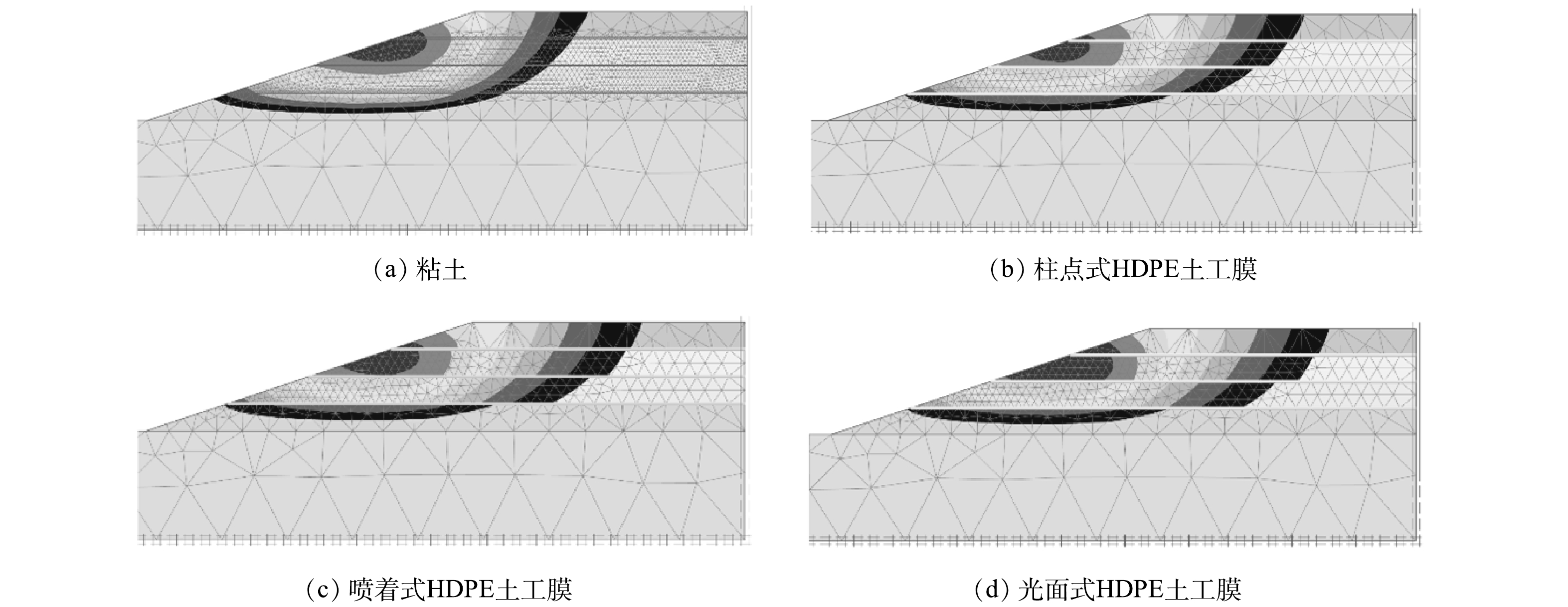
图4为不同材料作为中间衬垫的填埋体滑移情况,结果表明,分层填埋体在坡面中部产生应变集中。随着填埋龄期和高度的增加,MSW填埋体位移集中愈发明显,滑移区域不断扩展。结合实验结果可发现,可能是由于填埋体下层MSW的粘聚力较小,第4填埋层产生局部蠕变,随着龄期增长粘聚力进一步减小,坡体中部块体受重力影响沿坡面向坡脚推进,在坡面上部形成应变集中区,最终坡顶开始出现张拉裂缝,导致整个坡面滑移由表向里发展。图4 (a) 中临界滑动面连贯的穿透粘土衬垫,滑移面从坡顶向坡脚发展,直至贯通整个坡面。而HDPE土工膜替代粘土作为中间衬垫时,临界滑动面沿每层土-膜界面处均有一定滑移,在填埋体不同分层中错综发展,如图4 (b)~4 (d) 所示。图4滑移面分布情况表明,3种土工膜相较于粘土覆盖均延缓了上层滑移面的贯通,阻断了下方填埋体滑裂带的形成。根据实验结果可发现,首先,HDPE土工膜抗拉强度高于MSW,抵抗了上层填埋体对下层的剪应力;其次,由于每层MSW的参数不同,坡体下层填埋龄期较长,MSW的粘聚力很小,几乎接近于0,抗滑能力差,而上层填埋龄期较短,MSW的粘聚力跟内摩擦角能很好抵抗滑移。这解释了土-膜界面滑移由下至上越来越小的原因,但同时也会造成填埋体分层处的位移剧增。
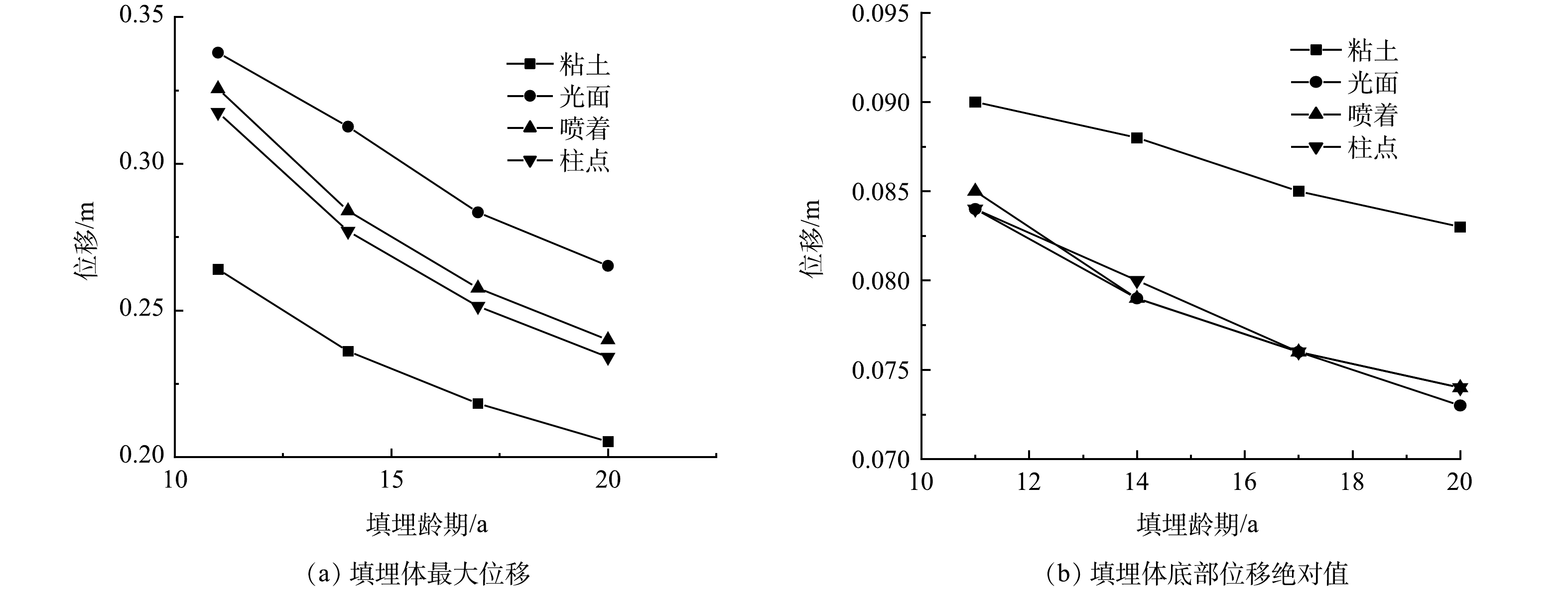
图5为11~20 a填埋体的位移数值变化。由图5 (a) 可知,临界滑动面在土-膜界面处产生水平滑移,故导致坡体最大位移明显高于粘土,分别比粘土覆盖的填埋体最大位移增加了29% (光面)、18% (喷着) 、17% (柱点) 。可以看出,具有一定表面粗糙度的柱点、喷着类型HDPE土工膜抗滑移能力稍好。由图5 (b) 可看出,底部位移绝对值由粘土覆盖的0.084 m减小到土工膜覆盖的0.073 m,降低了10%,3种类型HDPE土工膜均能很好抑制填埋体底部位移的发展。HDPE土工膜阻断了临界滑动面的形成,大大减小了底部位移。综合效果为:柱点>喷着>光面。
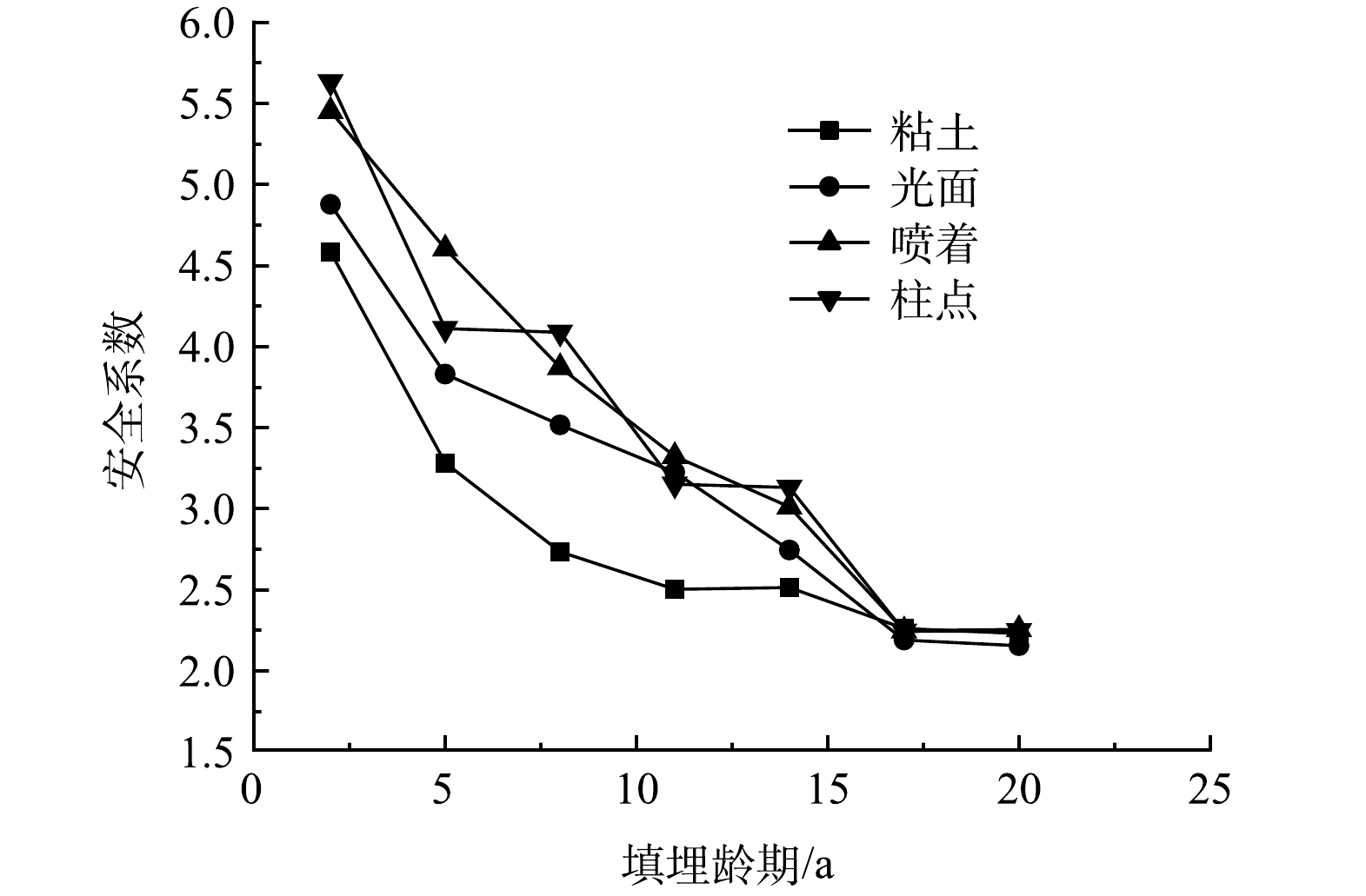
3.3 3种类型HDPE土工膜在龄期影响下填埋体稳定安全系数变化
图6为20 a内填埋体整体稳定安全系数的变化。根据《生活垃圾卫生填埋场岩土工程技术规范》 (CJJ176-2012) [23]规定,填埋体整体稳定安全系数需高于1.25才能保证其稳定,数值模拟结果均达到要求。在17 a内,HDPE土工膜作为中间衬垫的填埋体整体稳定安全系数均高于粘土覆盖的填埋体。17 a之后的整体稳定安全系数则与粘土衬垫接近。这可能是因为,土工膜材料自身劣化导致其力学性能降低至与粘土接近引起的[25]。在20 a的模拟过程中,柱点类型的整体稳定安全系数从5.632减小到2.246;喷着类型与柱点类型降幅相近,从5.456减小到2.256;光面类型的整体稳定安全系数从4.877减小到2.154,与粘土作为中间衬垫相比分别增大了 28% (柱点) 、30% (喷着) 和18.5% (光面) 。可见,当MSW进行分层填埋时,HDPE土工膜能够代替粘土作为中间衬垫,将起到很好的安全稳定作用。
4. 结论
1) 随着龄期增长,土-膜界面粘聚力总体呈现下降趋势,内摩擦角先上升而后趋于平稳。表面越粗糙的HDPE土工膜与MSW的摩擦越大,柱点类型HDPE土工膜有更好的界面抗剪效果。
2) HDPE土工膜比粘土衬垫更能减小MSW填埋体底部位移绝对值,但会增加最大位移。
3) HDPE土工膜能延缓上层滑移面的贯通、阻断下方填埋体滑裂带的形成,提升坡体稳定性。其中柱点式与喷着式土工膜较粘土衬垫能提高30%左右安全性,起到很好的安全稳定作用。
-
表 1 正负离子模式下常见的加和物
Table 1. The common adduct ion in positive and negative ion mode
ESI (+) ESI (−) M+H,M+Na,M+K M-H,M+Na-2H,M+K-2H M+NH4,M+ACN+H M+Cl,M+Br,M+FA-H M+2ACN+H, M+ACN+Na,M+CH3OH+H M+Hac-H,M+TFA-H,M-H2O-H -
[1] FANG W D, PENG Y, MUIR D, et al. A critical review of synthetic chemicals in surface waters of the US, the EU and China [J]. Environment International, 2019, 131: 104994. doi: 10.1016/j.envint.2019.104994 [2] HERNÁNDEZ F, BAKKER J, BIJLSMA L, et al. The role of analytical chemistry in exposure science: Focus on the aquatic environment [J]. Chemosphere, 2019, 222: 564-583. doi: 10.1016/j.chemosphere.2019.01.118 [3] STEHLE S, SCHULZ R. Agricultural insecticides threaten surface waters at the global scale [J]. PNAS, 2015, 112(18): 5750-5755. doi: 10.1073/pnas.1500232112 [4] OAKS J L, GILBERT M, VIRANI M Z, et al. Diclofenac residues as the cause of vulture population decline in Pakistan [J]. Nature, 2004, 427(6975): 630-633. doi: 10.1038/nature02317 [5] TANG Y, CRAVEN C B, WAWRYK N J P, et al. Advances in mass spectrometry-based omics analysis of trace organics in water. Trends in Analytical Chemistry[J], 2020, 128, 115918. [6] ESCHER B I, STAPLETON H M, SCHYMANSKI E L. Tracking complex mixtures of chemicals in our changing environment [J]. Science, 2020, 367(6476): 388-392. doi: 10.1126/science.aay6636 [7] SCHYMANSKI E L, JEON J, GULDE R, et al. Identifying small molecules via high resolution mass spectrometry: Communicating confidence [J]. Environmental Science & Technology, 2014, 48(4): 2097-2098. [8] ALYGIZAKIS N A, GAGO-FERRERO P, HOLLENDER J, et al. Untargeted time-pattern analysis of LC-HRMS data to detect spills and compounds with high fluctuation in influent wastewater [J]. Journal of Hazardous Materials, 2019, 361: 19-29. doi: 10.1016/j.jhazmat.2018.08.073 [9] WANG X B, YU N Y, YANG J P, et al. Suspect and non-target screening of pesticides and pharmaceuticals transformation products in wastewater using QTOF-MS [J]. Environment International, 2020, 137: 105599. doi: 10.1016/j.envint.2020.105599 [10] HELMUS R, TER LAAK TL, van WEZEL AP, et al. patRoon: Open source software platform for environmental mass spectrometry based non-target screening[J].Cheminform, 2021 , 13(1):1 . [11] SMITH C A, WANT E J, O'MAILLE G, et al. XCMS: processing mass spectrometry data for metabolite profiling using nonlinear peak alignment, matching, and identification [J]. Analytical Chemistry, 2006, 78(3): 779-787. doi: 10.1021/ac051437y [12] HOHRENK L L, ITZEL F, BAETZ N, et al. Comparison of software tools for liquid chromatography–high-resolution mass spectrometry data processing in nontarget screening of environmental samples [J]. Analytical Chemistry, 2020, 92(2): 1898-1907. doi: 10.1021/acs.analchem.9b04095 [13] MYERS O D, SUMNER S J, LI S Z, et al. One step forward for reducing false positive and false negative compound identifications from mass spectrometry metabolomics data: New algorithms for constructing extracted ion chromatograms and detecting chromatographic peaks [J]. Analytical Chemistry, 2017, 89(17): 8696-8703. doi: 10.1021/acs.analchem.7b00947 [14] TAUTENHAHN R, BÖTTCHER C, NEUMANN S. Highly sensitive feature detection for high resolution LC/MS [J]. BMC Bioinformatics, 2008, 9(1): 1-16. doi: 10.1186/1471-2105-9-1 [15] KATAJAMAA M, ORESIC M, Processing methods for differential analysis of LC/MS profile data[J]. BMC Bioinformatics, 2005, 6: 179. [16] PLUSKAL T, CASTILLO S, VILLAR-BRIONES A, et al. MZmine 2: Modular framework for processing, visualizing, and analyzing mass spectrometry-based molecular profile data [J]. BMC Bioinformatics, 2010, 11(1): 1-11. doi: 10.1186/1471-2105-11-1 [17] TAUTENHAHN R, PATTI G J, RINEHART D, et al. XCMS online: A web-based platform to process untargeted metabolomic data [J]. Analytical Chemistry, 2012, 84(11): 5035-5039. doi: 10.1021/ac300698c [18] STOLT R, TORGRIP R J O, LINDBERG J, et al. Second-order peak detection for multicomponent high-resolution LC/MS data [J]. Analytical Chemistry, 2006, 78(4): 975-983. doi: 10.1021/ac050980b [19] ÅBERG K M, TORGRIP R J O, KOLMERT J, et al. Feature detection and alignment of hyphenated chromatographic-mass spectrometric data: Extraction of pure ion chromatograms using Kalman tracking [J]. Journal of Chromatography A, 2008, 1192(1): 139-146. doi: 10.1016/j.chroma.2008.03.033 [20] NI Y, SU M M, QIU Y P, et al. ADAP-GC 3.0: Improved peak detection and deconvolution of co-eluting metabolites from GC/TOF-MS data for metabolomics studies [J]. Analytical Chemistry, 2016, 88(17): 8802-8811. doi: 10.1021/acs.analchem.6b02222 [21] DU X, SMIRNOV A, PLUSKAL T, et al. Metabolomics data preprocessing using ADAP and MZmine 2[J]. Methods Mol Biol. 2020;2104:25-48. [22] MYERS O D, SUMNER S J, LI S Z, et al. Detailed investigation and comparison of the XCMS and MZmine 2 chromatogram construction and chromatographic peak detection methods for preprocessing mass spectrometry metabolomics data [J]. Analytical Chemistry, 2017, 89(17): 8689-8695. doi: 10.1021/acs.analchem.7b01069 [23] HU Y X, CAI B, HUAN T. Enhancing metabolome coverage in data-dependent LC-MS/MS analysis through an integrated feature extraction strategy [J]. Analytical Chemistry, 2019, 91(22): 14433-14441. doi: 10.1021/acs.analchem.9b02980 [24] JU R, LIU X Y, ZHENG F J, et al. A graph density-based strategy for features fusion from different peak extract software to achieve more metabolites in metabolic profiling from high-resolution mass spectrometry [J]. Analytica Chimica Acta, 2020, 1139: 8-14. doi: 10.1016/j.aca.2020.09.029 [25] BAKER E S, PATTI G J. Perspectives on data analysis in metabolomics: Points of agreement and disagreement from the 2018 ASMS fall workshop [J]. Journal of the American Society for Mass Spectrometry, 2019, 30(10): 2031-2036. doi: 10.1007/s13361-019-02295-3 [26] KUHL C, TAUTENHAHN R, BÖTTCHER C, et al. CAMERA: an integrated strategy for compound spectra extraction and annotation of liquid chromatography/mass spectrometry data sets [J]. Analytical Chemistry, 2012, 84(1): 283-289. doi: 10.1021/ac202450g [27] SINDELAR M, PATTI G J. Chemical discovery in the era of metabolomics [J]. Journal of the American Chemical Society, 2020, 142(20): 9097-9105. doi: 10.1021/jacs.9b13198 [28] ZENG Z D, LIU X Y, DAI W D, et al. Ion fusion of high-resolution LC-MS-based metabolomics data to discover more reliable biomarkers [J]. Analytical Chemistry, 2014, 86(8): 3793-3800. doi: 10.1021/ac500878x [29] DEFELICE B C, MEHTA S S, SAMRA S, et al. Mass spectral feature list optimizer (MS-FLO): A tool to minimize false positive peak reports in untargeted liquid chromatography-mass spectroscopy (LC-MS) data processing [J]. Analytical Chemistry, 2017, 89(6): 3250-3255. doi: 10.1021/acs.analchem.6b04372 [30] SENAN O, AGUILAR-MOGAS A, NAVARRO M, et al. CliqueMS: a computational tool for annotating in-source metabolite ions from LC-MS untargeted metabolomics data based on a coelution similarity network [J]. Bioinformatics, 2019, 35(20): 4089-4097. doi: 10.1093/bioinformatics/btz207 [31] KÖPPE T, JEWELL K S, DIETRICH C, et al. Application of a non-target workflow for the identification of specific contaminants using the example of the Nidda river basin [J]. Water Research, 2020, 178: 115703. doi: 10.1016/j.watres.2020.115703 [32] BROECKLING C D, AFSAR F A, NEUMANN S, et al. RAMClust: a novel feature clustering method enables spectral-matching-based annotation for metabolomics data [J]. Analytical Chemistry, 2014, 86(14): 6812-6817. doi: 10.1021/ac501530d [33] JU R, LIU X Y, ZHENG F J, et al. Removal of false positive features to generate authentic peak table for high-resolution mass spectrometry-based metabolomics study [J]. Analytica Chimica Acta, 2019, 1067: 79-87. doi: 10.1016/j.aca.2019.04.011 [34] DALY R, ROGERS S, WANDY J, et al. MetAssign: probabilistic annotation of metabolites from LC-MS data using a Bayesian clustering approach [J]. Bioinformatics, 2014, 30(19): 2764-2771. doi: 10.1093/bioinformatics/btu370 [35] KOUŘIL Š, de SOUSA J, VÁCLAVÍK J, et al. CROP: correlation-based reduction of feature multiplicities in untargeted metabolomic data [J]. Bioinformatics, 2020, 36(9): 2941-2942. doi: 10.1093/bioinformatics/btaa012 [36] FRAISIER-VANNIER O, CHERVIN J, CABANAC G, et al. MS-CleanR: A feature-filtering workflow for untargeted LC-MS based metabolomics [J]. Analytical Chemistry, 2020, 92(14): 9971-9981. doi: 10.1021/acs.analchem.0c01594 [37] LJONCHEVA M, STEPIŠNIK T, DŽEROSKI S, et al. Cheminformatics in MS-based environmental exposomics: Current achievements and future directions [J]. Trends in Environmental Analytical Chemistry, 2020, 28: e00099. doi: 10.1016/j.teac.2020.e00099 [38] GORNIK T, KOVACIC A, HEATH E, et al. Biotransformation study of antidepressant sertraline and its removal during biological wastewater treatment [J]. Water Research, 2020, 181: 115864. doi: 10.1016/j.watres.2020.115864 [39] WEIZEL A, SCHLÜSENER M P, DIERKES G, et al. Analysis of the aerobic biodegradation of glucocorticoids: Elucidation of the kinetics and transformation reactions [J]. Water Research, 2020, 174: 115561. doi: 10.1016/j.watres.2020.115561 [40] PURSCHKE K, VOSOUGH M, LEONHARDT J, et al. Evaluation of nontarget long-term LC–HRMS time series data using multivariate statistical approaches [J]. Analytical Chemistry, 2020, 92(18): 12273-12281. doi: 10.1021/acs.analchem.0c01897 [41] HOHRENK L L, VOSOUGH M, SCHMIDT T C. Implementation of chemometric tools to improve data mining and prioritization in LC-HRMS for nontarget screening of organic micropollutants in complex water matrixes [J]. Analytical Chemistry, 2019, 91(14): 9213-9220. doi: 10.1021/acs.analchem.9b01984 [42] WANG X B, YU N Y, QIAN Y L, et al. Non-target and suspect screening of per- and polyfluoroalkyl substances in Chinese municipal wastewater treatment plants [J]. Water Research, 2020, 183: 115989. doi: 10.1016/j.watres.2020.115989 [43] LI Y Q, YU N Y, DU L T, et al. Transplacental transfer of per- and polyfluoroalkyl substances identified in paired maternal and cord sera using suspect and nontarget screening [J]. Environmental Science & Technology, 2020, 54(6): 3407-3416. [44] WANG Y, YU N Y, ZHU X B, et al. Suspect and nontarget screening of per- and polyfluoroalkyl substances in wastewater from a fluorochemical manufacturing park [J]. Environmental Science & Technology, 2018, 52(19): 11007-11016. [45] KOELMEL J P, PAIGE M K, ARISTIZABAL-HENAO J J, et al. Toward comprehensive per- and polyfluoroalkyl substances annotation using FluoroMatch software and intelligent high-resolution tandem mass spectrometry acquisition [J]. Analytical Chemistry, 2020, 92(16): 11186-11194. doi: 10.1021/acs.analchem.0c01591 [46] FU Y Q, ZHANG Y H, ZHOU Z H, et al. Screening and determination of potential risk substances based on liquid chromatography–high-resolution mass spectrometry [J]. Analytical Chemistry, 2018, 90(14): 8454-8461. doi: 10.1021/acs.analchem.8b01153 [47] ZHANG M J, LIU Y L, CHEN J, et al. Sensitive untargeted screening of nerve agents and their degradation products using liquid chromatography-high resolution mass spectrometry [J]. Analytical Chemistry, 2020, 92(15): 10578-10587. doi: 10.1021/acs.analchem.0c01508 [48] ESPOSITO G, TETA R, MARRONE R, et al. A fast detection strategy for cyanobacterial blooms and associated cyanotoxins (FDSCC) reveals the occurrence of lyngbyatoxin A in Campania (South Italy) [J]. Chemosphere, 2019, 225: 342-351. doi: 10.1016/j.chemosphere.2019.02.201 [49] TETA R, DELLA SALA G, GLUKHOV E, et al. Combined LC–MS/MS and molecular networking approach reveals new cyanotoxins from the 2014 cyanobacterial bloom in green lake, Seattle [J]. Environmental Science & Technology, 2015, 49(24): 14301-14310. [50] le DARÉ B, FERRON P J, ALLARD P M, et al. New insights into quetiapine metabolism using molecular networking [J]. Scientific Reports, 2020, 10(1): 19921. doi: 10.1038/s41598-020-77106-x [51] HOLLENDER J, SCHYMANSKI E L, SINGER H P, et al. Nontarget screening with high resolution mass spectrometry in the environment: Ready to go? [J]. Environmental Science & Technology, 2017, 51(20): 11505-11512. [52] POCHIRAJU S S, LINDEN K, GU A Z, et al. Development of a separation framework for effects-based targeted and non-targeted toxicological screening of water and wastewater [J]. Water Research, 2020, 170: 115289. doi: 10.1016/j.watres.2019.115289 [53] SCHEUBERT K, HUFSKY F, PETRAS D, et al. Significance estimation for large scale metabolomics annotations by spectral matching [J]. Nature Communications, 2017, 8: 1494. doi: 10.1038/s41467-017-01318-5 [54] STEIN S E, SCOTT D R. Optimization and testing of mass spectral library search algorithms for compound identification [J]. Journal of the American Society for Mass Spectrometry, 1994, 5(9): 859-866. doi: 10.1016/1044-0305(94)87009-8 [55] VINAIXA M, SCHYMANSKI E L, NEUMANN S, et al. Mass spectral databases for LC/MS- and GC/MS-based metabolomics: State of the field and future prospects [J]. TrAC Trends in Analytical Chemistry, 2016, 78: 23-35. [56] HUFSKY F, SCHEUBERT K, BÖCKER S. New kids on the block: Novel informatics methods for natural product discovery [J]. Natural Product Reports, 2014, 31(6): 807. doi: 10.1039/c3np70101h [57] XUE J C, GUIJAS C, BENTON H P, et al. METLIN MS2 molecular standards database: A broad chemical and biological resource [J]. Nature Methods, 2020, 17(10): 953-954. doi: 10.1038/s41592-020-0942-5 [58] TSUGAWA H, CAJKA T, KIND T, et al. MS-DIAL: Data-independent MS/MS deconvolution for comprehensive metabolome analysis [J]. Nature Methods, 2015, 12(6): 523-526. [59] DÜHRKOP K, FLEISCHAUER M, LUDWIG M, et al. SIRIUS 4: A rapid tool for turning tandem mass spectra into metabolite structure information [J]. Nature Methods, 2019, 16(4): 299-302. [60] RÖST H L, SACHSENBERG T, AICHE S, et al. OpenMS: A flexible open-source software platform for mass spectrometry data analysis [J]. Nature Methods, 2016, 13(9): 741-748. doi: 10.1038/nmeth.3959 [61] WANG M X, CARVER J J, PHELAN V V, et al. Sharing and community curation of mass spectrometry data with Global Natural Products Social Molecular Networking [J]. Nature Biotechnology, 2016, 34(8): 828-837. [62] TSUGAWA H, IKEDA K, TAKAHASHI M, et al. A lipidome atlas in MS-DIAL 4 [J]. Nature Biotechnology, 2020, 38(10): 1159-1163. doi: 10.1038/s41587-020-0531-2 [63] TSUGAWA H, NAKABAYASHI R, MORI T, et al. A cheminformatics approach to characterize metabolomes in stable-isotope-labeled organisms [J]. Nature Methods, 2019, 16(4): 295-298. doi: 10.1038/s41592-019-0358-2 [64] QIAN Y L, WANG X B, WU G, et al. Screening priority indicator pollutants in full-scale wastewater treatment plants by non-target analysis [J]. Journal of Hazardous Materials, 2021, 414: 125490. doi: 10.1016/j.jhazmat.2021.125490 [65] ALLARD P M, GENTA-JOUVE G, WOLFENDER J L. Deep metabolome annotation in natural products research: Towards a virtuous cycle in metabolite identification [J]. Current Opinion in Chemical Biology, 2017, 36: 40-49. doi: 10.1016/j.cbpa.2016.12.022 [66] RUTTKIES C, NEUMANN S, POSCH S. Improving MetFrag with statistical learning of fragment annotations [J]. BMC Bioinformatics, 2019, 20(1): 1-14. doi: 10.1186/s12859-018-2565-8 [67] SCHYMANSKI E L, GALLAMPOIS C M J, KRAUSS M, et al. Consensus structure elucidation combining GC/EI-MS, structure generation, and calculated properties [J]. Analytical Chemistry, 2012, 84(7): 3287-3295. doi: 10.1021/ac203471y [68] GETZINGER G J, HIGGINS C P, FERGUSON P L. Structure database and in silico spectral library for comprehensive suspect screening of per- and polyfluoroalkyl substances (PFASs) in environmental media by high-resolution mass spectrometry [J]. Analytical Chemistry, 2021, 93(5): 2820-2827. doi: 10.1021/acs.analchem.0c04109 [69] DJOUMBOU-FEUNANG Y, PON A, KARU N, et al. CFM-ID 3.0: Significantly improved ESI-MS/MS prediction and compound identification [J]. Metabolites, 2019, 9(4): 72. doi: 10.3390/metabo9040072 [70] LI Z C, LU Y, GUO Y F, et al. Comprehensive evaluation of untargeted metabolomics data processing software in feature detection, quantification and discriminating marker selection [J]. Analytica Chimica Acta, 2018, 1029: 50-57. doi: 10.1016/j.aca.2018.05.001 [71] COBLE J B, FRAGA C G. Comparative evaluation of preprocessing freeware on chromatography/mass spectrometry data for signature discovery [J]. Journal of Chromatography A, 2014, 1358: 155-164. doi: 10.1016/j.chroma.2014.06.100 [72] LIBISELLER G, DVORZAK M, KLEB U, et al. IPO: a tool for automated optimization of XCMS parameters [J]. BMC Bioinformatics, 2015, 16: 118. doi: 10.1186/s12859-015-0562-8 [73] ELIASSON M, RÄNNAR S, MADSEN R, et al. Strategy for optimizing LC-MS data processing in metabolomics: A design of experiments approach [J]. Analytical Chemistry, 2012, 84(15): 6869-6876. doi: 10.1021/ac301482k [74] ZHENG H, CLAUSEN M R, DALSGAARD T K, et al. Time-saving design of experiment protocol for optimization of LC-MS data processing in metabolomic approaches [J]. Analytical Chemistry, 2013, 85(15): 7109-7116. doi: 10.1021/ac4020325 [75] MCLEAN C, KUJAWINSKI E B. AutoTuner: High fidelity and robust parameter selection for metabolomics data processing [J]. Analytical Chemistry, 2020, 92(8): 5724-5732. doi: 10.1021/acs.analchem.9b04804 -



 DownLoad:
DownLoad: