水蚤分子生态毒理学研究进展
Advances in Ecotoxicogenomics with Water Fleas
-
摘要: 水蚤是广泛分布于各类淡水水体中的浮游动物,在水生生态系统中具有重要地位,也是水生毒理学研究中常用的模式生物。近年来,分子毒理学的发展为水蚤生态毒理学研究提供了新的工具和研究思路。本文分别从基因组学、转录组学、蛋白质组学、代谢组学和表观遗传组学方面,综述了不同环境污染物(重金属、农药和杀菌剂等有机污染物、环境激素类化合物、纳米材料和藻毒素等)对水蚤的生态毒理学效应及分子机制,为通过水蚤生态毒理学研究进行环境污染生物标志物筛选及生态风险评估提供参考。Abstract: Water fleas are the keystone aquatic organisms in most fresh water habitats, which have been often adopted as the model testing organisms in ecotoxicological experiments due to the advantages of high sensitivity to toxic substances, easy cultivation and high growth rate, etc. In recent years, the development of molecular toxicology has provided new tools and concept for the study of aquatic ecotoxicology. The omics-based technologies can facilitate molecular ecotoxicology studies in water fleas, better understanding and improving the knowledge on how environmental stressors, such as heavy metals, organic compounds and nanoparticles, cause toxicity in ecologically relevant organisms. In this review, the commonly used omic technologies, including genomic (or transcriptomic), protomic, metabolomic analyses as well as epigenetic technology have been discussed in order to provide references for the application of water fleas in ecotoxicology for screening of biomarkers and ecological risk assessment.
-
Key words:
- water flea /
- molecular ecotoxicology /
- ecotoxigenomics /
- biomarker
-
作为我国农业的支柱产业之一[1],畜禽养殖业向集约化、规模化方向发展,但规模化养殖产生了大量粪尿等污染物,畜禽养殖污染防治日益得到重视[2]。据相关统计[3]表明,以生猪养殖为代表的畜禽养殖业排放的有机物和总氮已经成为我国农业面源污染之首。
生猪养殖废水具有高氨氮(110~1 650 mg·L−1)、高有机污染物(2 000~30 000 mg·L−1)、高总氮(220~2 055 mg·L−1)等污染特征[4],其主要有还田处理,自然处理和生物处理等方式,其中还田处理和自然处理模式由于占地面积较大、二次污染等原因,而较少被采用。生物处理模式中多采用厌氧-好氧联合处理模式[5],但其存在处理工艺流程较长、出水难以稳定达标、冬季运行效果差等问题[6, 7]。另一方面,由于生猪养殖废水属于低C/N比废水(4~7),缺氧阶段缺乏足够碳源,从而影响微生物生长和反硝化脱氮效果[8]。为增强废水生物处理脱氮效率,需要外加碳源,导致其处理成本和能耗较高[9]。姜超等[10]和SUI等[11]通过建立关联氧化还原电位、pH控制点的SMBR工艺,通过优化好氧曝气时间实现了实时控制短程硝化过程,
NH+4 -N平均出水浓度为11.6 mg·L−1,去除率为98.3%;耗氧有机污染物的平均出水浓度(以COD计)为358 mg·L−1,去除率为95.3%;TN平均出水浓度为81 mg·L−1,去除率为92.7%;亚氮累积率在85%以上,因此,实现了常温下短流程高效处理畜禽养殖废水。本文在实验室规模SMBR工艺研究的基础上,通过研发中试规模SMBR工艺处理生猪养殖废水,考察了在低温环境下该工艺对污染物的去除效果及关键功能菌群的演替特征,其可为高效处理规模化畜禽养殖废水提供技术支撑。
1. 材料与方法
1.1 生猪养殖废水处理中试装置
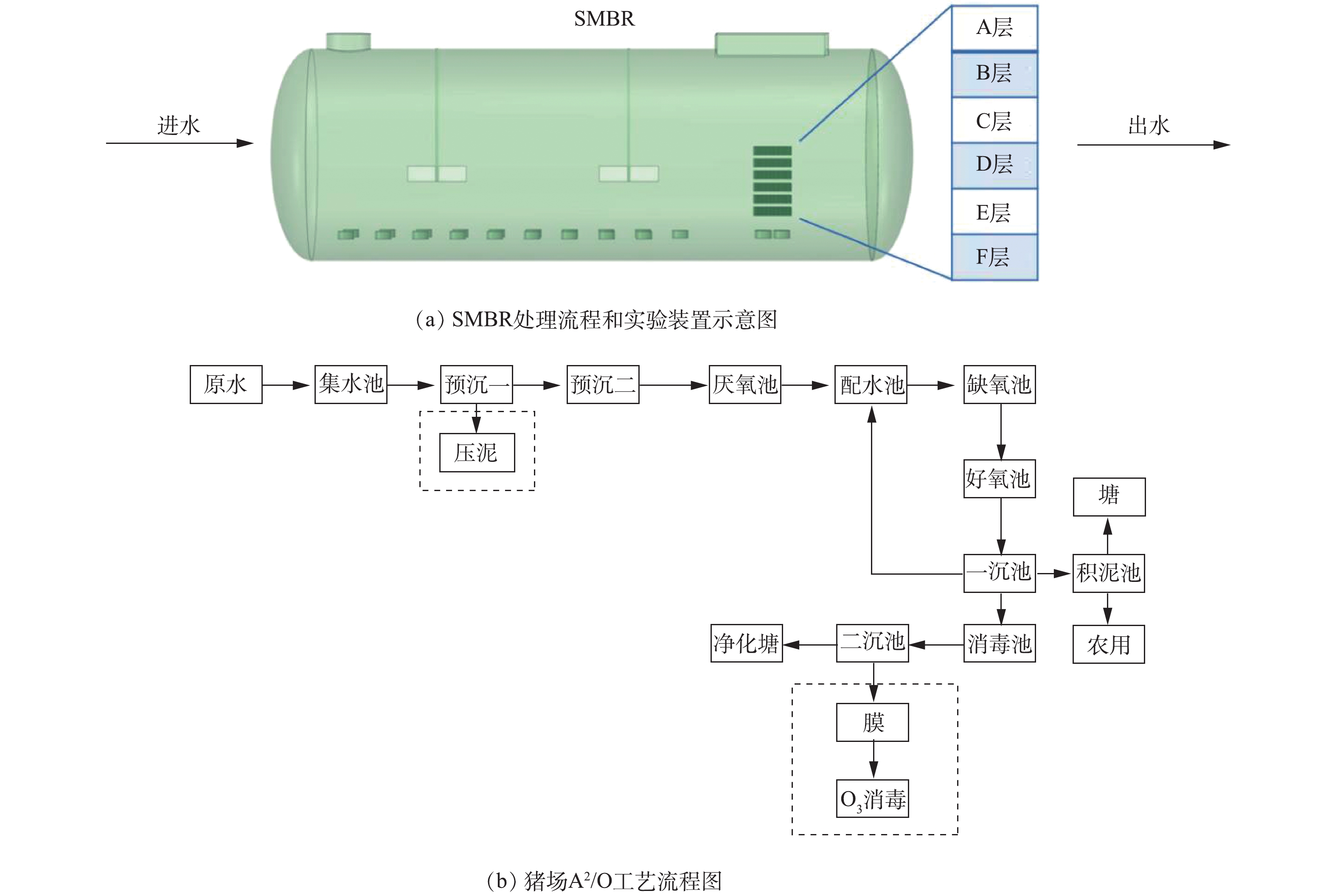
本中试研究在北京某养猪场实施。生猪养殖废水处理中试装置见图1。如图1(a)所示,本研究的中试规模序批式膜生物反应器(sequence membrane bio-reactor,SMBR)由碳钢(壁厚8 mm)制成,呈卧式圆柱体(2.8 m×8 m,总体积为50 m3,有效体积为30~35 m3,北京创迪环保科技有限公司)。如图1(a)所示,内置A~F共计6层陶瓷膜组件(膜孔径1 μm,单片膜单侧有效过滤面积0.5 m×0.11 m(共两侧),江西博鑫精陶环保科技有限公司),每层设置陶瓷膜48片,单片陶瓷膜短边垂直曝气管方向布置,膜片与膜片间隔0.8 cm,单层陶瓷膜设置1个出水管收集出水,6层(共288片陶瓷膜)设置统一出水管与抽吸泵相连,膜组件(共6层)总有效过滤面积为31.6 m2。SMBR设计处理规模10 m3·d−1,实际处理规模5~6 m3·d−1,SRT为15~20 d。反应器采用序批式模式运行:进水0.5 h、缺氧搅拌1.5 h、好氧曝气3 h(溶解氧为0.5~2 mg·L−1)、反应器膜出水0.6~1 h。
该猪场生猪养殖废水处理采用连续流厌氧-缺氧-好氧工艺(anaerobic-anoxic-oxic process, A2/O)(图1(b)),该生物反应器覆盖有阳光棚,冬季具有一定的保温作用,其主要运行参数见表1。
表 1 SMBR和A2/O运行基本参数Table 1. Basic parameters of SMBR and A2/O工艺 阶段或池型 处理量/(m3·d−1) SRT/d HRT/d  -N负荷/(kg·(m3·d)−1)
-N负荷/(kg·(m3·d)−1)有机物负荷/(kg(m3·d)−1) MLSS/(mg·L−1) 水温/℃ SMBR Ⅰ 10 15~20 5.6~6.6 0.11~0.14 1.48~1.75 7 200 20±2 Ⅱ 10 15~20 5.6~6.6 0.11~0.14 1.48~1.75 6 860 15±2 Ⅲ 10 15~20 5.6~6.6 0.11~0.14 1.48~1.75 8 240 10±2 A2/O 厌氧池 150 — 10 0.21 1.12~3.78 1 480 20±3 缺氧池 150 — 10 0.07 1.19~2.20 6 700 20±3 好氧池 150 — 10 0.01~0.02 0.35~0.8 4 340 20±3 1.2 实验用水与接种污泥
猪场A2/O工艺处理实验用水为生猪养殖废水经预沉淀、螺旋挤压分离、沉淀一池、沉淀二池处理。SMBR实验用水为北京市某猪场生猪养殖废水经预沉淀和螺旋挤压固液分离处理,其水质特征如表2所示。SMBR接种污泥取自该猪场废水处理站A2/O工艺中的好氧池污泥,首次接种后反应器内污泥浓度(MLSS)在6 000 mg·L−1左右,MLVSS在4 500 mg·L−1左右。
表 2 生猪养殖废水水质Table 2. Water quality parameters of swine wastewater阶段 水温/℃ COD/(mg·L−1)  -N/(mg·L−1)
-N/(mg·L−1) -N/(mg·L−1)
-N/(mg·L−1)TN/(mg·L−1) 阶段Ⅰ 20±2 9 816±2 190 733.44±78.90 16.96±11.68 880.31±112.24 阶段Ⅱ 15±2 9 520±938 766.50±66.63 36.10±9.47 882.36±69.02 阶段Ⅲ 10±2 8 532±2 668 724.00±72.99 43.31±7.16 904.30±206.32 1.3 水质指标测定方法
实验过程中对常规水质指标进行周期性采样检测,其中
NH+4 -N测试采用纳氏试剂比色法;NO−2 -N测试采用N-(1-萘基)-乙二胺光度法;NO−3 -N测试采用紫外分光光度法;总氮测试采用碱性过硫酸钾紫外分光光度法;有机污染物测定采用快速测定仪测试(哈希中国);SS、VSS测试采用重量法;pH、氧化还原电位和DO测试采用便携式测定仪(德国WTW)测定。1.4 DNA提取
分别在SMBR稳定运行的第28天(阶段Ⅰ)、第46天(阶段Ⅱ)和第63天(阶段Ⅲ),以及在现场A2/O工艺中的厌氧池、缺氧池、好氧池各取污泥混合液50 mL后,实验室提取每个样品1~5 mL,10 000 r·min−1离心10 min,弃上清液,使用试剂盒Fast DNA Spin Kit for Soil(MP, Biomedicals, USA)提取DNA。
1.5 高通量测序与OUT分类
采用Illumina Miseq平台(Illumina, USA)测序分析[11],测序数据经优化后,样品经均一化后均含有40 875条序列,有效序列采用(ribosomal database project, RDP)核糖体数据库进行物种分类。
2. 结果与讨论
2.1 SMBR的生猪养殖废水处理效果
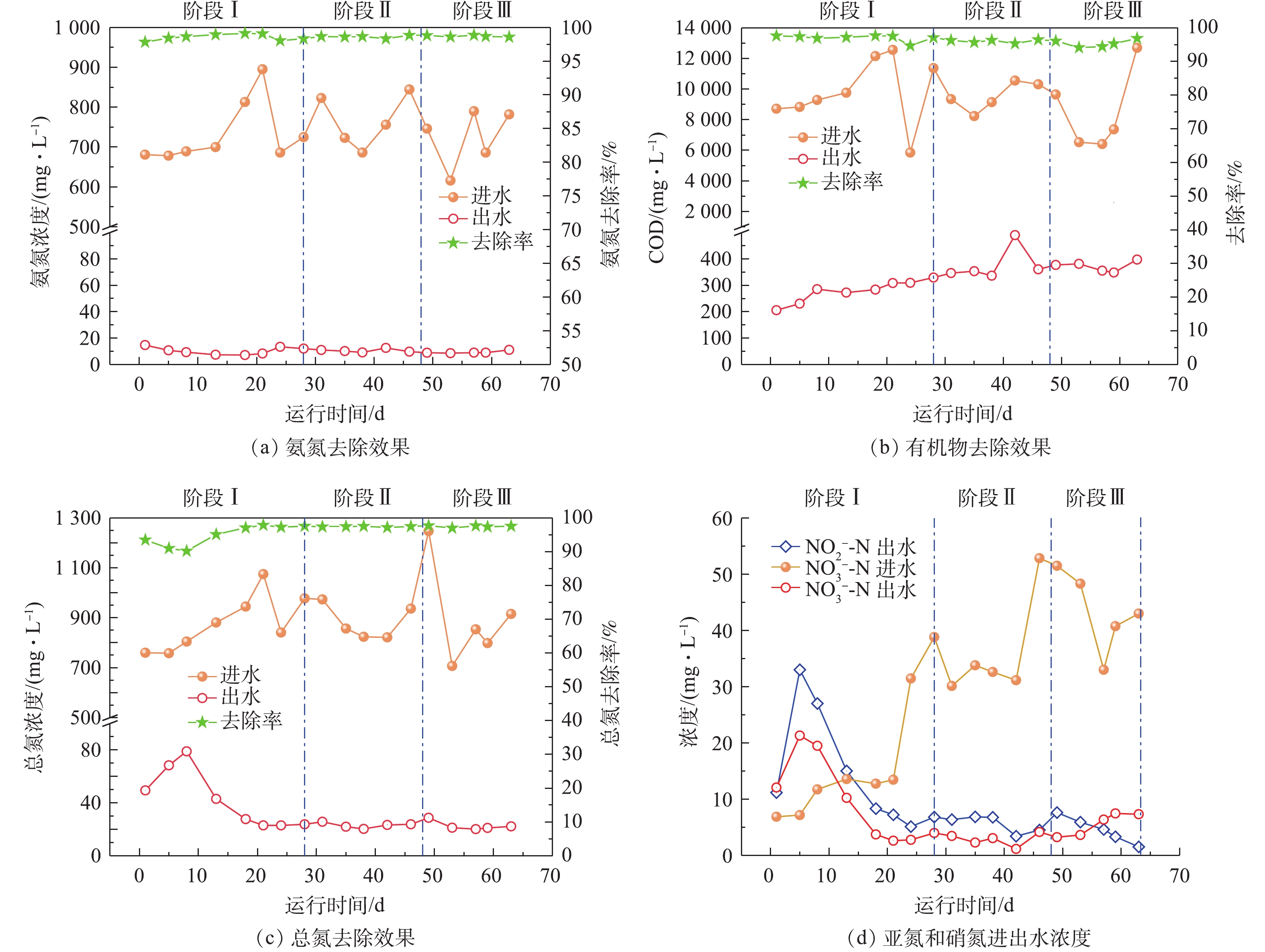
在SMBR的运行过程中,耗氧有机污染物的平均进水浓度(以COD计)为9 377 mg·L−1,平均出水浓度为332 mg·L−1,去除率为96.5%;总氮平均进水浓度为887 mg·L−1,平均出水浓度为31 mg·L−1,去除率为96.5%;氨氮平均进水浓度为740 mg·L−1,平均出水浓度为10 mg·L−1,去除率为98.6%;SS去除率可以达到100%。
SMBR去除污染物效果见图2。如图2(a)所示,在SMBR运行过程中,氨氮平均进水浓度为740 mg·L−1,平均出水浓度为10 mg·L−1,去除率高达98.6%,这表明尽管SMBR的运行温度逐步降低,但依然可以实现较好的氨氮去除效果;由图2(b)可以看出,耗氧有机污染物的出水浓度随着温度的降低有所增加,3个阶段耗氧有机污染物的平均出水浓度(以COD计)分别为279、378和373 mg·L−1,这说明反应器运行温度的降低可能会影响到微生物降解有机污染物的活性,导致出水有机物浓度随着反应器运行温度的降低有所提高;如图2(c)所示,在阶段Ⅰ初期,总氮平均进水浓度为880 mg·L−1,平均出水浓度逐步升高,在第8天达到最高出水浓度为79 mg·L−1,总氮去除率仅为89%,这是由于反应器启动初期,运行不稳定导致脱氮效果较差。此后当反应器稳定运行,出水总氮逐步降低,平均出水浓度降低至29 mg·L−1,总氮去除率提高至97%。
由于接种污泥来自好氧池排泥,含有高浓度的硝酸盐,以及反应器运行初期脱氮效果较差,所以导致硝态氮出水浓度高于硝态氮进水浓度(图2(d))。随着反应器的稳定运行,硝态氮出水浓度逐步降低至6 mg·L−1。在一定温度范围内(5~40 ℃),微生物的转化率与温度之间的关系可以用简化的阿伦尼乌斯方程[12]描述(式(1))。
rT=r293exp[−θ(293−T)] (1) 式中:rT为反应速率,mol·(L·s)−1;r293为标准温度下的反应速率,mol·(L·s)−1;θ为反应温度系数,K−1;T为反应温度,K。
在硝化反应中,反应温度系数θ随着温度的降低而降低[13]。当温度低于20 ℃时,亚硝化细菌最大比增长速率低于硝化细菌[14],亚硝化细菌产生的亚硝酸盐容易被硝化细菌继续氧化成硝酸盐。因此,在SMBR运行中随着水温的降低,亚硝化速率降低,出水亚氮浓度也逐步降低,阶段Ⅰ和阶段Ⅱ的亚氮累积率分别为62%和67%,而当水温降低至10 ℃后,亚氮累积率仅为44%。
总体而言,在SMBR整体运行过程中,虽然运行温度逐步降低,但是SMBR对污染物均具有较好的去除效果,能够获得较高且稳定的污染物去除率,满足《畜禽养殖业污染物排放标准》(GB 18596-2001)所规定的集约化畜禽养殖业污染物最高允许日均排放浓度。
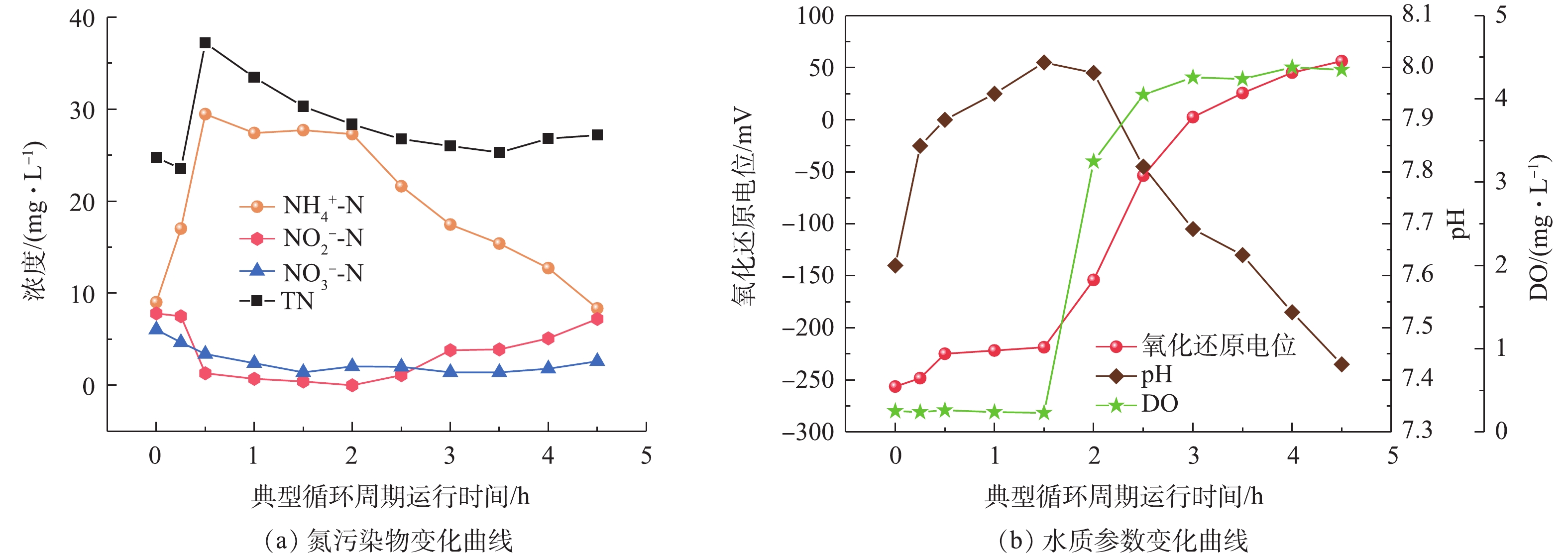
图3为SMBR运行第21天(阶段Ⅰ)的一个典型循环周期内,含氮污染物指标和水质参数的变化曲线。第0~0.5小时为反应器进水阶段,反应器氨氮浓度逐步升高,直至进水完成其浓度达到29.46 mg·L−1,此时反应器总氮浓度也达到37.2 mg·L−1。在缺氧搅拌期间,微生物利用进水中的有机物进行反硝化作用,亚硝酸盐和硝酸盐的浓度降低至0 mg·L−1和1.4 mg·L−1,总氮浓度也因为反硝化脱氮作用逐步降低至30.3 mg·L−1,在此期间产生的碱度使反应器中的pH不断升高。
第1.5~4.5小时为好氧单元,其中在曝气阶段的末端开启膜出水。在开启生物曝气的情况下,由于微生物的硝化作用,反应器内的氨氮浓度逐步降低至4.5 mg·L−1,亚氮浓度则是逐步升高至7.2 mg·L−1。由于反应器搅拌不均匀,存在死区,导致其成为缺氧环境,在好氧单元生成的亚氮或者硝氮在此发生反硝化作用,所以总氮在好氧单元略有降低。在好氧单元由于硝化作用消耗碱度,致使反应器在好氧单元期间实时pH逐步降低。
2.2 A2/O工艺处理生猪养殖废水效果
该猪场A2/O工艺的生猪养殖废水处理效果如表3所示。氨氮平均进水浓度为745 mg·L−1,出水浓度为7 mg·L−1,去除率达到99%;总氮平均进水浓度为844.5 mg·L−1,出水浓度为107 mg·L−1,去除率为87.2%;耗氧有机污染物的平均进水浓度(以COD计)为5 850 mg·L−1,出水浓度为216 mg·L−1,去除率达到96.3%。
表 3 A2/O工艺处理生猪养殖废水效果Table 3. Performance of A2/O process on swine wastewater treatmentmg·L−1 项目 氨氮 亚氮 硝态氮 总氮 COD 进水 745±79 0 37±9 844.5±5 5 850±56 厌氧池 835±1 0 23±3 909±50 2 245±91 缺氧池 65±9 0 20±3 96±31 274±21 好氧池 5±0.2 0 98±18 108±1 215±13 出水 7±2 0 94±25 107±7 216±10 去除率/% 99±0.2 — — 87.2±0.9 96.3±0.3 生猪养殖废水在进入厌氧池之前有曝气沉淀、螺旋挤压固液分离和沉淀池预处理等步骤,降低进水中的SS,其中,由于曝气沉淀预处理的持续曝气原因,导致废水中的部分氨氮通过氧化作用转化为硝氮,使得进入到厌氧池中的废水含有一定浓度的硝态氮。因此,厌氧池中实际为缺氧环境,微生物利用进水中的有机污染物进行反硝化,硝态氮浓度由37 mg·L−1降低至23 mg·L−1,COD值由5 850 mg·L−1降低至2 245 mg·L−1,有机物去除率达到61.6%。厌氧池出水到配水池,在此过程中一沉池污泥回流到配水池。在此过程中,由于污泥回流的稀释作用,导致配水池中的氨氮、总氮和COD大幅降低。在缺氧池中由于废水中缺少足够的可生物降解有机物,所以硝态氮在缺氧池仅去除3 mg·L−1左右。在好氧池中,溶解氧高达6.1~7.2 mg·L−1,微生物在好氧环境中充分发挥硝化作用,氨氮浓度由65 mg·L−1降低至5 mg·L−1,硝态氮由20 mg·L−1升高至98 mg·L−1。在A2/O工艺处理中,畜禽养殖废水的总氮去除率仅为87.2%,有机物去除率为96.3%,说明由于曝气池由于曝气过量,导致进水中的部分有机物在好氧池中进行氧化分解,并未完全利用进行反硝化,导致总氮去除率较低。
2.3 2种工艺的微生物群落结构演替变化
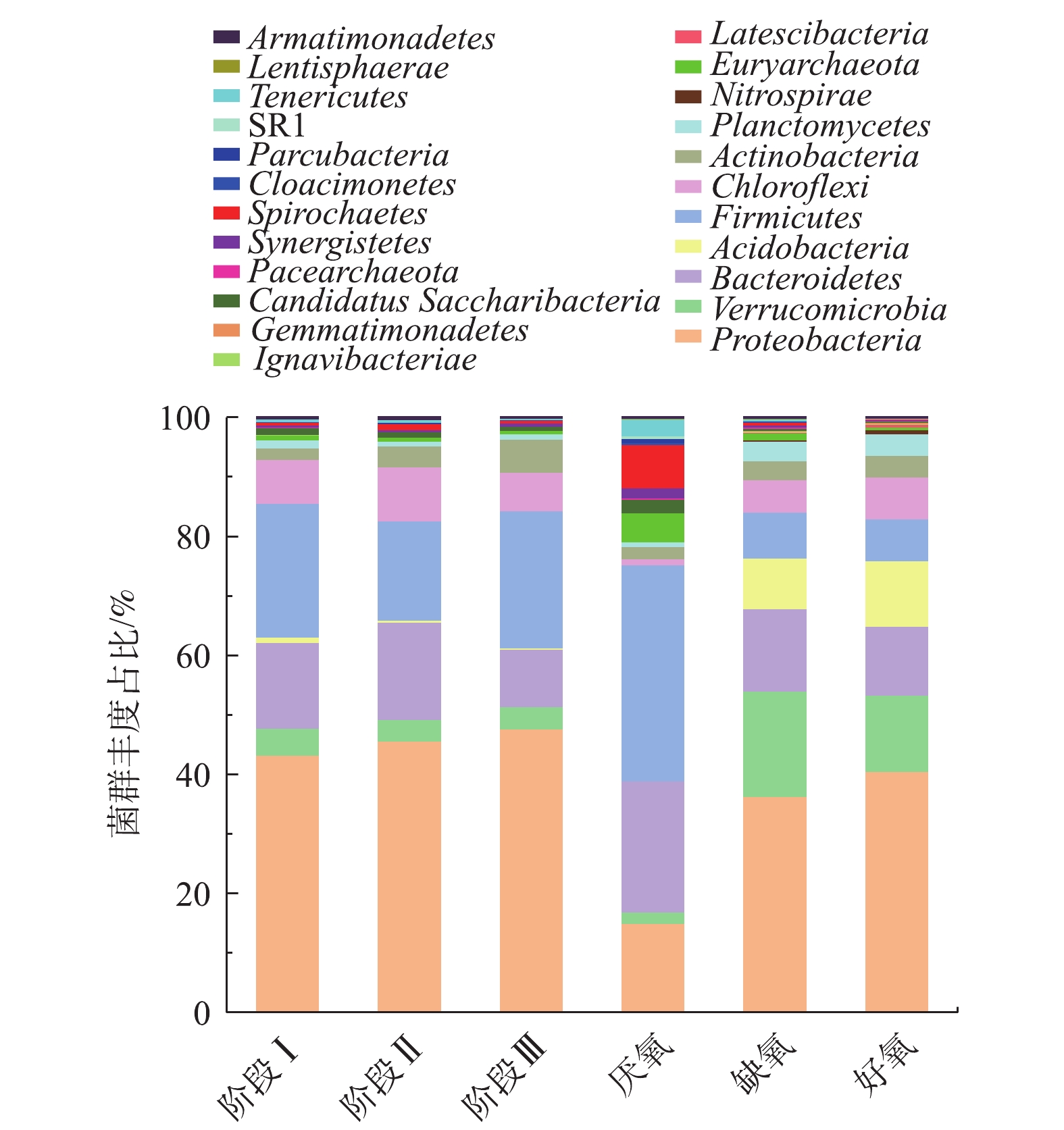
在不同运行温度下SMBR反应器的微生物群落结构表征如图4所示。在3个不同的运行温度下,SMBR反应器内门水平下的群落结构组成基本保持不变,主要由变形菌门(Proteobacteria)(43.29%~47.72%)、拟杆菌门(Bacteroidetes)(9.58%~16.32%)、绿弯菌门(Chloroflexi)(6.53%~9.06%)、厚壁菌门(Firmicutes)(16.67%~23.02%)等组成,其中,变形菌门包含了大多数具有硝化能力的菌属,如Nitrosomonas、Nitrosococcus、Nitrobacter、Nitrococcusi等硝化菌属[15],是污水处理过程的常见微生物[16]。随着运行温度的降低,变形菌门的丰度略有上升,而拟杆菌门和绿弯菌门的丰度则呈现降低的趋势。值得注意的是,放线菌门(Actinobacteria)的丰度随着温度的降低逐步上升。有研究[17]表明,放线菌过度繁殖是导致活性污泥丝状膨胀和起泡现象的主要原因。而在SMBR反应器中由于内置陶瓷膜过滤废水,所以尽管放线菌有增长的趋势,但是并不会导致污泥流失的情况发生。
A2/O工艺中厌氧池、缺氧池和好氧池的门水平群落结构丰度占比如图4所示。厌氧池中的变形菌门、拟杆菌门和厚壁菌门分别占比15%、22%和36%,其余细菌占比均低于10%;由于好氧池污泥回流到缺氧池,所以两者微生物群落组成类似,主要由变形菌门、疣微菌门(Verrucomicrobia)、拟杆菌门、酸杆菌门和厚壁菌门等菌群组成,其中,Verrucomicrobia门是高效活性污泥常见门类,对有机物的降解起重要作用[18]。
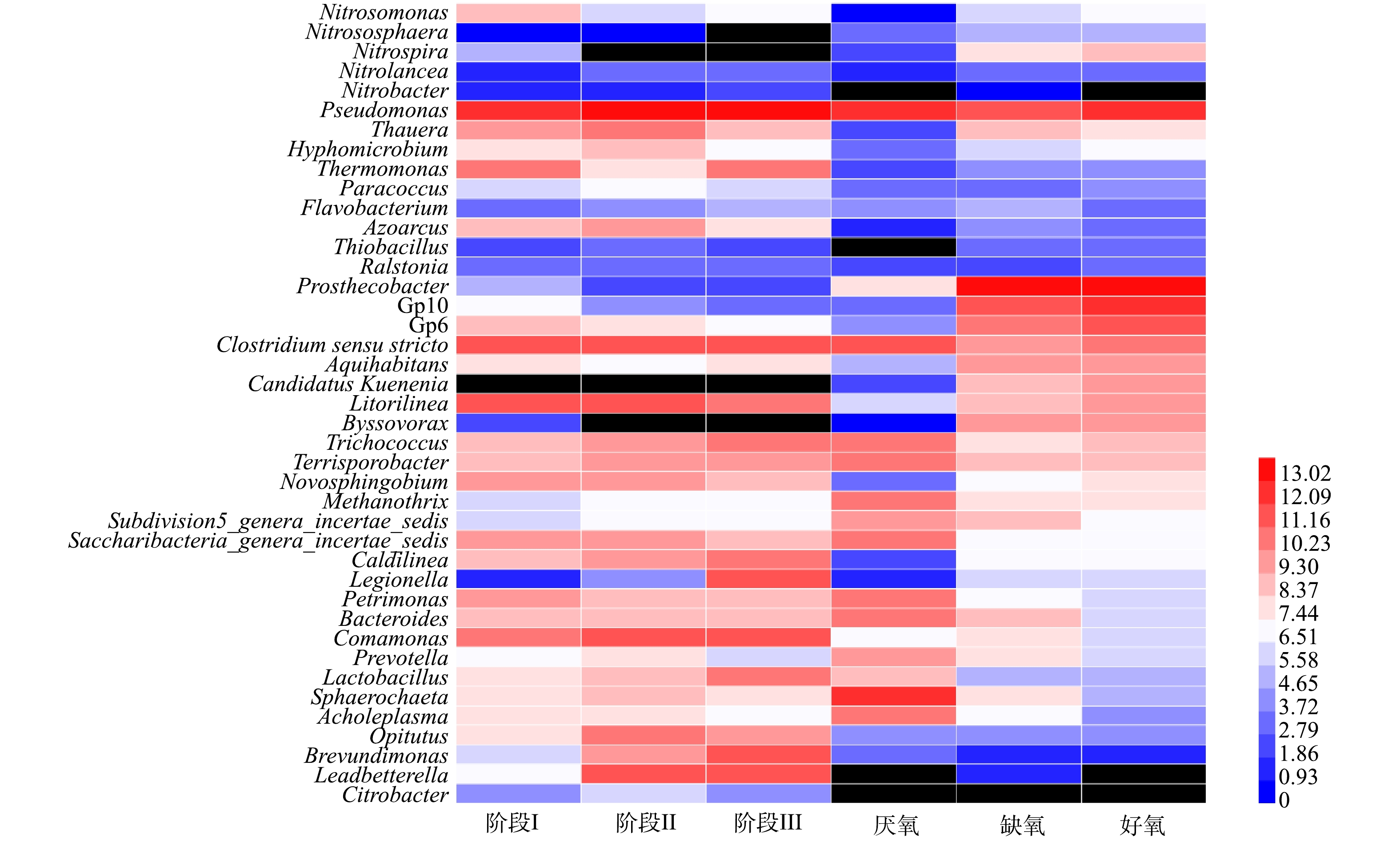
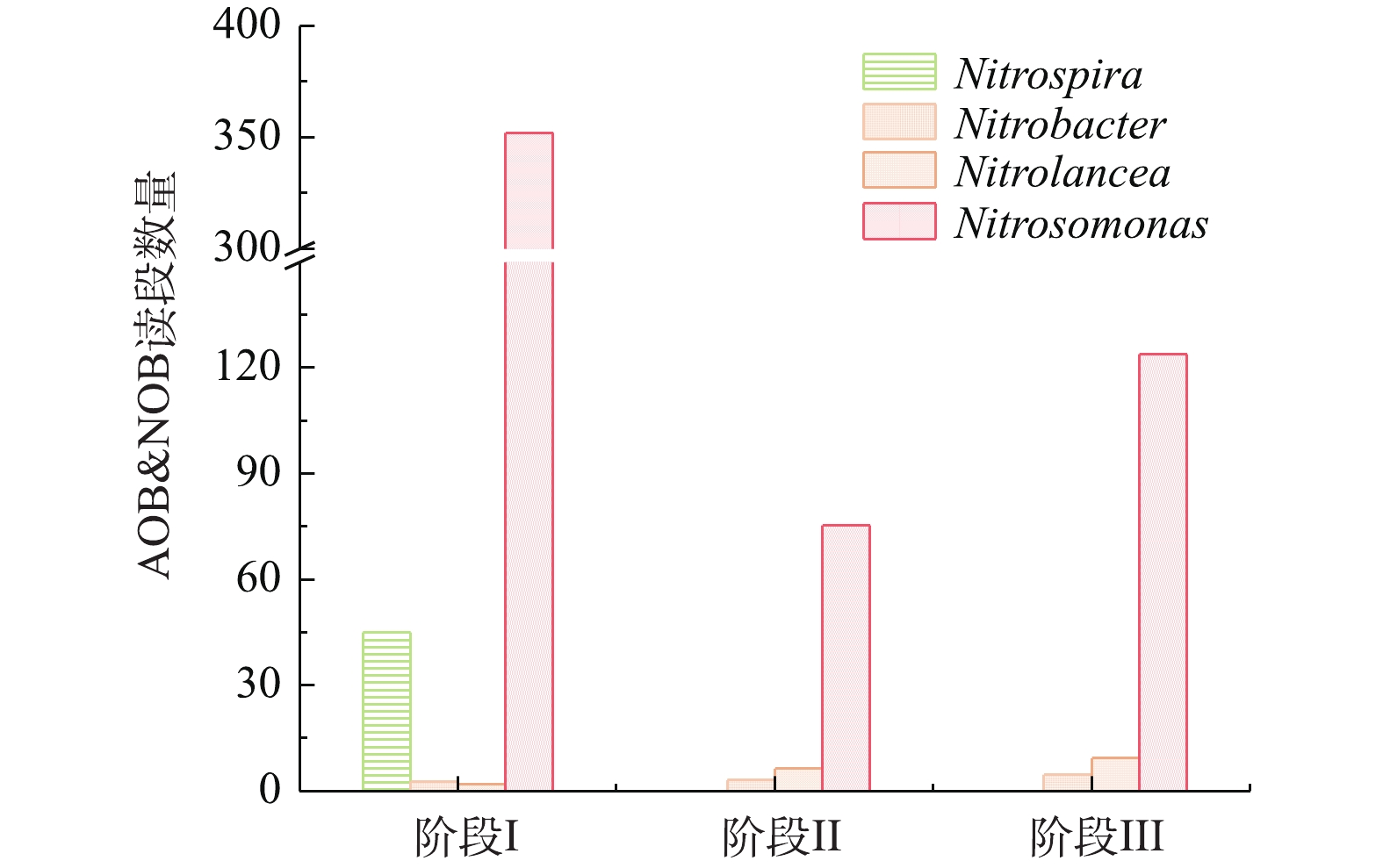
为了深入研究SMBR和A2/O工艺中的微生物群落组成,在属水平上进行了菌属丰度分析,主要结果见图5。Nitrosomonas、Nitrososphaera、Nitrospira、Nitrobacter和Nitrolancea等菌属是SMBR反应器和A2/O工艺的主要硝化菌属,反硝化菌属主要有Pseudomonas、Thauera、Hyphomicrobium、Thermomonas、Paracoccus、Flavobacterium、Azoarcus、Thiobacillus和Ralstonia等,并且SMBR反应器中反硝化菌属的丰度远高于A2/O工艺,这也是SMBR具有较高脱氮效率的原因之一。
图6反映了SMBR在不同阶段中AOB和NOB读段数量的变化。SMBR中主要的AOB为Nitrosomonas,NOB主要为Nitrospira、Nitrobacter和Nitrolancea。随着温度的降低,AOB和NOB的数量均有所降低,但是AOB仍然是主要的硝化细菌。结果表明,控制曝气时间,防止NOB将亚态氮转化为硝态氮,NOB未能获得足够的能量进行生长繁殖。随着SMBR反应器的稳定运行,NOB逐渐被淘汰,而AOB的优势地位越来越大,因此控制合适的曝气时间有利于AOB的富集和NOB的淘洗。
3. 结论
1) SMBR对畜禽养殖废水具有较好的处理效果,氨氮、总氮和有机物的去除率分别达到98.6%、96.5%和96.5%;猪场现有A2/O工艺对畜禽养殖废水中的氨氮、总氮和有机物的去除率分别为99%、88%和97%,其中总氮去除率低于SMBR的处理效果。
2)高通量测序与OTU分类结果表明,SMBR和A2/O工艺中具有类似的硝化菌属和反硝化菌属,但是SMBR的反硝化菌属的丰度较高,也是SMBR的脱氮效率高于A2/O的可能原因。
3)与现有A2/O工艺相比,SMBR工艺具有短流程、高效脱氮、稳定运行等优势,处理生猪养殖废水的应用前景广阔。
-
Kim H J, Koedrith P, Seo Y R. Ecotoxicogenomic approaches for understanding molecular mechanisms of environmental chemical toxicity using aquatic invertebrate, Daphnia model organism[J]. International Journal of Molecular Sciences, 2015, 16(6):12261-12287 Organization for Economic Cooperation and Development (OECD). Guideline for Testing of Chemicals. Daphnia sp., Acute Immobilisation Test. OECD 202[R]. Paris:OECD, 2004 Organization for Economic Cooperation and Development (OECD). Guidelines for Testing of Chemicals. Daphnia magna Reproduction Test. OECD 211[R]. Paris:OECD, 2012 Croce R, Cina F, Lombardo A, et al. Aquatic toxicity of several textile dye formulations:Acute and chronic assays with Daphnia magna and Raphidocelis subcapitata[J]. Ecotoxicology and Environmental Safety, 2017, 144:79-87 Vukov O, Smith D S, McGeer J C. Acute dysprosium toxicity to Daphnia pulex and Hyalella azteca and development of the biotic ligand approach[J]. Aquatic Toxicology, 2016, 170:142-151 Martins C, Jesus F T, Nogueira A J A. The effects of copper and zinc on survival, growth and reproduction of the cladoceran Daphnia longispina:Introducing new data in an "old" issue[J]. Ecotoxicology, 2017, 26:1157-1169 Garcia-Reyero N, Poynton H C, Kennedy A J, et al. Biomarker discovery and transcriptomic responses in Daphnia magna exposed to munitions constituents[J]. Environmental Science & Technology, 2009, 43:4188-4193 Taylor N S, Weber R J M, Southam A D, et al. A new approach to toxicity testing in Daphnia magna:Application of high throughput FT-ICR mass spectrometry metabolomics[J]. Metabolomics, 2009, 5:44-58 Watanabe H, Kobayashi K, Kato Y, et al. Transcriptome profiling in crustaceans as a tool for ecotoxicogenomics[J]. Cell Biology and Toxicology, 2008, 24:641-647 胡利腾,夏立萍,武敏敏,等.太平洋真宽水蚤(Eurytemora pacifica) Cu/ZnSOD基因克隆及在重金属胁迫下的表达分析[J].海洋与湖沼, 2018, 49(2):384-394 Hu L T, Xia L P, Wu M M, et al. Cloning and expression analysis of Cu/Zn SOD gene of Eurytemora pacifica under metal stress[J]. Oceanologia Et Limnologia Sinica, 2018, 49(2):384-394(in Chinese)
Roncalli V, Cieslak M C, Lenz P H. Transcriptomic responses of the calanoid copepod Calanus finmarchicus to the saxitoxin producing dinoflagellate Alexandrium fundyense[J]. Scientific Report, 2016, 6:25708-25720 韦晓慧.海洋酸化条件下铜、镉对日本虎斑猛水蚤(Tigriopus japonicus)发育、繁殖和超氧化物歧化酶活性的影响[D].青岛:中国海洋大学, 2013:1-3 Wei X H. Effect of simulated ocean acidification condition and heavy metals of Cu and Cd on the development, reproduction and SOD activity of Tigriopus japonicus[D]. Qingdao:Ocean University of China, 2013:1 -3(in Chinese)
Santos E M, Paull G C, van Look K J, et al. Gonadal transcriptome responses and physiological consequences of exposure to estrogen in breeding zebrafish (Danio rerio)[J]. Aquatic Toxicology, 2007, 83:134-142 Ju Z L, Wells M C, Walter R B. DNA microarray technology in toxicogenomics of aquatic models:Methods and applications[J]. Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology, 2007, 145:5-14 Costigan S L, Werner J, Ouellet J D, et al. Expression profiling and gene ontology analysis in fathead minnow (Pimephales promelas) liver following exposure to pulp and paper mill effluents[J]. Aquatic Toxicology, 2012, 122-123:44-55 Poynton H C, Varshavsky J R, Chang B, et al. Daphnia magna ecotoxicogenomics provides mechanistic insights into metal toxicity[J]. Environmental Science & Technology, 2007, 41:1044-1050 Poynton H C, Zuzow R, Loguinov A V, et al. Gene expression profiling in Daphnia magna, part Ⅱ:Validation of a copper specific gene expression signature with effluent from two copper mines in California[J]. Environmental Science & Technology, 2008, 42:6257-6263 Poynton H C, Lazorchak J M, Impellitteri C A, et al. Toxicogenomic responses of nanotoxicity in Daphnia magna exposed to silver nitrate and coated silver nanoparticles[J]. Environmental Science & Technology, 2012, 46:6288-6296 Soetaert A, Moens L N, van der Ven K, et al. Molecular impact of propiconazole on Daphnia magna using a reproduction-related cDNA array[J]. Comparative Biochemistry and Physiology Part C:Toxicology & Pharmacology, 2006, 142:66-76 Watanabe H, Tatarazako N, Oda S, et al. Analysis of expressed sequence tags of the water flea Daphnia magna[J]. Genome, 2005, 48:606-609 Heckmann L H, Connon R, Hutchinson T H, et al. Expression of target and reference genes in Daphnia magna exposed to ibuprofen[J]. BMC Genomics, 2006, 7:175 Olmstead A W, LeBlanc G A. Juvenoid hormone methyl farnesoate is a sex determinant in the crustacean Daphnia magna[J]. The Journal of Experimental Zoology, 2002, 293:736-739 Eads B D, Andrews J, Colbourne J K. Ecological genomics in Daphnia:Stress responses and environmental sex determination[J]. Heredity, 2008, 100(2):184-190 Jeong S W, Lee S M, Yum S S, et al. Genomic expression responses toward bisphenol-A toxicity in Daphnia magna in terms of reproductive activity[J]. Molecular & Cellular Toxicology, 2013, 9:149-158 Ansorge W J. Next-generation DNA sequencing techniques[J]. New Biotechnology, 2009, 25(4):195-203 Li Z, Li W, Zha J, et al. Transcriptome analysis reveals benzotriazole ultraviolet stabilizers regulate networks related to inflammation in juvenile zebrafish (Danio rerio) brain[J]. Environmental Toxicology, 2019, 34(2):112-122 Jackman K W, Veldhoen N, Miliano R C, et al. Transcriptomics investigation of thyroid hormone disruption in the olfactory system of the Rana[Lithobates] catesbeiana tadpole[J]. Aquatic Toxicology, 2018, 202:46-56 Yadetie F, Zhang X, Hanna E M, et al. RNA-Seq analysis of transcriptome responses in Atlantic cod (Gadus morhua) precision-cut liver slices exposed to benzo[a]pyrene and 17α-ethynylestradiol[J]. Aquatic Toxicology, 2018, 201:174-186 Colli-Dula R C, Fang X, Moraga-Amador D, et al. Transcriptome analysis reveals novel insights into the response of low-dose benzo (a) pyrene exposure in male tilapia[J]. Aquatic Toxicology, 2018, 201:162-173 Beauvais-Flück R, Slaveykova V I, Cosio C. Effects of two-hour exposure to environmental and high concentrations of methylmercury on the transcriptome of the macrophyte Elodea nuttallii[J]. Aquatic Toxicology, 2018, 194:103-111 Orsini L, Gilbert D, Podicheti R, et al. Daphnia magna transcriptome by RNA-Seq across 12 environmental stressors[J]. Scientific Data, 2017, 31(4):170006 Russo C, Isidori M, Deaver J A, et al. Toxicogenomic responses of low level anticancer drug exposures in Daphnia magna[J]. Aquatic Toxicology, 2018, 203:45-50 Schwarzenberger A, Sadler T, Motameny S, et al. Deciphering the genetic basis of microcystin tolerance[J]. BMC Genomics, 2014, 15:776-784 Le T H, Lim E S, Hong N H, et al. Proteomic analysis in Daphnia magna exposed to As (Ⅲ), As (Ⅴ) and Cd heavy metals and their binary mixtures for screening potential biomarkers[J]. Chemosphere, 2013, 93:2341-2348 Rainville L C, Carolan D, Varela A C, et al. Proteomic evaluation of citrate-coated silver nanoparticles toxicity in Daphnia magna[J]. Analyst, 2014, 7:1678-1686 Schwarzenberger A, Zitt A, Kroth P, et al. Gene expression and activity of digestive proteases in Daphnia:Effects of cyanobacterial protease inhibitors[J]. BMC Physiology, 2010, 10:6 Taylor N S, Weber R J M, Southam A D, et al. A new approach to toxicity testing in Daphnia magna:Application of high throughput FT-ICR mass spectrometry metabolomics[J]. Metabolomics, 2009, 5:44-58 Taylor N S, Weber R J, White T A, et al. Discriminating between different acute chemical toxicities via changes in the Daphnid metabolome[J]. Toxicology Sciences, 2010, 118:307-317 Poynton H C, Taylor N S, Hicks J, et al. Metabolomics of microliter hemolymph samples enables an improved understanding of the combined metabolic and transcriptional responses of Daphnia magna to cadmium[J]. Environmental Science & Technology, 2011, 45:3710-3717 Nagato E G, D'eon J C, Lankadurai B P, et al. H-1 NMR-based metabolomics investigation of Daphnia magna responses to sub-lethal exposure to arsenic, copper and lithium[J]. Chemosphere, 2013, 93:331-337 Taylor N S, Gavin A, Viant M R. Metabolomics discovers early-response metabolic biomarkers that can predict chronic reproductive fitness in individual Daphnia magna[J]. Metabolites, 2018, 8(3):42 Vandegehuchte M B, Lemière F, Vanhaecke L, et al. Direct and transgenerational impact on Daphnia magna of chemicals with a known effect on DNA methylation[J]. Comparative Biochemistry Physiology Part C:Toxicology & Pharmacology, 2010, 151:278-285 Vandegehuchte M B, De Coninck D, Vandenbrouck T, et al. Gene transcription profiles, global DNA methylation and potential transgenerational epigenetic effects related to Zn exposure history in Daphnia magna[J]. Environmental Pollution, 2010, 158(10):3323-3329 Vandegehuchte M B, Lemière F, Janssen C R. Quantitative DNA-methylation in Daphnia magna and effects of multigeneration Zn exposure[J]. Comparative Biochemistry and Physiology. Part C:Toxicology & Pharmacology, 2009, 150(3):343-348 -
 点击查看大图
点击查看大图
计量
- 文章访问数: 6621
- HTML全文浏览数: 6621
- PDF下载数: 206
- 施引文献: 0



 下载:
下载: