化学品是指人工合成或从自然界中提取、富集,具有特定功能属性的化学物质。化学品在促进人类社会的发展、提升生活质量方面,发挥了重要作用。然而,随着化学品使用量不断增加,其对人体和生态健康的不利效应逐渐显现。众多研究表明,化学品在人和生物体内广泛检出[1-4],且表现出神经毒性、发育毒性、内分泌干扰效应和生殖毒性等有害效应[5-8]。因此,化学品也被认为是威胁人体与生态健康的重要风险源,有必要对其开展风险评价并采取相应的风险管控措施[9]。
生物积累性是化学品风险评价的重要方面。一般认为,生物积累性表征化学品在生物体内的积累潜力,泛指化学品在生物体内积累,使生物体内浓度(cO)远高于周围环境中浓度的现象[10]。目前,生物积累性评价已被纳入欧盟《化学品的注册、评估、授权和限制(REACH)》法规[11]。我国2020年颁布的《新化学物质环境管理登记指南》中明确规定,应重点管控环境持久性、生物积累性和有毒的化学物质[12]。因此,筛查具有生物积累性的化学品,尤其重要。
化学品的生物积累性常采用生物富集因子(BCF)和生物放大因子(BMF)等参数衡量,其中BCF指仅经水暴露达到平衡时,cO与水中化学品浓度(cW)的比值;BMF指经摄食暴露达到平衡时,cO与食物中化学品浓度(cFood)之比[10]。化学品的BCF和BMF值,可通过实验测试获取。最常使用的标准测试方法为经济合作与发展组织(OECD)发布的鱼类生物积累测试导则(简称OECD TG305导则)[13]。然而,基于该方法获取化学品的生物积累参数存在实验周期长、测试成本高且违背动物伦理3R (replacement, reduction, refinement)原则的问题,难以满足大量化学品生物积累性评价的需求。因此,有必要发展化学品生物积累参数的高通量预测方法。
近年来,机器学习算法飞速发展,已被成功用于预测化学品的生物积累参数。例如,Zhao等[14]采用多元线性回归(MLR)、支持向量机(SVM)和径向基函数神经网络(RBFNN),建立了预测473种有机化学品BCF的单一算法和集成算法模型。Dearden和Hewitt[15]采用人工神经网络(ANN),建立了624种化学品BCF的预测模型。Strempel等[16]基于条件推理树(CIT)和随机森林(RF)算法,构建了713种有机化合物BCF的分类和回归模型。郑玉婷等[17]采用MLR,构建了780种有机化合物BCF的预测模型。丁蕊等[18]基于集成学习算法,构建了鱼体内1 384种有机化学品BCF的预测模型。Fatemi等[19]使用ANN,建立了36个有机氯化合物BMF的预测模型。综上,现有生物积累参数的模型预测终点集中于BCF,而针对BMF的预测模型相对较少。此外,这些模型均将BCF和BMF作为2个独立的终点来预测,即单任务(single-task, ST)模型,需要对每个终点进行建模,也忽略了2种参数之间的关联信息,导致模型预测准确性有限。基于多任务学习(multi-task learning, MTL)算法构建的多任务(multi-task, MT)模型,有望解决上述问题。
MTL本质上是迁移学习的一种,通过共享表示信息,可以学习不同任务与特征的相关性,自动使模型聚焦到有用的特征上,进而实现注意力聚焦[20-22]。同时,MTL也能克服单任务学习中由于任务与特征之间交互复杂导致的特征无法获取的问题,可以高效识别对任务预测重要的特征。总之,MTL是通过学习相关任务间的共性和差异信息,改善各个任务的预测性能,实现多个终点的同时预测。相比ST学习,MTL的泛化能力更强,且适用于数据量较少的数据集[21-23]。
目前,MTL已被成功用于预测化学品的理化性质和毒理效应。例如,前人利用MTL,结合深度神经网络(DNN)算法,构建了同时预测化学品水溶解度(logS)和分配系数(logP)的MT-DNN模型,其预测准确性优于ST模型[24]。Wu和Wei[25]通过构建MT-DNN模型,预测了化学品对4种生物(黑头呆鱼(Pimephales promelas)、大型溞(Daphnia magna)、梨形四膜虫(Tetrahymena pyriformis)和大鼠(Rattus norvegicus))的急性毒性值(LC50或LD50),结果表明MT-DNN模型的预测准确性高于集成模型。然而,目前尚未有将MTL算法应用于生物积累参数预测的研究。BCF和BMF均表征化学物质在生物体内的积累潜力,可合理假设二者具有相似的内在机理。因此,MTL算法可在化学品生物积累参数预测方面发挥作用。综上,本研究旨在基于MTL策略,创建有机化学品生物积累参数的MT模型。
从文献[18-19,26-29]和权威数据库(NITE (https://www.nite.go.jp/en/index.html), CEFIC-LRI (http://cefic-lri.org/), ECHA eChemPortal (https://www.echemportal.org/echemportal/), ECOTOX (https://cfpub.epa.gov/ecotox/), DSL (https://www.ec.gc.ca/lcpe-cepa/default.asp?n=5F213FA8-1)和QSAR Toolbox (http://oasis-lmc.org/products/software/toolbox.aspx))中,搜集了有机化学品鱼体BCF和BMF的实测值,经过去重和去掉无机盐的数据预处理,共获得BCF实测值1 381条,BMF实测值204条。其中,同时具有BCF和BMF实测值的有机化学品共计52种,包括烷烃、烯烃、稠环芳烃、有机氯化合物和全氟化合物等(结构如图1所示)。所得BCF和BMF值通过以10为底的对数转换为logBCF和logBMF,作为预测终点。所建数据集以4∶1的比例,划分训练集(41个化合物)和验证集(11个化合物)。

图1 同时具有生物富集因子(BCF)和生物放大因子(BMF)实测值的52种有机化学品的分子结构
Fig. 1 Molecular structures of 52 organic chemicals that possess experimentally determined values for both bioconcentration factors (BCF) and biomagnification factors (BMF)
不同分子表征方法对于模型预测效果影响显著[24],因此本研究选取了5种分子结构表征方法,包括Dragon描述符和4种分子指纹(表1)。具体而言,使用Dragon 6.0软件[30]计算得到了4 885种不同类型的分子结构描述符。所计算的Dragon描述符进行了如下处理:去掉常数与近似常数项,去掉成对相关系数≥0.95的描述符,以及至少有一个缺失值的描述符,采用标准差法进行标准化处理[31]。此外,使用开源的化学信息学Python软件包——RDKit (version 2021.9.3, www.rdkit.org)计算得到4种分子指纹(MACCS, AFP, ECFP, RDKFP)。
表1 Dragon描述符和分子指纹的名称、缩写及维度
Table 1 Names, abbreviations and dimensions of Dragon descriptors and molecular fingerprints
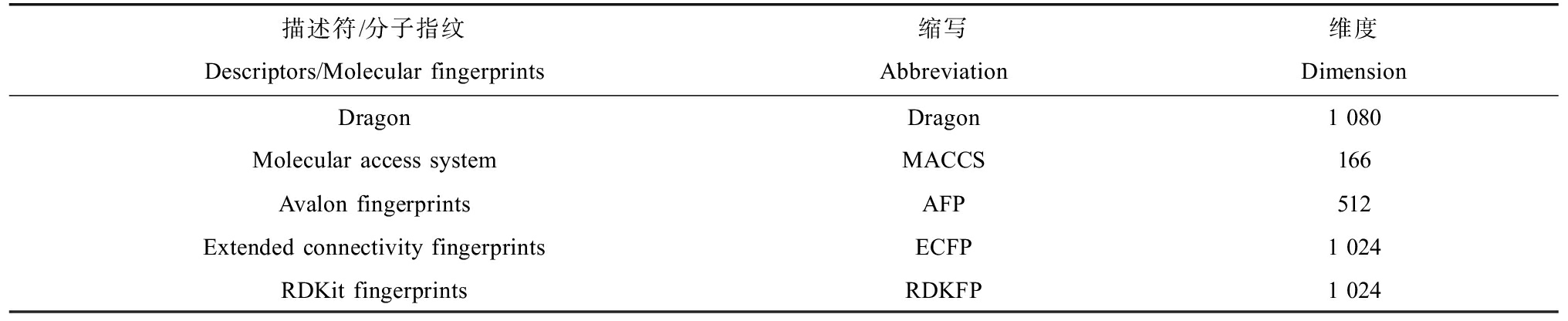
描述符/分子指纹Descriptors/Molecular fingerprints缩写Abbreviation维度DimensionDragonDragon1 080Molecular access systemMACCS166Avalon fingerprintsAFP512Extended connectivity fingerprintsECFP1 024RDKit fingerprintsRDKFP1 024
首先基于反向传播(back-propagation, BP)神经网络机器学习算法,建立了单任务模型,使用Python 3.7软件实现。BP神经网络是目前应用最广泛的神经网络算法,属于一种按照误差逆传播算法训练的多层前馈网络。BP神经网络包含输入层、隐藏层和输出层,该网络的主要特点是信号前向传递,误差反向传播[32]。考虑到神经网络算法本身可通过调整权重自动提取对当前任务有用的特征,故本研究中未对描述符进行进一步筛选。
本研究共采用5种分子结构的表征方式(表1),分别作为模型输入,构建了5个BCF的单任务(BCF-ST)模型以及5个BMF的单任务(BMF-ST)模型。基于训练集化合物的logBCF或logBMF实验值进行模型训练,采用验证集化合物进行模型验证。一般对于数据量较少的任务,选取2层隐藏层即可满足需要。因此,本研究规定神经网络的隐藏层数目为2。模型迭代次数(Epoch)设为300,以均方误差(MSE)作为损失函数。采用Python程序包hyperopt (0.1.2, https://github.com/hyperopt)对模型超参数进行了优化,包括各隐藏层神经元个数(NNC_i,其中i表示第i层隐藏层)、学习率、激活函数、隐藏层单元随机失活的概率(Dropout率)、批处理量和权重初始化参数等。优化后的超参数见表2。选取10折交叉验证系数![]() 最大的模型作为最优模型。
最大的模型作为最优模型。
表2 化学品生物富集因子(BCF)及生物放大因子(BMF)单任务及多任务预测模型的最优超参数
Table 2 Optimal hyperparameters of single-task and multi-task models on prediction of bioconcentration factors (BCF) and biomagnification factors (BMF) for chemicals

DragonMACCSAFPECFPRDKFP生物富集因子的单任务(BCF-ST)预测模型 Single-task prediction models on bioconcentration factorNNC_13010504020NNC_23030101010学习率 Learning rate0.010.010.010.010.01激活函数 Activation functionTanhEluReluReluEluDropout率 Dropout rate0.20.30.30.30.3批处理量 Batch size1616161616权重初始化参数Weight initialization parametershe_uniformhe_uniformrandom_uniformhe_uniformhe_uniform生物放大因子的单任务(BMF-ST)预测模型 Single-task prediction models on biomagnification factorNNC_13010504020NNC_23030101010学习率 Learning rate0.010.010.010.010.01激活函数 Activation functionTanhEluReluReluEluDropout率 Dropout rate0.20.30.30.30.3批处理量 Batch size1616161616权重初始化参数Weight initialization parametershe_uniformhe_uniformrandom_uniformhe_uniformhe_uniform多任务(MT)预测模型 Multi-task prediction modelsNNC_12090102030NNC_21030101010学习率 Learning rate0.010.010.010.010.01激活函数 Activation functionTanhTanhReluReluEluDropout率 Dropout rate0.20.20.30.30.2批处理量 Batch size1616161616权重初始化参数Weight initialization parametersglorot_uniformglorot_uniformrandom_uniformglorot_uniformhe_uniform
注:NNC_1和NNC_2分别表示第一层隐藏层和第二层隐藏层的神经元个数。
Note: NNC_1 and NNC_2 stand for the number of neurons in the first and the second hidden layers, respectively.
使用表1中的描述符或分子指纹作为输入,建立了同时预测logBCF和logBMF的多任务(MT)模型,模型框架见图2,即以Dragon描述符或分子指纹等多维度特征作为输入,在隐藏层中同时训练logBCF和logBMF,最终输出这2个终点的预测值。MT模型采用基于隐藏层参数的硬共享机制,即不同任务之间共享底层特征而保留任务特异性的高层特征,也就是说共享的底层参数如激活函数、Dropout率等,是完全相同的[20-22]。所构建的BCF和BMF多任务模型,本质上属于定量构效关系(QSAR)模型,根据QSAR原理,BCF和BMF的大小均取决于分子结构特征。同一组分子特征参数(包括描述符和分子指纹)可以较为详尽地表征分子结构特征,本文采用的描述符或分子指纹维度范围为166~1 080维,其中包含了与BCF和BMF存在关联的特征。因此,通过MTL挖掘出与BCF和BMF相关的特征,可实现不同环境行为参数的同步预测。此外,考虑到不同描述符包含的分子信息有交叉,以多种描述符作为输入建模会引入噪声,因此本研究选取同一类描述符作为输入,构建了单输入-多输出的MT模型。MT模型的构建采用BP神经网络机器学习算法。神经网络算法是目前构建MT模型最常使用的机器学习方法之一,已被成功用于预测化学品的理化性质和急性毒性[24-25]。采用2层隐藏层和300次迭代进行训练,且损失函数表示为2个终点的MSE之和。其他的超参数,也采用hyperopt软件包进行了优化,优化后的超参数见表2。

图2 多任务模型框架示意图
Fig. 2 Diagram of multi-task prediction models
采用训练集的决定系数![]() 均方根误差(RMSEtra)和平均绝对误差(MAEtra)评价模型的拟合效果,基于验证集的决定系数
均方根误差(RMSEtra)和平均绝对误差(MAEtra)评价模型的拟合效果,基于验证集的决定系数![]() 均方根误差(RMSEext)和平均绝对误差(MAEext)评价模型的预测能力。应用训练集的十折交叉验证(10-fold cross validation)系数
均方根误差(RMSEext)和平均绝对误差(MAEext)评价模型的预测能力。应用训练集的十折交叉验证(10-fold cross validation)系数![]() 评价模型的稳健性,即将数据集划分为10个大小相似的互斥子集,经10次建模计算得到的测试结果的均值作为
评价模型的稳健性,即将数据集划分为10个大小相似的互斥子集,经10次建模计算得到的测试结果的均值作为![]() 交叉验证方法常被用于评价模型的泛化能力及避免过拟合。上述指标的计算方法及公式可参考相关文献[33]。
交叉验证方法常被用于评价模型的泛化能力及避免过拟合。上述指标的计算方法及公式可参考相关文献[33]。
采用验证集与训练集分子间的谷本相似度(Tanimoto similarity)表征模型应用域[34-35]。具体而言,使用RDKit包生成训练集和验证集中化合物的MACCS分子指纹,对每个验证集分子A,计算其与训练集分子B的谷本相似度,计算公式如下:

SAB是分子A和B的谷本相似度,n是指纹的特征位数,XjA和XjB分别是分子A和分子B的第j个指纹特征。定义相似度阈值(Scutoff)和最少相似分子数量(Nmin),若SAB大于Scutoff的化合物数目超过Nmin值,则判定验证集分子B位于应用域内[34-35]。设置Scutoff值范围为0.25~0.95,每隔0.1取值;由于本研究数据集中化学品数量较少,因此Nmin值设定为1[34-35]。
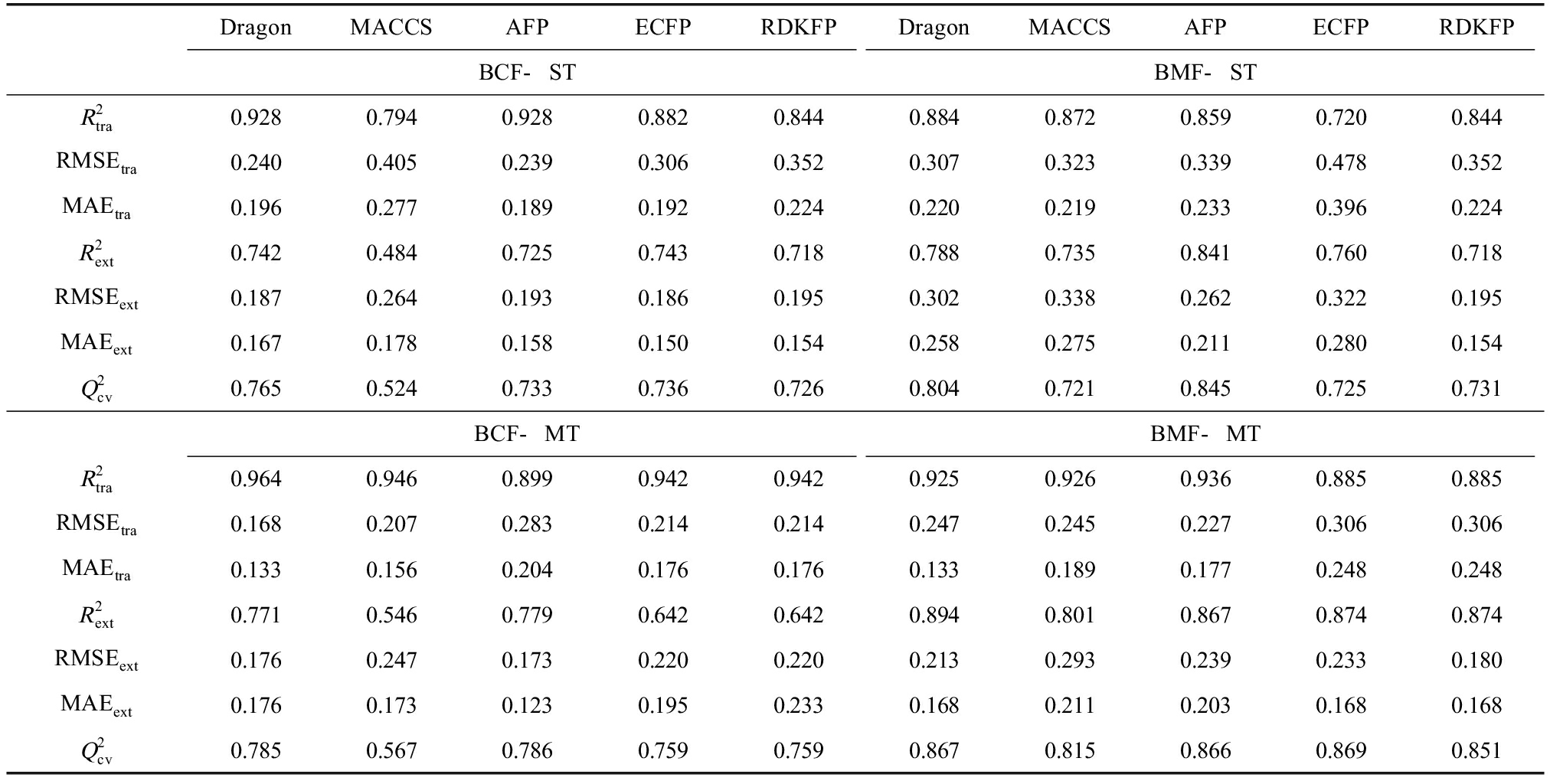
BCF-ST和BMF-ST的模型表现如表3所示。对于BCF-ST模型,当以Dragon描述符及分子指纹AFP为模型输入时,![]() 最高(均为0.928)。但基于Dragon描述符模型的
最高(均为0.928)。但基于Dragon描述符模型的![]() 和
和![]() 略高于分子指纹
略高于分子指纹![]() 和
和![]() 分别为0.725和0.733),二者在训练集和验证集上的预测误差RMSE和MAE相当,表明基于分子Dragon描述符的BCF-ST模型有较好的拟合效果、预测能力和稳健性。采用其他3种分子指纹,即MACCS, ECFP和RDKFP构建的模型,
分别为0.725和0.733),二者在训练集和验证集上的预测误差RMSE和MAE相当,表明基于分子Dragon描述符的BCF-ST模型有较好的拟合效果、预测能力和稳健性。采用其他3种分子指纹,即MACCS, ECFP和RDKFP构建的模型,![]() 均在0.9以下,其他评价指标也多劣于采用Dragon描述符及分子指纹AFP构建的BCF-ST模型。综上,基于分子Dragon描述符构建的BCF-ST模型,拟合优度和预测效果最好。
均在0.9以下,其他评价指标也多劣于采用Dragon描述符及分子指纹AFP构建的BCF-ST模型。综上,基于分子Dragon描述符构建的BCF-ST模型,拟合优度和预测效果最好。
表3 化学品生物富集因子及生物放大因子的单任务和多任务模型统计学指标对比
Table 3 Comparison of statistical parameters for single-task and multi-task models on prediction of bioconcentration factors and biomagnification factors of chemicals
DragonMACCSAFPECFPRDKFPDragonMACCSAFPECFPRDKFPBCF-STBMF-STR2tra0.9280.7940.9280.8820.8440.8840.8720.8590.7200.844RMSEtra0.2400.4050.2390.3060.3520.3070.3230.3390.4780.352MAEtra0.1960.2770.1890.1920.2240.2200.2190.2330.3960.224R2ext0.7420.4840.7250.7430.7180.7880.7350.8410.7600.718RMSEext0.1870.2640.1930.1860.1950.3020.3380.2620.3220.195MAEext0.1670.1780.1580.1500.1540.2580.2750.2110.2800.154Q2cv0.7650.5240.7330.7360.7260.8040.7210.8450.7250.731BCF-MTBMF-MTR2tra0.9640.9460.8990.9420.9420.9250.9260.9360.8850.885RMSEtra0.1680.2070.2830.2140.2140.2470.2450.2270.3060.306MAEtra0.1330.1560.2040.1760.1760.1330.1890.1770.2480.248R2ext0.7710.5460.7790.6420.6420.8940.8010.8670.8740.874RMSEext0.1760.2470.1730.2200.2200.2130.2930.2390.2330.180MAEext0.1760.1730.1230.1950.2330.1680.2110.2030.1680.168Q2cv0.7850.5670.7860.7590.7590.8670.8150.8660.8690.851
注:BCF-ST和BMF-ST分别表示生物富集因子的单任务模型和生物放大因子的单任务模型;BCF-MT和BMF-MT分别表示多任务模型预测生物富集因子和生物放大因子;![]() 和MAEtra分别表示训练集的决定系数、均方根误差和平均绝对误差;
和MAEtra分别表示训练集的决定系数、均方根误差和平均绝对误差;![]() 和MAEext分别表示验证集的决定系数、均方根误差和平均绝对误差;
和MAEext分别表示验证集的决定系数、均方根误差和平均绝对误差;![]() 表示10折交叉验证系数。
表示10折交叉验证系数。
Note:BCF-ST and BMF-ST stand for the single-task models on prediction of bioconcentration factor and biomagnification factor, respectively; BCF-MT and BMF-MT stand for the multi-task models on prediction of bioconcentration factor and biomagnification factor, respectively; ![]() RMSEtra, and MAEtra stand for the determination coefficient, root mean square error, and mean absolute error for the training set, respectively;
RMSEtra, and MAEtra stand for the determination coefficient, root mean square error, and mean absolute error for the training set, respectively; ![]() RMSEext, and MAEext stand for the determination coefficient, root mean square error, and mean absolute error for the validation set, respectively;
RMSEext, and MAEext stand for the determination coefficient, root mean square error, and mean absolute error for the validation set, respectively; ![]() standards for the 10-fold cross validation coefficient.
standards for the 10-fold cross validation coefficient.
对BMF-ST模型而言,基于Dragon描述符构建的模型![]() 最高(0.884)。采用其他4种分子指纹构建的模型,
最高(0.884)。采用其他4种分子指纹构建的模型,![]() 按照由大到小的顺序为MACCS(0.872)>AFP(0.859)>RDKFP(0.844)>ECFP(0.720)。然而,从验证集预测的效果看,以分子指纹AFP为输入时所建模型
按照由大到小的顺序为MACCS(0.872)>AFP(0.859)>RDKFP(0.844)>ECFP(0.720)。然而,从验证集预测的效果看,以分子指纹AFP为输入时所建模型![]() 最高,为0.841,其次是采用Dragon描述符建立的模型
最高,为0.841,其次是采用Dragon描述符建立的模型![]() 总体上,采用Dragon描述符所构建的BMF-ST模型,拟合效果较好。预测能力和稳健性方面,基于分子指纹AFP的BMF-ST模型效果,略优于基于Dragon描述符的模型。
总体上,采用Dragon描述符所构建的BMF-ST模型,拟合效果较好。预测能力和稳健性方面,基于分子指纹AFP的BMF-ST模型效果,略优于基于Dragon描述符的模型。
MT模型(BCF-MT和BMF-MT)的结果见表3。整体上,不同分子结构表征方式作为模型输入时,MT模型的拟合效果、预测能力和稳健性均强于ST模型。基于Dragon描述符的MT模型性能最佳,具体表现为针对预测终点![]() 和
和![]() 分别为0.964和0.771;针对预测终点
分别为0.964和0.771;针对预测终点![]() 和
和![]() 分别为0.925和0.894,预测误差RMSEext和MAEext值均较低(0.133~0.247)。所建MT模型的
分别为0.925和0.894,预测误差RMSEext和MAEext值均较低(0.133~0.247)。所建MT模型的![]() 值均较高(0.785~0.867),表明模型具有较好的泛化能力。当预测BCF和BMF时,采用Dragon描述符作为模型输入更适合。最优MT模型的预测值和实测值的关系见图3,可见模型的预测值与实测值间相关性良好,无明显离群点,预测误差均处于1个log单位之内。
值均较高(0.785~0.867),表明模型具有较好的泛化能力。当预测BCF和BMF时,采用Dragon描述符作为模型输入更适合。最优MT模型的预测值和实测值的关系见图3,可见模型的预测值与实测值间相关性良好,无明显离群点,预测误差均处于1个log单位之内。
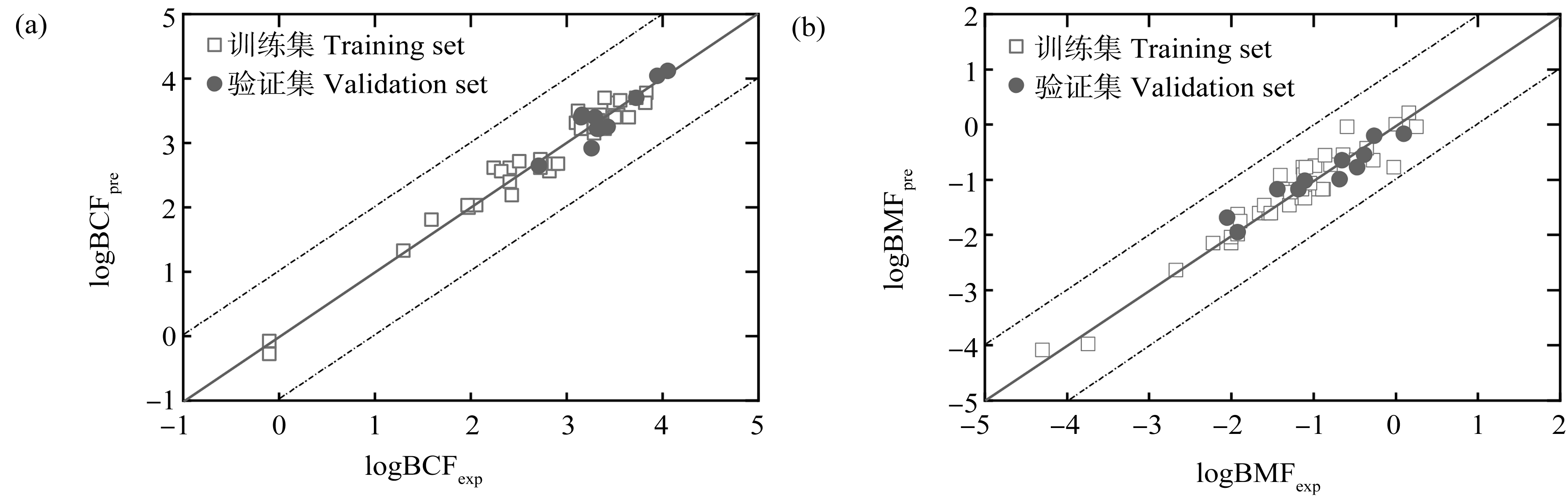
图3 有机化学品生物富集因子(a)和生物放大因子(b)的实测值与基于Dragon描述符的多任务模型的预测值的比较
注:BCF和BMF分别表示生物富集因子和生物放大因子,logBCFpre, logBCFexp, logBMFpre和logBMFexp分别表示logBCF的预测值、logBCF的实测值、logBMF的预测值和logBMF的实测值。
Fig. 3 Comparison of experimental bioconcentration factor (a) and biomagnification factor (b) of organic chemicals and predicted values obtained using multi-task model based on Dragon descriptors
Note: BCF and BMF stand for the bioconcentration factor and biomagnification factor, respectively; logBCFpre, logBCFexp, logBMFpre, and logBMFexp represent the predicted logBCF, experimental logBCF, predicted logBMF, and experimental logBMF, respectively.
研究考察了不同相似度阈值(Scutoff)对模型应用域及模型效果的影响,结果见图4。当Scutoff取不同值时,针对BCF和BMF,所建MT模型的拟合优度![]() 呈现相同的变化趋势。当Scutoff介于0.25至0.45时,
呈现相同的变化趋势。当Scutoff介于0.25至0.45时,![]() 值最高,为0.813~0.839;当Scutoff为0.55和0.65时,
值最高,为0.813~0.839;当Scutoff为0.55和0.65时,![]() 值为0.791~0.796;当Scutoff为0.75和0.85时,
值为0.791~0.796;当Scutoff为0.75和0.85时,![]() 值最低,为0.640~0.693;当Scutoff为0.95时,
值最低,为0.640~0.693;当Scutoff为0.95时,![]() 值为0.728~0.750。总体上,随着Scutoff值的增加(0.25至0.85),
值为0.728~0.750。总体上,随着Scutoff值的增加(0.25至0.85),![]() 逐渐减小;当Scutoff值增加至0.95时,
逐渐减小;当Scutoff值增加至0.95时,![]() 略有上升。
略有上升。
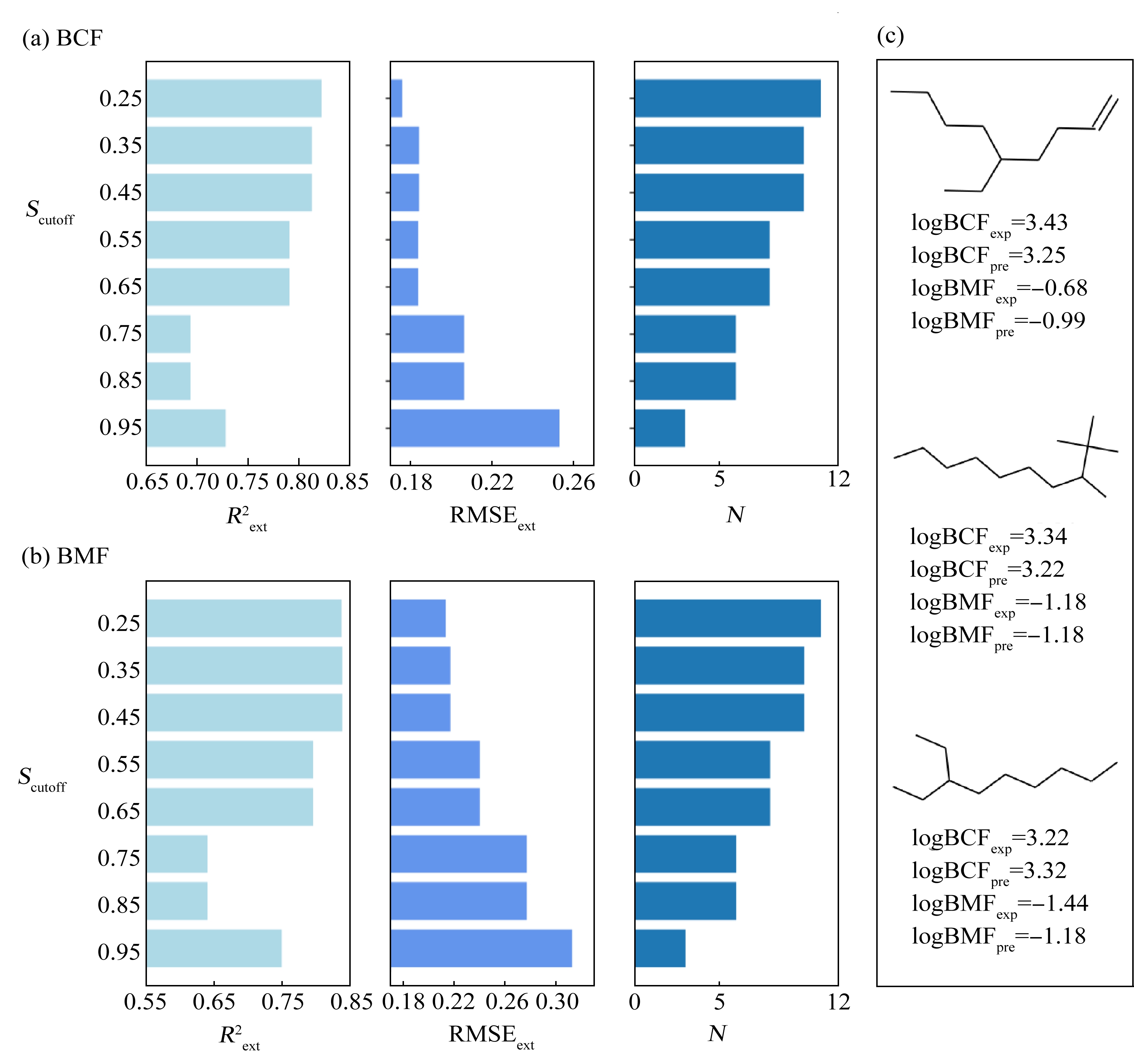
图4 不同相似度阈值(Scutoff)值对多任务模型应用域及预测效果的影响
注:(a) 生物富集因子;(b) 生物放大因子;(c) Scutoff值从0.85变化至0.95时,从应用域中剔除的化合物的结构及对应logBCF和logBMF的预测和实测值;Scutoff表示相似度阈值;![]() 和RMSEtra分别表示外部验证集的决定系数和均方根误差;N表示在应用域中的化合物的个数。
和RMSEtra分别表示外部验证集的决定系数和均方根误差;N表示在应用域中的化合物的个数。
Fig. 4 Effects of different similarity threshold (Scutoff) values on the application domains and predictive performances of multi-task models
Note: (a) Bioconcentration factor; (b) Biomagnification factor; (c) Molecular structures and corresponding predicted and experimental logBCF and logBMF values of chemicals deleted from the application domain when Scutoff value varied from 0.85 to 0.95; Scutoff represents the similarity threshold; ![]() and RMSEext stand for the determination coefficient, and root mean square error for the validation set, respectively; N represents the number of chemicals in the application domain.
and RMSEext stand for the determination coefficient, and root mean square error for the validation set, respectively; N represents the number of chemicals in the application domain.
本研究基于BP神经网络,以Dragon描述符和4种分子指纹作为模型输入,针对有机化学品的鱼体BCF和BMF,分别建立了ST和MT预测模型。MT模型的拟合效果、稳健性和预测能力,均强于ST模型,表明MT模型可以考虑不同任务间的关联性,通过学习挖掘关联信息,从而提升了模型的预测性能[21-23]。前人采用MT模型,预测了化学品的毒性效应,也发现MT模型的预测效果优于ST模型[25]。本研究也是首次将MT模型应用于多种生物积累参数的预测。今后的研究中,当预测相互关联的多个终点时,建议考虑采用MTL建模策略,在简化建模流程、减少计算量的同时,提升模型的预测效果。
基于不同化学品分子表征方法建立的模型,拟合效果、预测能力和稳健性略有差异。其中,基于分子指纹MACCS的模型出现过拟合现象,即![]() 远高于
远高于![]() 的值。导致上述现象的原因可能是,MACCS仅有166维,分别表示化合物分子中是否存在某一特定结构片段,这种表示方法可能会丢失大量分子结构信息,从而导致模型效果不佳。前人基于MACCS指纹,应用极端梯度提升树(XGBoost)算法构建致癌性预测模型时,也发现模型效果弱于基于分子指纹ECFP和RDKFP的模型[22]。
的值。导致上述现象的原因可能是,MACCS仅有166维,分别表示化合物分子中是否存在某一特定结构片段,这种表示方法可能会丢失大量分子结构信息,从而导致模型效果不佳。前人基于MACCS指纹,应用极端梯度提升树(XGBoost)算法构建致癌性预测模型时,也发现模型效果弱于基于分子指纹ECFP和RDKFP的模型[22]。
应用域表征的结果表明,模型![]() 值随Scutoff值的升高而降低,但当Scutoff值从0.85升高至0.95时,
值随Scutoff值的升高而降低,但当Scutoff值从0.85升高至0.95时,![]() 值升高,即从应用域中剔除的3个化学物质(结构见图4(c)),对于
值升高,即从应用域中剔除的3个化学物质(结构见图4(c)),对于![]() 值影响显著。可见,所剔除的物质为长链烷烃或烯烃类物质,结构简单,而本研究数据集中涉及的大多数目标化合物具有苯环结构或者杂原子(图1),与这3个化合物的结构相差较大。因此,模型训练过程可能未充分捕捉这几种化合物的结构信息,导致对其预测效果欠佳。
值影响显著。可见,所剔除的物质为长链烷烃或烯烃类物质,结构简单,而本研究数据集中涉及的大多数目标化合物具有苯环结构或者杂原子(图1),与这3个化合物的结构相差较大。因此,模型训练过程可能未充分捕捉这几种化合物的结构信息,导致对其预测效果欠佳。
由于此前研究尚未见有BCF和BMF的MT模型报道,现有以logBCF或logBMF为预测终点的ST模型,与本研究MT模型采用的数据集相差较大,因此未将本模型与现有ST模型进行对比。本研究搜集到具有BCF实测值的化合物1 381个,具有BMF实测值的化合物204个,其中同时具有BCF和BMF值的化合物共52个,主要受限于化学品BMF的数据量。考虑到摄食暴露也是化学品进入生物体的重要途径[36-37],今后的研究有必要进一步发展化学品BMF值的高通量预测模型。最后,本研究所建模型仅覆盖了有机氯化合物、稠环芳烃、烷烃、烯烃和全氟化合物等化学物质,种类依然有限,因此有必要扩充建模的数据集,获得更多化学品的BMF, BCF数据,以期获得应用域更广、预测准确性更高的模型。考虑到通过实验测试补充化学品BMF数据相对繁琐,测试成本和时间消耗较大,可以进一步优化MTL建模策略,比如针对数据量不平衡的多个终点构建MT预测模型,来克服化学品生物积累性数据缺失的瓶颈。
本研究构建的MT模型,可用于有机化学品生物积累参数的预测,为后续研究生物积累参数的MT模型奠定基础,有望为化学品生物积累性评价提供技术支持。
[1] Chen D, Kannan K, Tan H L, et al. Bisphenol analogues other than BPA: Environmental occurrence, human exposure, and toxicity—A review [J]. Environmental Science &Technology, 2016, 50(11): 5438-5453
[2] Liu R Z, Mabury S A. Synthetic phenolic antioxidants in personal care products in Toronto, Canada: Occurrence, human exposure, and discharge via greywater [J]. Environmental Science &Technology, 2019, 53(22): 13440-13448
[3] Provencher J F, Malaisé F, Mallory M L, et al. 44-year retrospective analysis of ultraviolet absorbents and industrial antioxidants in seabird eggs from the Canadian Arctic (1975 to 2019) [J]. Environmental Science &Technology, 2022, 56(20): 14562-14573
[4] Li Y N, Yao J Z, Zhang J, et al. First report on the bioaccumulation and trophic transfer of perfluoroalkyl ether carboxylic acids in estuarine food web [J]. Environmental Science &Technology, 2022, 56(10): 6046-6055
[5] Gaballah S, Swank A, Sobus J R, et al. Evaluation of developmental toxicity, developmental neurotoxicity, and tissue dose in zebrafish exposed to GenX and other PFAS [J]. Environmental Health Perspectives, 2020, 128(4): 47005
[6] Zhang T T, Zhou X, Xu A M, et al. Toxicity of polybrominated diphenyl ethers (PBDEs) on rodent male reproductive system: A systematic review and meta-analysis of randomized control studies [J]. The Science of the Total Environment, 2020, 720: 137419
[7] Li F, Li X H, Shao J P, et al. Estrogenic activity of anthraquinone derivatives: in vitro and in silico studies [J]. Chemical Research in Toxicology, 2010, 23(8): 1349-1355
[8] Luo T L, Chen J W, Song B, et al. Time-gated luminescence imaging of singlet oxygen photoinduced by fluoroquinolones and functionalized graphenes in Daphnia magna [J]. Aquatic Toxicology, 2017, 191: 105-112
[9] Rockström J, Steffen W, Noone K, et al. A safe operating space for humanity [J]. Nature, 2009, 461(7263): 472-475
[10] 陈景文, 全燮. 环境化学[M]. 大连: 大连理工大学出版社, 2009: 170-176
[11] European Union. Regulation (EC) No. 1907/2006 of the European Parliament and of the Council of 18 December 2006, concerning the Registration, Evaluation, Authorization, and Restriction of Chemicals (REACH) [R]. Brussels: European Union, 2006
[12] 中华人民共和国生态环境部. 新化学物质环境管理登记指南[R]. 北京: 中华人民共和国生态环境部, 2020
[13] Organisation for Economic Co-operation and Development (OECD). OECD guidelines for the testing of chemicals, Test No. 305: Bioaccumulation in fish: Aqueous and dietary exposure [R]. Paris: OECD, 2012
[14] Zhao C Y, Boriani E, Chana A, et al. A new hybrid system of QSAR models for predicting bioconcentration factors (BCF) [J]. Chemosphere, 2008, 73(11): 1701-1707
[15] Dearden J C, Hewitt M. QSAR modelling of bioconcentration factor using hydrophobicity, hydrogen bonding and topological descriptors [J]. SAR and QSAR in Environmental Research, 2010, 21(7-8): 671-680
[16] Strempel S, Nendza M, Scheringer M, et al. Using conditional inference trees and random forests to predict the bioaccumulation potential of organic chemicals [J]. Environmental Toxicology and Chemistry, 2013, 32(5): 1187-1195
[17] 郑玉婷, 乔显亮, 于洋, 等. 有机化学品生物富集因子定量结构-活性关系模型[J]. 生态毒理学报, 2019, 14(2): 214-221
Zheng Y T, Qiao X L, Yu Y, et al. Quantitative structure-activity relationship model for bioconcentration factors of organic chemicals [J]. Asian Journal of Ecotoxicology, 2019, 14(2): 214-221 (in Chinese)
[18] 丁蕊, 陈景文, 于洋, 等. 基于集成学习算法构建有机化学品鱼体生物富集因子的QSAR预测模型[J]. 环境化学, 2021, 40(5): 1295-1304
Ding R, Chen J W, Yu Y, et al. Using ensemble learning algorithms to develop QSAR models on bioconcentration factors of organic chemicals in multispecies fish [J]. Environmental Chemistry, 2021, 40(5): 1295-1304 (in Chinese)
[19] Fatemi M H, Abraham M H, Haghdadi M. Prediction of biomagnification factors for some organochlorine compounds using linear free energy relationship parameters and artificial neural networks [J]. SAR and QSAR in Environmental Research, 2009, 20(5-6): 453-465
[20] Caruana R. Multitask learning [J]. Machine Learning, 1997, 28(1): 41-75
[21] Muratov E N, Bajorath J, Sheridan R P, et al. QSAR without borders [J]. Chemical Society Reviews, 2020, 49(11): 3525-3564
[22] Wu Z X, Jiang D J, Wang J K, et al. Mining toxicity information from large amounts of toxicity data [J]. Journal of Medicinal Chemistry, 2021, 64(10): 6924-6936
[23] Zhang Y, Yang Q. An overview of multi-task learning [J]. National Science Review, 2018, 5(1): 30-43
[24] Wu K D, Zhao Z X, Wang R X, et al. TopP-S: Persistent homology-based multi-task deep neural networks for simultaneous predictions of partition coefficient and aqueous solubility [J]. Journal of Computational Chemistry, 2018, 39(20): 1444-1454
[25] Wu K D, Wei G W. Quantitative toxicity prediction using topology based multitask deep neural networks [J]. Journal of Chemical Information and Modeling, 2018, 58(2): 520-531
[26] Arnot J, Gobas F. A review of bioconcentration factor (BCF) and bioaccumulation factor (BAF) assessments for organic chemicals in aquatic organisms [J]. Environmental Reviews, 2006, 14(4): 257-297
[27] Arnot J A, Quinn C L. Development and evaluation of a database of dietary bioaccumulation test data for organic chemicals in fish [J]. Environmental Science &Technology, 2015, 49(8): 4783-4796
[28] Grisoni F, Consonni V, Vighi M. Acceptable-by-design QSARs to predict the dietary biomagnification of organic chemicals in fish [J]. Integrated Environmental Assessment and Management, 2019, 15(1): 51-63
[29] Mansouri K, Consonni V, Durjava M K, et al. Assessing bioaccumulation of polybrominated diphenyl ethers for aquatic species by QSAR modeling [J]. Chemosphere, 2012, 89(4): 433-444
[30] Talete S R L. DRAGON (Software for Molecular Descriptor Calculation) Version 6.0 [CP].Italy: TALETE SRL, 2012
[31] Bikesh K, Kesari V, S Thoke A. Investigations on impact of feature normalization techniques on classifier’s performance in breast tumor classification [J]. International Journal of Computer Applications, 2015, 116(19): 11-15
[32] Rumelhart D E, Hinton G E, Williams R J. Learning representations by back-propagating errors [J]. Nature, 1986, 323(6088): 533-536
[33] 覃礼堂, 刘树深, 肖乾芬, 等. QSAR模型内部和外部验证方法综述[J]. 环境化学, 2013, 32(7): 1205-1211
Qin L T, Liu S S, Xiao Q F, et al. Internal and external validations of QSAR model: Review [J]. Environmental Chemistry, 2013, 32(7): 1205-1211 (in Chinese)
[34] Wang Z Y, Chen J W, Hong H X. Applicability domains enhance application of PPARγ agonist classifiers trained by drug-like compounds to environmental chemicals [J]. Chemical Research in Toxicology, 2020, 33(6): 1382-1388
[35] Wang Z Y, Chen J W, Hong H X. Developing QSAR models with defined applicability domains on PPARγ binding affinity using large data sets and machine learning algorithms [J]. Environmental Science &Technology, 2021, 55(10): 6857-6866
[36] Wang H T, Xia X H, Wang Z X, et al. Contribution of dietary uptake to PAH bioaccumulation in a simplified pelagic food chain: Modeling the influences of continuous vs intermittent feeding in zooplankton and fish [J]. Environmental Science &Technology, 2021, 55(3): 1930-1940
[37] Wang H T, Xia X H, Liu R, et al. Multicompartmental toxicokinetic modeling of discrete dietary and continuous waterborne uptake of two polycyclic aromatic hydrocarbons by zebrafish Danio rerio [J]. Environmental Science &Technology, 2020, 54(2): 1054-1065