研究表明,含内分泌干扰物(endocrine disruptor chemicals, EDCs)类的化学品在农业、工业和日常生活中被广泛使用[1],已在废水、地表水、自来水中陆续检出,表明其对水生生物乃至人类的影响正在逐渐扩大[2-5]。 EDCs 可以直接作用于内分泌系统,能够以阻断或模仿人类和动物体内自然激素的方式干扰激素行为,从而对心血管、代谢、免疫,尤其是生物的生殖系统造成影响,导致种群数量下降[6-9];大部分的EDCs 具有低剂量有效性、半衰期长和生物富集、生物放大等特点,因此会在环境中持久存在,造成较为长远的影响[10-12]。 研究数据表明,我国多处水域均受到EDCs 污染,由此带来的生态风险需要引起高度的重视[13-15]。
EDCs 生态风险的科学评估则依赖于繁殖毒性数据的获取。 EDCs 的繁殖毒性数据主要来自与生物的生活史或部分生活史相关的实验。 这些实验周期长、成本高,难以在短期内积累足够的EDCs 繁殖毒性数据,使得 EDCs 的生态风险评估非常困难[15-18],不利于以后科学开展生态风险评估和环境管理工作。 使用数学模型来预测毒性效应已成为国际生态毒理学研究热点[19]。 数学建模工具可以在一定的框架下对现有的毒性实验进行拓展,有利于深入了解剂量与反应关系之间的复杂性[15,20],从而保护生态系统,降低生态风险。 使用模型预测毒性效应数据相比实验获取也有一定的优势,如扩充实验数据、减少时间和物力消耗以及生物牺牲量[21-22],还可以对多种化学品的联合作用进行分析[23]等。
定量构效关系(quantitative structure-activity relationship, QSAR)模型被广泛应用于预测毒性效应。QSAR 是将一组化合物的某种性质或活性与这些化合物的化学成分或结构信息进行定量关联的方法,可以用来预测化合物的毒性值、作用模式,筛选和排序化学品等[24-26],该方法通常与其他模型方法如机器学习耦合使用;其中机器学习在生态毒理学中得到了越来越多的应用,其一般原理是根据一定的规则将输入变量与输出变量之间的关系一般化,并用于预测未知的相似情况[27-28];机器学习方法可以更好地处理非线性问题,对于关系复杂或未知的输入、输出变量也有很好的适应性,且通常具有良好的精度,可以减少重复性试验等[29-31]。 而 EDCs 繁殖毒性是慢性毒性的一种,急性毒性终点不适用于测量EDCs 的慢性繁殖毒性效应。 卵黄蛋白原(vitelloge-nin, VTG)、性腺指数(gonado-somatic index, GSI)、第二性征、血浆中的类固醇浓度和性腺组织病变被认为是用于评估EDCs 繁殖毒性终点的生物标志物,这些终点的变化需要长时间观测,一般采用无观察效应浓度(no observed effect concentration, NOEC)或最低可观察效应浓度(lowest observed effect concentration, LOEC)指标表示[32],这就造成了EDCs 毒性数据较少,从而鲜见利用上述模型对EDCs 水生生物繁殖毒性进行预测[21]。
因此本文将首先对近年来应用机器学习方法预测化合物水生生物毒性效应的相关研究进展进行总结,并在搜集到的可靠数据的基础上,利用QSAR建立用于预测EDCs 水生生物毒性效应的机器学习模型,从而为日后的化学品生态风险评估和检测优先性等提供指导。
1 材料与方法(Materials and methods)
1.1 机器学习预测化学物质水生生物毒性文献的检索与评述
通过Web of Science 和中国知网数据库对近年来国内外使用机器学习方法预测水生生物毒性文章进行检索,采用的检索词如表1 所示。 对检索到的文献作如下分析:当前研究的主要目的;文献中使用到的机器学习模型以及每种模型的使用频率;对每项研究涉及的不同研究对象进行汇总,如化合物、归属于不同营养级的水生生物以及毒性终点等;另外还包括文献内涉及到的研究手段与数据处理方法等。
表1 用于检索使用机器学习预测内分泌干扰物水生生物毒性效应文献的关键词
Table 1 Key words for searching papers that applied machine learning to predict the toxicity effects of endocrine disruptor chemicals on aquatic organisms
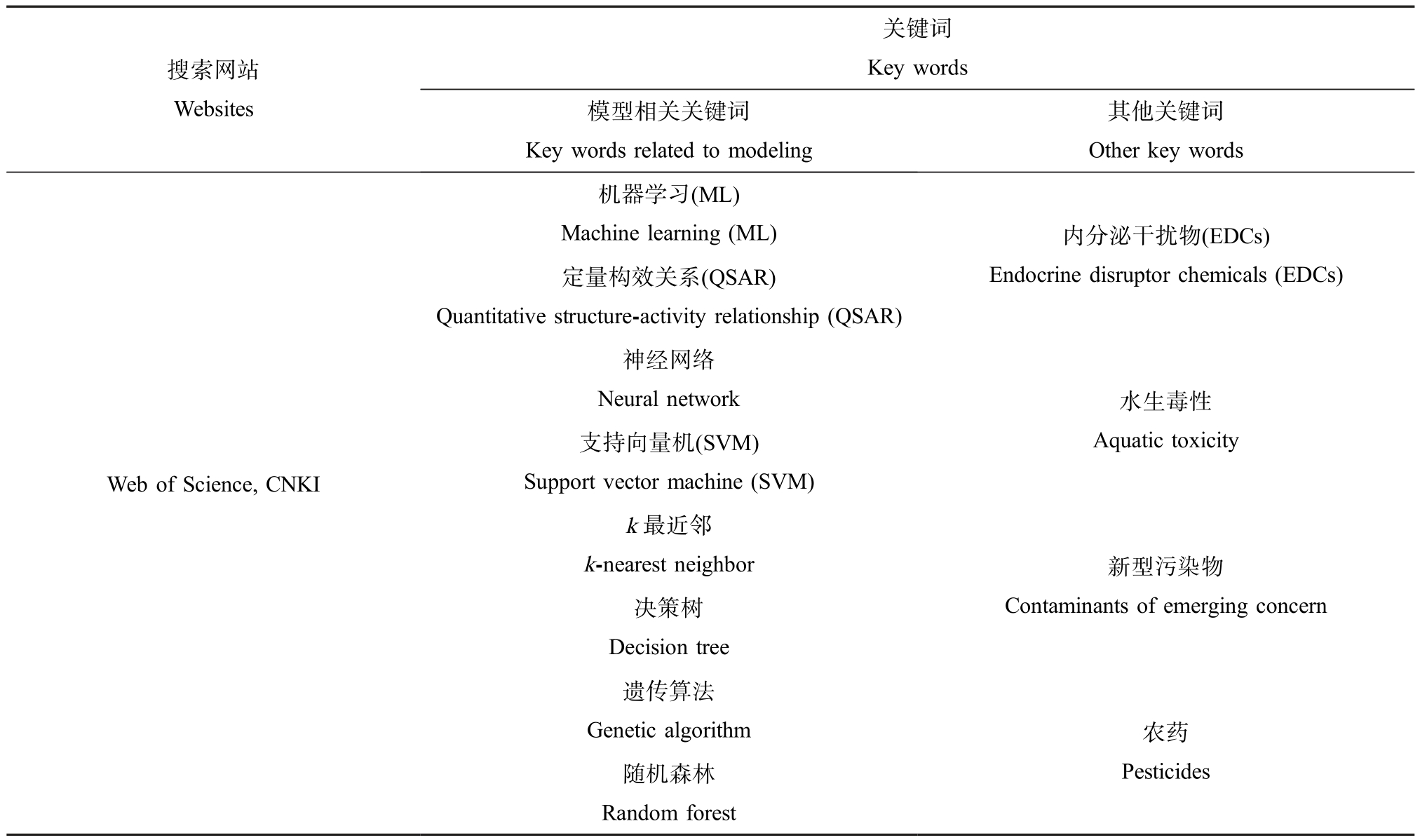
搜索网站Websites关键词Key words模型相关关键词Key words related to modeling其他关键词Other key words机器学习(ML)Machine learning (ML)内分泌干扰物(EDCs)Endocrine disruptor chemicals (EDCs)水生毒性Aquatic toxicity Web of Science, CNKI定量构效关系(QSAR)Quantitative structure-activity relationship (QSAR)神经网络Neural network支持向量机(SVM)Support vector machine (SVM)k 最近邻k-nearest neighbor决策树Decision tree遗传算法Genetic algorithm随机森林Random forest新型污染物Contaminants of emerging concern农药Pesticides
1.2 基于机器学习和QSAR 的EDCs 毒性预测的模型构建
1.2.1 数据获取与预处理
参考文献中描述的毒性数据筛选方法[33],在美国环境保护局(US EPA) ECOTOX 数据库检索了以NOEC、LOEC 等作为毒性终点,与黑头软口鲦(Pimephales promelas)繁殖毒性相关的数据。 若搜集所得数据集内的相同化学品在相同毒性终点上存在不同的数据点,则取几何平均值;筛选后得到了83种不同化学品对黑头软口鲦的繁殖毒性数据,考虑到数据量的因素,未对化学品继续筛选[34]。
分子描述符是一组将分子的不同属性(如物理化学、拓扑和结构等)进行量化表示的数值[35-36]。 为了获得分子描述符,首先需要收集不同化学物质对应的简化分子线性输入规范(simplified molecular input line entry specification, SMILES);SMILES 数据收集自PubChem 网站(https://pubchem.ncbi.nlm.nih.gov/);使用了 PaDEL-descriptor 软件[37]的 python 接口用于计算分子描述符,该软件可以根据SMILES为每种化合物计算出共1 875 种分子描述符。
在获得的描述符数据集中,并不是所有的描述符对于模型构建都是必要的。 具体筛选方法如下。
(1) 一些化合物的某些分子描述符的计算值可能为空值或无穷值(体现在excel 或csv 文件中即为无数据和Inf/Infinity),这些数值无法被输入至机器学习模型中用于训练,由于数据集中化合物的数量较少,因此删除了具有非法值的描述符[38]。
(2) 常数项或半常数项(该系列的80%及以上数值都相等)的描述符通常对模型的贡献较小,因此采取方差过滤法并选取0.01 作为过滤界限[39-40]。
(3) 一些分子描述符之间具有线性相关性,若成对的描述符之间的Pearson 相关系数>0.99,则只留下其中一个[34]。
(4) 经过上述筛选,大多数冗杂特征被去除,但仍需要选择最优子集。 这个选择过程被认为是比较困难的,因为没有合适的规则作为指导,通常以个人经验与其他算法相结合的方式进行[41-42]。 本文使用了递归特征消除(recursive feature elimination,RFE)[43],RFE 可以结合具有判断变量重要性的机器学习算法,重复建模为特征的重要性进行排序并逐渐删除指定个数特征,直到剩余规定数量的特征为止。 为了消除数据之间由于数量级差异带来的影响,首先对所有描述符作了标准化,公式如下所示:
式中:Xi 为第n个描述符的第i个数值,μn 为第n个描述符的平均值,Sn 为第n个描述符的标准差;然后使用结合随机森林的RFE 法选择最终特征子集。
了解化合物的可能毒性范围有利于开展初步生态风险评估工作[44]。 根据中华人民共和国国家标准《化学品水生环境危害分类指导第3 部分:水生毒性》(GB/T 36700.3—2018),对于慢性毒性不大于100 μg·L-1 的物质,认为其毒性较高,反之则认为其毒性较低;在此标准的指导下,选取了100 μg·L-1 作为分类界限,NOEC 小于等于该值的化合物为类别“1”,大于该值的为类别“0”。 数据集被以4∶1 的比例划分为训练集和测试集,测试集用于模型的效果评价,不用于模型的训练。
1.2.2 机器学习模型的构建
采用的支持向量机(support vector machine,SVM)模型与线性神经网络(linear neural network,LNN)模型,分别由 scikit-learn[45] 和 Keras 搭建。SVM 模型可以执行线性和非线性的分类与回归任务,且被认为非常适用于中小型数据集[46],其中应用到的核函数为高斯径向基(Gaussian radial basis function, RBF),该核函数常被应用于SVM 的构建中。LNN 模型中,每个神经元都代表一个多元线性函数,如下式所示。
式中:Y 为该神经元的输出值,X1 ~Xn 为输入特征,W1 ~Wn 为权重,b 为偏置值,采用了单隐藏层结构[47];Sigmoid 函数为激活函数,可以将输出的数值范围变为0 ~1,即“预测为正类”的概率值;二元交叉熵作为损失函数。
1.2.3 模型评估标准
在二元分类中,模型的预测性能根据真阳性(true positives, TP)、真阴性(true negatives, TN)、假阳性(false positives, FP)、假阴性(false negatives, FN)的数量以及敏感性(sensitivity, SE)、特异性(specificity,SP)和预测准确度(accuracy, Acc)来判定[44];此外还应用了受试者工作特征(receiver operating characteristic, ROC)曲线与曲线下面积(area under curve,AUC)来评价模型的分类性能;ROC 曲线的x 轴为假阳性率(false positive rate),y 轴为真阳性率(true positive rate);AUC 取值为 0.5 ~1.0,当 AUC=1.0 时表示这是一个完美的分类器,而AUC=0.5 时说明该分类器没有分类能力[48-49]。 所涉及到的评价参数的含义和计算式如表2 所示。
表2 二元分类模型能力判定标准
Table 2 Assessment standard of binary classification models

性能指标Performance indicators计算公式Formula正确预测为阳性的数量(TP)True positive (TP)正确预测为阴性的数量(TN)True negative (TN)错误预测为阳性的数量(FP)False positive (FP)错误预测为阴性的数量(FN)False negative (FN)敏感性(SE)Sensitivity (SE)TP TP+FN特异性(SP)Specificity (SP)TN TN+FP正确率(Acc)Accuracy (Acc)TP+TN TP+TN+FP+FN正确预测为阳性的比例Proportion that correctly predicted as positive TP TP+FN错误预测为阳性的比例Proportion that incorrectly predicted as positive FP FP+TN
1.2.4 应用领域
经济合作与发展组织关于QSAR 模型的指导文件[50]中指出,“一个(Q)SAR 模型需要定义其应用域(application domain, AD)”,即根据模型训练集中化学物质的结构或物理化学等信息确定模型的预测能力限制范围,对超出该范围的化学物质(与训练集中物质的相似性不足)的预测结果被认为可靠程度较低。 由于相似性有很多不同的表达方式(一般通过理化性质来定义),因此AD 的评估也可以是多样化的,如杠杆方法[51]和基于Euclidean 距离的AD 分析法[52-54]。 其中Euclidean 方法将化学分子表示为多维向量中的一点(维数等于每种描述符中的变量数量),并以Euclidean 距离计算任意2个分子之间的相 似 性。 Ambit Discovery 软 件 (http://ambit.sourceforge.net/download_ambitdiscovery.html)可以直接构建基于Euclidean 距离的AD 分析,并显示处于AD 之外的化合物,因此 AD 分析将使用该软件进行。
2 结果(Results)
2.1 机器学习预测化学物质毒性进展
根据检索词共筛选出英文文献61 篇,中文文献2 篇,发文数量与年份增长之间的关系如图1 所示。由图1 可知,结合机器学习方法来预测化合物对水生生物毒性的文章数量从2009年开始增多并且呈现明显的上升趋势,说明这种策略正得到越来越多的认可。 这一方面是由于机器学习方法所具备的优势,另一方面也和计算机技术的发展为机器学习的应用提供了更优秀的条件有关[55]。
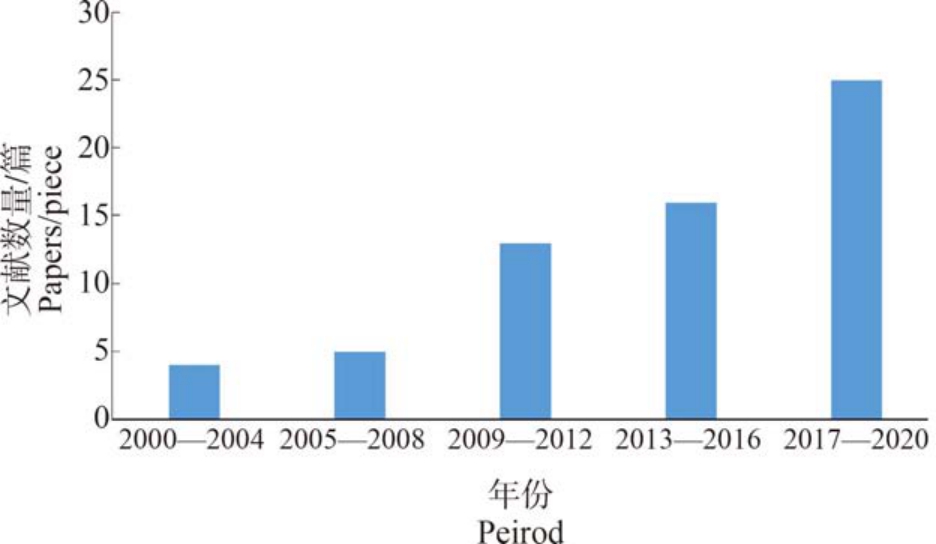
图1 近年来使用机器学习或建模方法预测化学品水生生物毒性的文章数量和趋势
Fig.1 The number and trend of papers that used machine learning or modeling methods to predict the toxicity of chemicals on aquatic organisms in recent years
每种算法的使用次数与应用方式(用于预测离散、连续型数据,或者变量筛选)如图2 所示。 其中,使用次数最多的是SVM,共25 次,且在回归与分类问题上的使用较为均衡,一定程度上体现了其广泛适用性[56-58];线性回归的使用次数仅次于SVM,并与神经网络一起更多地被应用于回归问题;遗传算法几乎仅被用于辅助作用,即作为一种选择描述符子集的手段,而不用于预测化合物的毒性效应;决策树、随机森林和k 最近邻等算法被较多地应用于分类问题[59-60]。

图2 被用于预测化学品水生生物毒性的算法及其应用的频率与目的
Fig.2 Algorithms used to predict the toxicity of chemicals on aquatic organisms and their frequency and purpose of application
文献中涉及的水生生物、化合物和毒性终点如图3 所示。 涉及的水生生物包括脊椎生物、无脊椎生物和藻类,其中脊椎生物即鱼类,如黑头软口鲦(Pimephales promelas)、斑马鱼(Brachydanio rerio)和虹鳟(Oncorhynchus mykiss)等;无脊椎生物中较多的是浮游生物,如梨形四膜虫(Tetrahymena pyriformis)、大型溞(Daphnia magna)等。 所探究的化合物种类也较多:按照结构信息,有取代苯类化合物、芳香族化合物和酚类化合物等;根据作用,包含农药(如生物杀灭剂、除草剂等)、个人护理产品(如抗抑郁药、降压药和麻醉药等)和工业化学品等。 根据危害方式,大多数文献所研究的毒性终点为急性毒性,如半抑制生长浓度[61]、半致死浓度[62]和半数效应浓度[63]等,这可能与其实验周期短、数据量较多、误差较低以及当前管控优先度较高等因素有关。 而在慢性毒性当中,以 NOEC 作为毒性终点的研究较少[34,64],且模型的性能也相对较差,如Sheffield 和Judson 等[34]的研究中为该终点构建了回归模型,评估回归模型常用的标准之一是由实际值与预测值所计算出的决定系数(R2),在其研究中所构建的部分模型的R2 为0.6 左右,尽管在QSAR 领域中R2>0.5时模型即被认为具有预测性能[65],但相较于大多数其他学者的研究而言则处于较低水平[66-68]。
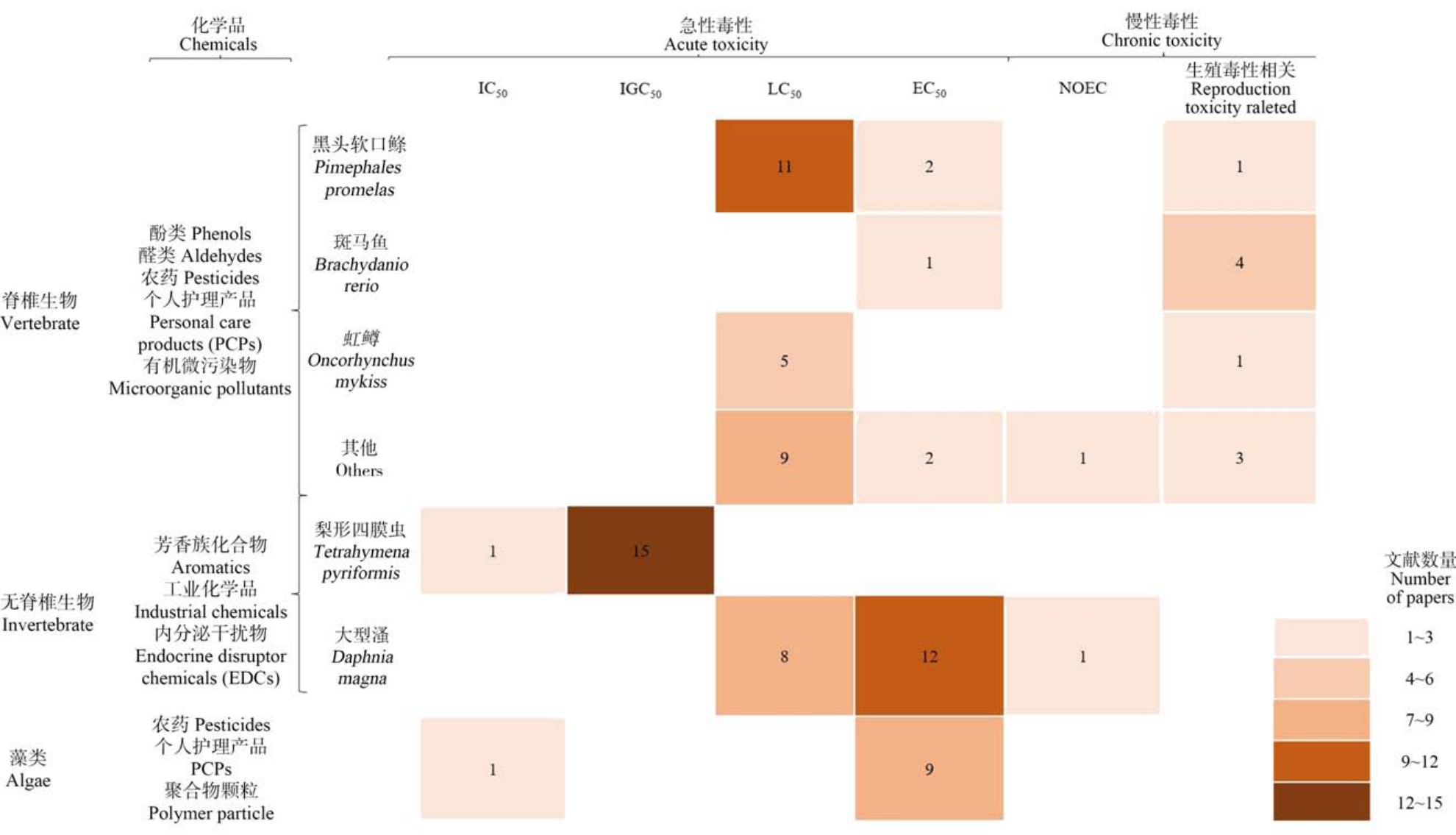
图3 各文献中使用到的水生物种与毒性终点
注:IC50 表示半抑制浓度;IGC50 表示半抑制生长浓度;LC50 表示半数致死浓度;EC50 表示半数效应浓度;NOEC 表示无观测效应浓度。
Fig.3 Aquatic creatures and toxicity endpoints applied in papers
Note: IC50 stands for 50% inhibitory concentration; IGC50 stands for 50% impairment growth concentration; LC50 stands for lethal concentration 50%;EC50 stands for concentration for 50% of maximal effect; NOEC stands for no observed effect concentration.
2.2 模型性能评估
2.2.1 描述符选择及AD 评估
经过RFE 方法筛选,最终选择了ATSC0m、ATSC7p、MATS3i 和 TpiPC 作为输入变量。 其中 ATSC0m、ATSC7p 和 MATS3i 是 2D 自相关描述符,ATSC0m 和ATSC7p 分别为原子质量加权和原子极化率加权的 Broto-Moreau 中心自相关描述符,MATS3i 是电离势加权的 Moran 中心自相关描述符,分别表征了原子质量、极化率与电离势的影响;TpiPC 则与步进计数的常规键序 ID 号相关[69-70]。使用Ambit Discovery 构建的AD 部分表征如图4所示,软件计算结果显示训练集与测试集中均无化合物落在AD 之外,这说明选取的训练集具有良好的代表性。
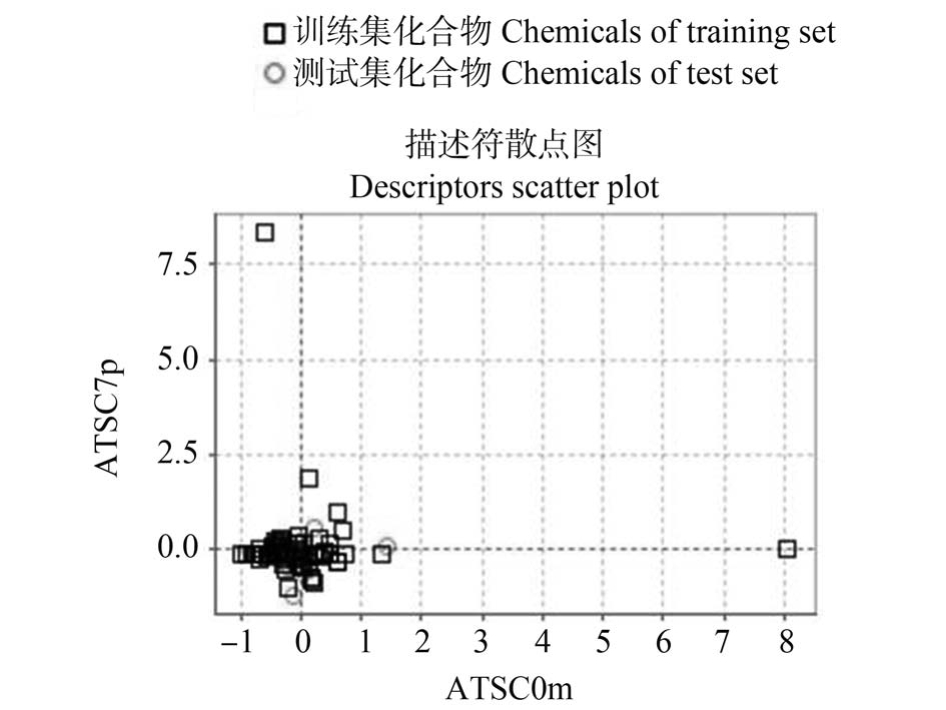
图4 基于Euclidean 距离的应用域表征
Fig.4 Application domain based on Euclidean distance
分子描述符的数值变化对毒性带来的影响如图5 所示,图 5 中(a)、(b)、(c)和(d)分别为 ATSC0m、ATSC7p、MATS3i 和 TpiPC。 蓝色柱状条代表标准化后的每个化合物的分子描述符的数值;橙色柱状条代表毒性,存在与否表示该化合物是否具有较高毒性。 可以看出,对于描述符ATSC0m 和TpiPC,随着数值的增大,橙色柱状条开始变得相对密集,即化合物倾向于具有高毒性;ATSC7p 则与之相反,随着其数值增大,更多的化合物毒性较低;MATS3i 显示出了不同的趋势,其增大与减小时化合物毒性均较低,而在均值附近时较多的化合物具有较高毒性。

图5 分子描述符数值大小与毒性之间的关系
注:横坐标表示不同的化合物,纵坐标表示标准化后的化合物毒性值。
Fig.5 Relationship between molecular descriptors and toxicity
Note: Abscissa represents different chemicals, and ordinate represents the toxicity of chemicals after standardization.
2.2.2 性能评估
数据集中化合物名称、CAS 号和模型的预测结果如表3 所示,其中模型Ⅰ为SVM,模型Ⅱ为LNN。
表3 化学品信息及模型预测结果
Table 3 Chemical information and predicted results of models

序号No.名称Chemical name CAS 号CAS No.毒性效应Toxicity effect实际值True value模型ⅠModel Ⅰ模型ⅡModel Ⅱ1盐酸舍曲林Sertraline hydrochloride 79559-97-0 1 1 1孕三烯酮Trenbolone 10161-33-8 1 1 1 3炔雌醇Ethinyl estradiol 57-63-6 1 1 1 4四溴菊酯Tralomethrin 66841-25-6 1 1 1 5盐酸安非他酮Bupropion hydrochloride 31677-93-7 1 1 1 6氰戊菊酯Fenvalerate 51630-58-1 1 1 1 7雌二醇Estradiol 50-28-2 1 1 1 8氟氯氰菊酯Cyfluthrin 68359-37-5 1 1 1 9哒螨灵* Pyridaben* 96489-71-3 1 1 1 10 甲基谷硫磷Azinphos-methyl 86-50-0 1 1 1 11 氟节胺Flumetralin 62924-70-3 1 1 1 12 4-壬基苯酚4-nonylphenol 104-40-5 1 0 1 13 二嗪磷Diazinon 333-41-5 1 1 1 14 氯化镉Cadmium chloride 10108-64-2 1 1 1 15 二硫化四甲基秋兰姆Thiram 137-26-8 1 1 0 16 盐酸文拉法辛* Venlafaxine hydrochloride* 99300-78-4 1 1 1 17 除虫菊酯Pyrethrins 8003-34-7 1 1 1 18 氟啶胺Fluazinam 79622-59-6 1 1 1 19 甲基异唑磷Isazophos-methyl 42509-83-1 1 1 1 20 螺甲螨酯* Spiromesifen* 283594-90-1 1 1 1 21 普萘洛尔Propranolol 525-66-6 1 1 0 22 盐酸氟西汀Fluoxetine hydrochloride 56296-78-7 1 1 1 23 吡唑醚菌酯* Pyraclostrobin* 175013-18-0 1 1 1 24 三氯杀螨醇Dicofol 115-32-2 1 1 1 25 螺内酯Spironolactone 52-01-7 1 1 1 26 氟虫腈* Fipronil* 120068-37-3 1 1 1 27 蒽Anthracene 120-12-7 1 1 1 28 噻嗪酮Buprofezin 69327-76-0 1 1 1 29 咪唑菌酮* Fenamidone* 161326-34-7 1 1 1 30 (2-溴-2-硝基乙烯基)苯 (2-bromo-2-nitrovinyl) benzene 7166-19-0 1 1 1 31 吡草醚* Pyraflufen-ethyl* 129630-19-9 1 1 1 32 硒酸钠Sodium selenate 13410-01-0 1 1 1 33 甲睾酮Methyltestosterone 58-18-4 1 1 1 2 34 N-[2-[4-(2-甲氧基苯基)-1-哌嗪基]乙基]-N-2-吡啶基-环己烷羧胺*N-[2-[4-(2-methoxyphenyl)-1-piperazinyl]ethyl]-N-2-pyridinyl-cyclohexanecarboxamide *162760-96-5 1 1 1 35 2,2’,4,4’,5,5’-六氯联苯2,2’,4,4’,5,5’-hexachlorobiphenyl 35065-27-1 1 1 1
续表3

序号No.名称Chemical name CAS 号CAS No.毒性效应Toxicity effect实际值True value模型ⅠModel Ⅰ模型ⅡModel Ⅱ36 2,2’,3,4,4’,5’-六氯联苯2,2’,3,4,4’,5’-hexachlorobiphenyl 35065-28-2 1 1 1 37 2,2’,5,5’-四氯联苯 2,2’,5,5’-tetrachlorobiphenyl 35693-99-3 1 1 1 38 2,2’,4,5,5’-五氯联苯 2,2’,4,5,5’-pentachlorobiphenyl 37680-73-2 1 1 1 39 敌草隆Diuron 330-54-1 1 1 0 40 比卡鲁胺* Bicalutamide* 90357-06-5 1 1 1 41 硫酸铜Copper sulfate 7758-98-7 1 1 1 42 地塞米松Dexamethasone 50-02-2 1 1 1 43 阿特拉津Atrazine 1912-24-9 1 1 1 44 唑菌腈* Fenbuconazole* 114369-43-6 1 1 1 45 1-氯-4-[1-(4-氯苯基)乙基]苯1,1’-ethylidenebis[4-chlorobenzene]3547-04-4 1 1 1 46 甲维盐* Emamectin benzoate* 155569-91-8 1 1 1 47 雄诺龙Stanolone 521-18-6 1 1 1 48 氯化铜Copper chloride 7447-39-4 1 0 0 49 邻苯二甲酸丁苄酯Benzyl butyl phthalate 85-68-7 1 1 1 50 1,3,5-三硝基苯1,3,5-trinitrobenzene 99-35-4 1 0 0 51 硝酸镍Nickel nitrate 13138-45-9 1 1 1 52 乙烯菌核利Vinclozolin 50471-44-8 0 0 0 53 2-甲基-4 氯苯氧乙酸异辛酯2,4-D-2-ethylhexyl 1928-43-4 0 0 0 54 氯苯嘧啶醇Fenarimol 60168-88-9 0 1 1 55 虫酰肼* Tebufenozide* 112410-23-8 0 1 1 56 2,4,6-三硝基甲苯2,4,6-trinitrotoluene 118-96-7 0 0 1 57 氰氟草酯* Cyhalofop-butyl* 122008-85-9 0 1 1 58 双酚A Bisphenol A 80-05-7 0 0 0 59 2,2’,4,4’-四羟基二苯甲酮2,2’,4,4’-tetrahydroxybenzophenone 131-55-5 0 0 1 60 酮康唑Ketoconazole 65277-42-1 0 0 1 61 氯化铵Ammonium chloride 12125-02-9 0 0 0 62 氯贝酸Clofibric acid 882-09-7 0 0 0 63 苯扎氯铵Benzyldimethyltetradecylammonium chloride 68424-85-1 0 0 1 64 氟吡菌胺* Fluopicolide* 239110-15-7 0 0 1 65 β-谷甾醇 Beta-Sitosterol 83-46-5 0 1 1 66 异丙甲草胺Metolachlor 51218-45-2 0 0 0 67 扑草净Prometryn 7287-19-6 0 1 1 68 黑索金Hexogen 121-82-4 0 0 0 69 直链烷基苯磺酸Sodium (1-methylundecyl) benzenesulfonate 42615-29-2 0 0 0 70 吉非罗齐Gemfibrozil 25812-30-0 0 0 0 71 全氟辛基磺酸钾Potassium perfluorooctane sulfonate 2795-39-3 0 0 0 72 四氟醚唑* Tetraconazole* 112281-77-3 0 0 0
续表3

注:*代表测试集化合物;模型Ⅰ为SVM,模型Ⅱ为LNN,下同。
Note: *denotes compounds of test set; model Ⅰ represents SVM, and model Ⅱ represents LNN; the same below.
序号No.名称Chemical name CAS 号CAS No.毒性效应Toxicity effect实际值True value模型ⅠModel Ⅰ模型ⅡModel Ⅱ73 扑灭通Prometon 1610-18-0 0 0 0 74 硫氰酸钾Potassium thiocyanate 333-20-0 0 0 0 75 阿替洛尔Atenolol 29122-68-7 0 0 0 76 氨甲基膦酸(Aminomethyl)phosphonic acid 1066-51-9 0 0 0 77 抗倒酯* Trinexapac-ethyl* 95266-40-3 0 0 0 78 啶虫脒* Acetamiprid* 135410-20-7 0 0 0 79 氯化钠Sodium chloride 7647-14-5 0 0 0 80 全氟辛酸Perfluorooctanoic acid 335-67-1 0 0 0 81 异佛尔酮Isophorone 78-59-1 0 0 0 82 硫酸钠Sodium sulfate 7757-82-6 0 0 0 83 绿草定三乙胺盐Triclopyr triethylamine salt 57213-69-1 0 0 0
训练集和测试集的评估如表4 所示。 其中,SVM 在训练集和测试集上的预测准确率分别为0.91 和0.88 左右,均达到了较好的水平,说明预测能力可以接受;模型对测试集的预测结果中,对高毒性与低毒性化合物的召回率,即SE 与SP 分别为1.00 与0.67,相比训练集中的0.93 与0.88 来说不够均衡,这可能是由于测试集数据量较少导致的,但是SE 较高可以减少实际有毒化合物漏检的可能性;训练集与测试集的预测准确率差距不大,说明模型没有发生过拟合。 SVM 与 LNN 构建模型得到的ROC 曲线分别如图6 和图7 所示,其中SVM 的训练集与测试集的AUC 分别为0.93 和0.88,远大于下限0.5,因此这是一个较好的分类器。
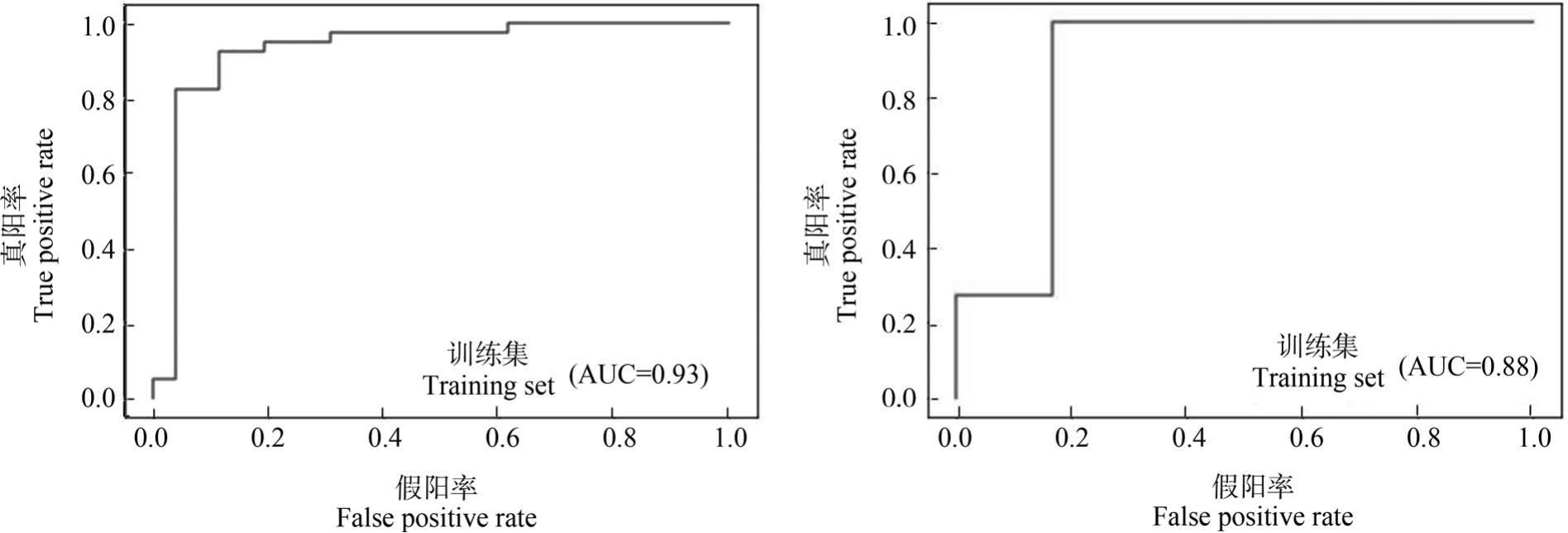
图6 由SVM 构建模型得到的训练集与测试集受试者工作特征(receiver operating characteristic, ROC)曲线
注:AUC 表示曲线下面积。
Fig.6 Receiver operating characteristic (ROC) curve for training set and test set based on SVM
Note: AUC stands for area under curve.
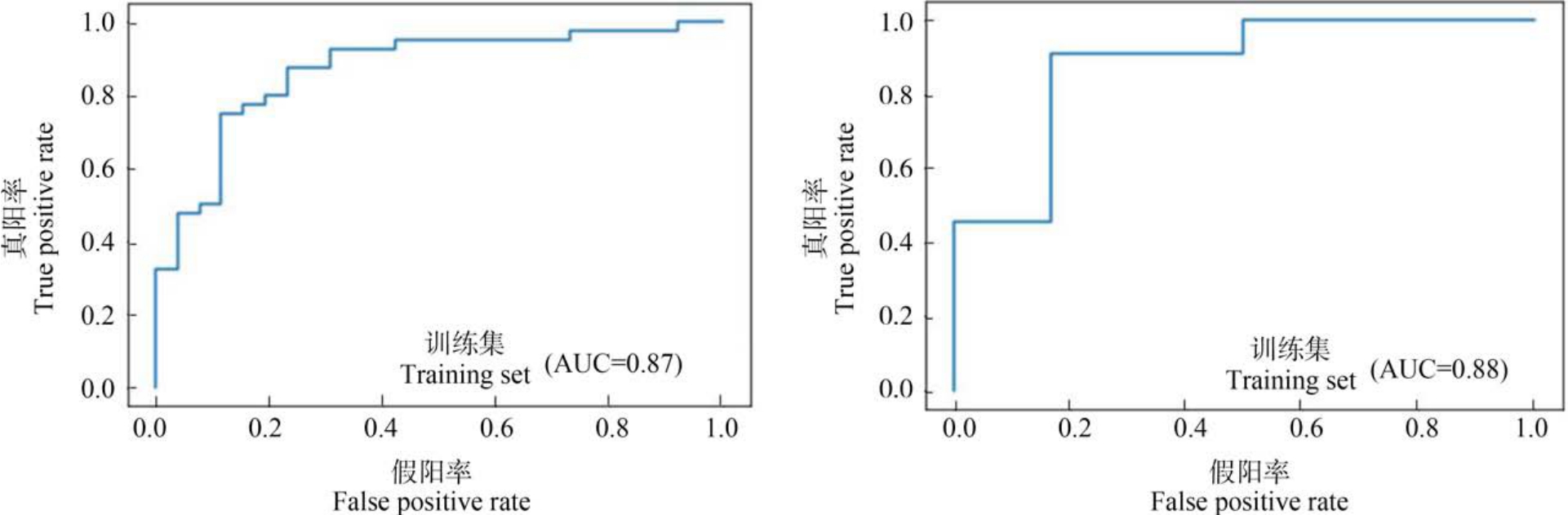
图7 由线性神经网络(linear neural network, LNN)构建模型得到的训练集与测试集ROC 曲线
Fig.7 ROC curve for training set and test set based on linear neural network (LNN)
表4 最终模型预测性能表征
Table 4 Statistical results of developed models
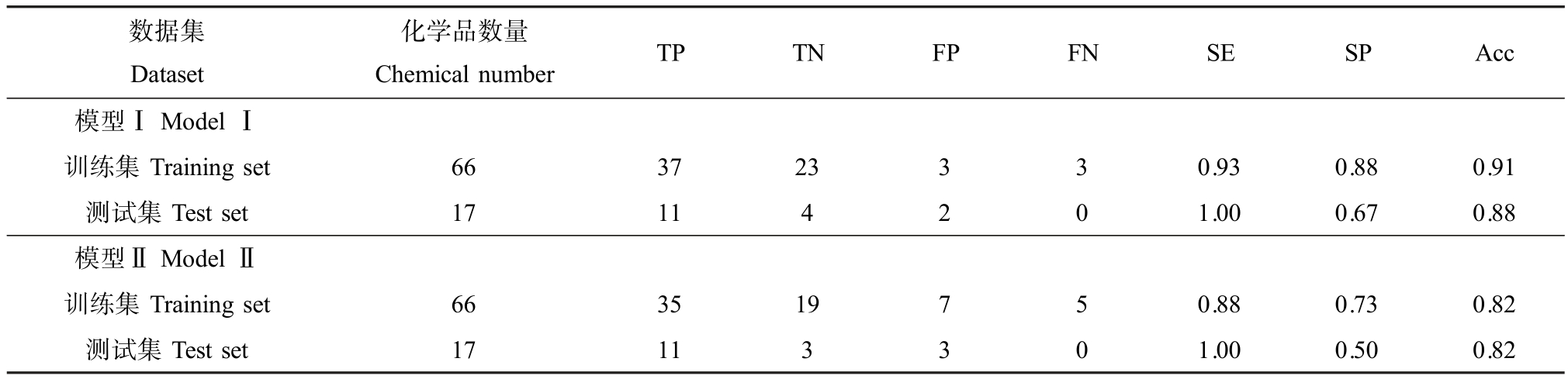
数据集Dataset化学品数量Chemical number TP TN FP FN SE SP Acc模型Ⅰ Model Ⅰ训练集Training set 66 37 23 3 3 0.93 0.88 0.91测试集Test set 17 11 4 2 0 1.00 0.67 0.88模型Ⅱ Model Ⅱ训练集Training set 66 35 19 7 5 0.88 0.73 0.82测试集Test set 17 11 3 3 0 1.00 0.50 0.82
LNN 在训练集和测试集上的预测准确率均为0.82 左右,未出现过拟合现象,SE 分别为0.88 与1.00,SP 分别为0.73 与0.50;该模型的预测结果同样有不均衡的SE 与SP 分布,可能进一步说明该问题的出现与数据集有关;训练集与测试集的AUC分别为0.87 与0.88,说明分类性能良好。
2.2.3 模型对比
(1) SVM 比 LNN 稳定。 如图 8 所示,保持超参数等条件不变,SVM 可以通过训练得到恒定最优解;而对于LNN,若训练次数不断增加,结果也在逐渐发生变化,如图9 所示,训练集预测准确率(Acc)上升,测试集预测准确率(val_acc)不变,但测试集损失函数(val_loss)却与训练集损失函数(loss)呈现相反趋势,说明模型倾向于朝过拟合发展,这可能与数据集较小有关。 SVM 的预测结果也略优于LNN,一定程度上说明SVM 较LNN 更适合于小数据集。
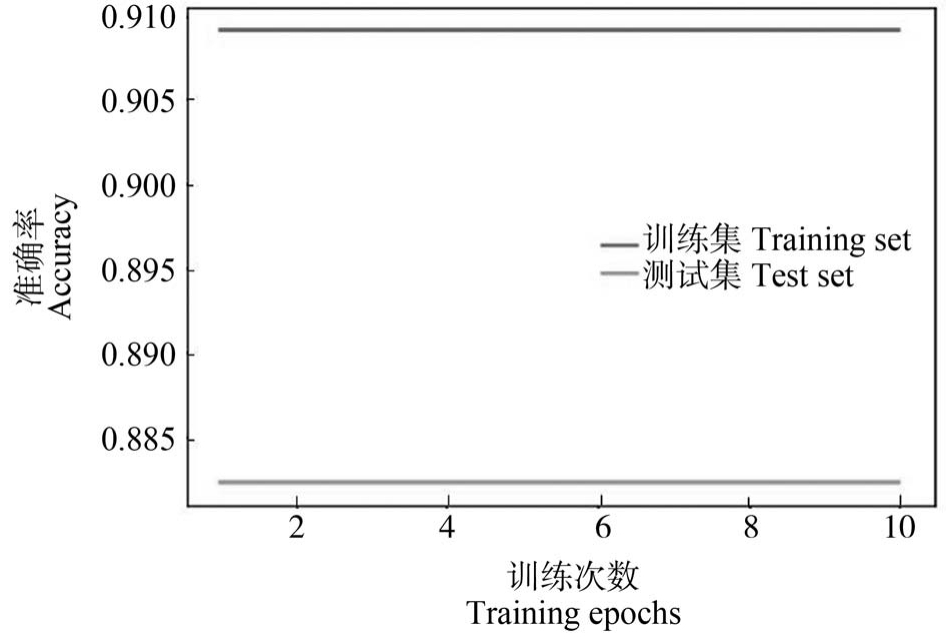
图8 经过10 次相互独立的训练后SVM 的预测准确率
Fig.8 The prediction accuracy of SVM after trained for ten times separately
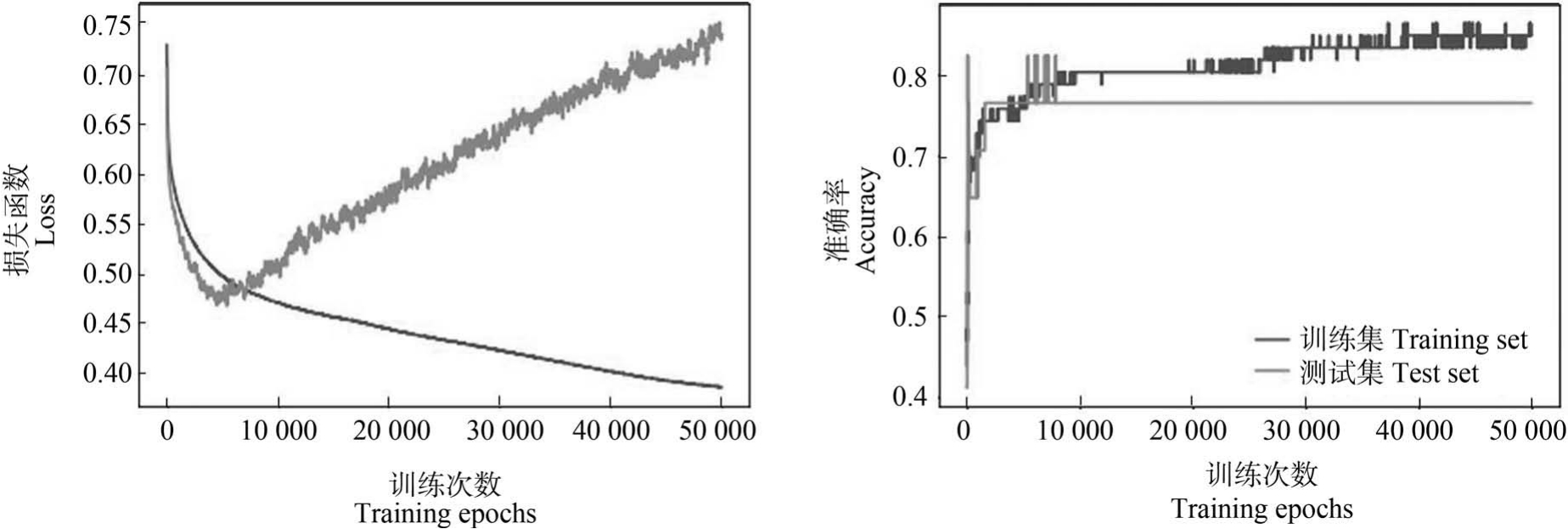
图9 LNN 的训练过程中结果持续变化
Fig.9 The result of LNN kept changing while training
(2) SVM 的训练难度相对较低。 如上所述,随着训练的进行,SVM 可以得到恒定最优解,而LNN不能;另外,在相同的训练次数内,LNN 的预测准确率也会呈现不同的变化趋势或规律,结束训练时得到的结果也可能不同,如图10 所示。
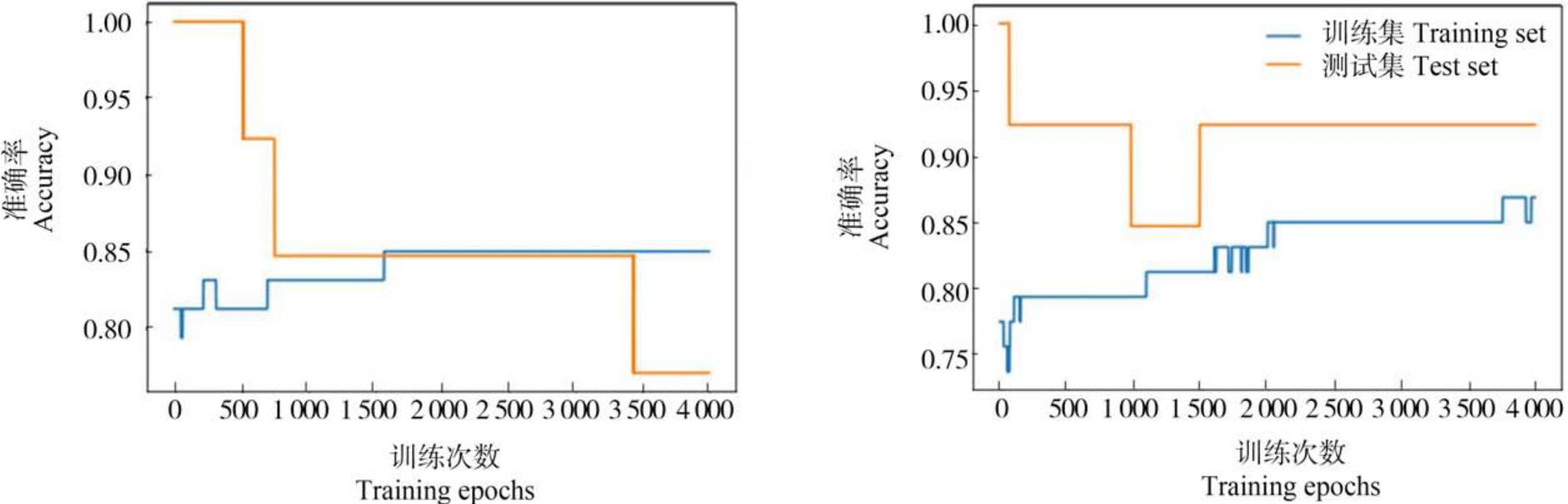
图10 相互独立的LNN 训练过程中出现不同结果
Fig.10 Separate training process of LNN led to different results
(3) SVM 的训练耗时相较于LNN 更短:SVM得到本实验中最优解的训练时间远<1 s,对LNN 每训练1 000 轮则需要20 s 左右(具体耗时与进行训练所使用的设备以及模型的超参数有关,此处仅针对本实验条件作讨论)。
3 讨论(Discussion)
本文对机器学习模型方法在水生毒性预测领域的应用研究进行了概括与总结,并使用 SVM 与LNN 结合QSAR,使用较少被其他研究者采用的EDCs 繁殖毒性的NOEC 作为终点,在黑头软口鲦数据集上构建了预测毒性高低的二分类模型;SVM在该领域中的使用频率最高;对急性毒性的研究多于慢性毒性;描述符子集的筛选是非常重要的步骤,结合了随机森林的RFE 方法较好地筛选出了合适的描述符子集,筛选结果说明化合物对黑头软口鲦的繁殖毒性可能与分子质量、极化率、电离势和相邻原子成键强度有关;根据准确率与ROC 曲线等分类模型评定标准可知,本文中所构建的模型均具有可接受的预测能力,其中SVM 的预测能力和训练表现等相较于LNN 更优,验证了SVM 更适用于中小数据集。 本实验中所使用的方法和构建的模型可为日后的AD 内未知化合物的检测优先性起到指导作用,并且为水生生物毒性领域中对EDCs 的繁殖毒性的研究提供了一定的支撑。
[1]Warner G R,Mourikes V E,Neff A M,et al.Mechanisms of action of agrochemicals acting as endocrine disrupting chemicals [J]. Molecular and Cellular Endocrinology,2020,502:110680
[2]Jakopin Ž. Assessment of the endocrine-disrupting potential of halogenated parabens: An in silico approach [J].Chemosphere,2021,264:128447
[3]Chung C, Park J, Song J E, et al.Determinants of protective behaviors against endocrine disruptors in young Korean women [J]. Asian Nursing Research, 2020, 14(3):165-172
[4]Vieira W T,de Farias M B,Spaolonzi M P,et al.Removal of endocrine disruptors in waters by adsorption, membrane filtration and biodegradation.A review [J].Environmental Chemistry Letters,2020,18(4):1113-1143
[5]华江环, 韩建, 郭勇勇, 等. 孕激素左炔诺孕酮长期低剂量暴露对雄性斑马鱼的生殖毒性[J]. 生态毒理学报,2019,14(2):176-186
Hua J H, Han J, Guo Y Y, et al. Reproductive toxicity in male zebrafish after long-term exposure to low concentra-tions of progestin levonorgestrel [J]. Asian Journal of Ecotoxicology,2019,14(2):176-186 (in Chinese)
[6]Vieira W T, de Farias M B, Spaolonzi M P, et al. Endocrine-disrupting compounds: Occurrence, detection methods, effects and promising treatment pathways—A critical review [J]. Journal of Environmental Chemical Engineering,2021,9(1):104558
[7]Bottalico L N, Weljie A M. Cross-species physiological interactions of endocrine disrupting chemicals with the circadian clock [J]. General and Comparative Endocrinology,2021,301:113650
[8]杨娜, 吴航利, 王佳, 等. 农药类内分泌干扰物对动物生殖系统干扰机制的研究进展[J]. 延安大学学报: 自然科学版,2020,39(2):87-91
Yang N,Wu H L,Wang J,et al.Research progress on the interference mechanism of pesticide endocrine disrupting chemicals on animal reproductive system [J]. Journal of Yan’an University: Natural Science Edition,2020,39(2):87-91 (in Chinese)
[9]Schilling J, Nepomuceno A I, Planchart A, et al.Machine learning reveals sex-specific 17β-estradiol-responsive expression patterns in white perch (Morone americana) plasma proteins [J]. Proteomics,2015,15(15):2678-2690
[10]蔡德雷, 陈江, 傅剑云, 等. 钱塘江水环境内分泌干扰物污染的研究[J]. 卫生研究,2011,40(4):481-484
Cai D L, Chen J,Fu J Y,et al.Study on contamination of endocrine disrupting chemicals in aquatic environment of Qiantang River [J]. Journal of Hygiene Research, 2011,40(4):481-484 (in Chinese)
[11]余方, 潘学军, 王彬, 等. 固相萃取-羟基衍生化-气相色谱/质谱联用测定滇池水体中酚类内分泌干扰物[J]. 环境化学,2010,29(4):744-748
Yu F,Pan X J,Wang B,et al.Determination of phenols in surface water of Dianchi Lake by solid extraction-hydroxyl derivatization-GC/MS [J]. Environmental Chemistry,2010,29(4):744-748 (in Chinese)
[12]张凤仙, 胡冠九, 郝英群, 等. 沿海三市饮用水源水内分泌干扰毒性研究[J]. 生态毒理学报, 2011, 6(3): 241-246
Zhang F X, Hu G J, Hao Y Q, et al. Study on endocrinedisrupting toxicity in drinking water sources of three coastal cities [J]. Asian Journal of Ecotoxicology,2011,6(3):241-246 (in Chinese)
[13]李金荣, 郭瑞昕, 刘艳华, 等. 五种典型环境内分泌干扰物赋存及风险评估的研究进展[J]. 环境化学, 2020,39(10):2637-2653
Li J R, Guo R X, Liu Y H, et al.Occurrence and risk assessment of five typical environmental endocrine disruptors [J]. Environmental Chemistry, 2020, 39(10): 2637-2653 (in Chinese)
[14]孟顺龙, 宋超, 范立民, 等. 水体中环境内分泌干扰物(EDCs)污染现状及其对鱼类的生殖危害[J]. 江苏农业学报,2013,29(1):202-208
Meng S L, Song C, Fan L M, et al. Pollution of environmental endocrine disrupting chemicals (EDCs) in water and its adverse reproductive effect on fish [J]. Jiangsu Journal of Agricultural Sciences,2013,29(1):202-208 (in Chinese)
[15]McTavish K, Stech H, Stay F.A modeling framework for exploring the population-level effects of endocrine disruptors [J]. Environmental Toxicology and Chemistry, 1998,17(1):58-67
[16]黄合田, 杨鸿波, 孙晓红, 等. 水产品中内分泌干扰物残留检测方法研究进展[J].水产科学,2021,40(2):285-293
Huang H T,Yang H B,Sun X H,et al.Detection methods of environmental endocrine disrupting chemicals in fishery products:Research progress [J].Fisheries Science,2021,40(2):285-293 (in Chinese)
[17]Jin X W, Zha J M, Xu Y P, et al. Derivation of aquatic predicted no-effect concentration (PNEC) for 2,4-dichlorophenol: Comparing native species data with non-native species data [J]. Chemosphere,2011,84(10):1506-1511
[18]Huang Q S, Bu Q W, Zhong W J, et al. Derivation of aquatic predicted no-effect concentration (PNEC) for ibuprofen and sulfamethoxazole based on various toxicity endpoints and the associated risks [J]. Chemosphere,2018,193:223-229
[19]Khan K, Roy K, Benfenati E. Ecotoxicological QSAR modeling of endocrine disruptor chemicals [J]. Journal of Hazardous Materials,2019,369:707-718
[20]Tinkov O V, Grigorev V Y, Razdolsky A N, et al. Effect of the structural factors of organic compounds on the acute toxicity toward Daphnia magna [J]. SAR and QSAR in Environmental Research,2020,31(8):615-641
[21]Fan J T, Yan Z G, Zheng X, et al. Development of inter species correlation estimation (ICE) models to predict the reproduction toxicity of EDCs to aquatic species [J].Chemosphere,2019,224:833-839
[22]Papa E, Villa F, Gramatica P. Statistically validated QSARs, based on theoretical descriptors, for modeling aquatic toxicity of organic chemicals in Pimephales promelas (fathead minnow) [J]. Journal of Chemical Information and Modeling,2005,45(5):1256-1266
[23]Mao W F, Song Y, Sui H X, et al. Analysis of individual and combined estrogenic effects of bisphenol, nonylphenol and diethylstilbestrol in immature rats with mathematical models [J]. Environmental Health and Preventive Medicine,2019,24(1):32
[24]Yangali-Quintanilla V, Sadmani A, McConville M, et al.A QSAR model for predicting rejection of emerging contaminants (pharmaceuticals, endocrine disruptors) by nanofiltration membranes [J]. Water Research, 2010, 44(2):373-384
[25]Kovarich S, Papa E, Gramatica P. QSAR classification models for the prediction of endocrine disrupting activity of brominated flame retardants [J]. Journal of Hazardous Materials,2011,190(1-3):106-112
[26]张文灏, 陈景文, 徐童, 等. 外源化合物在鱼体内生物半减期的QSAR 模型[J]. 生态毒理学报, 2019, 14(3):90-98
Zhang W H, Chen J W, Xu T, et al. QSAR models for predicting biological half-life of xenobiotics in fish [J].A-sian Journal of Ecotoxicology,2019,14(3):90-98 (in Chinese)
[27]雷太龙. 基于机器学习方法的药物毒性的理论预测研究[D]. 杭州: 浙江大学,2017:1-18
Lei T L. Theoretical prediction of drug toxicity based on machine learning approaches [D].Hangzhou: Zhejiang U-niversity,2017:1-18 (in Chinese)
[28]Cipullo S, Snapir B, Prpich G, et al. Prediction of bioavailability and toxicity of complex chemical mixtures through machine learning models [J].Chemosphere,2019,215:388-395
[29]Roohi R, Jafari M,Jahantab E,et al.Application of artificial neural network model for the identification the effect of municipal waste compost and biochar on phytoremediation of contaminated soils [J]. Journal of Geochemical Exploration,2020,208:106399
[30]薛同来, 赵冬晖, 韩菲, 等. SVR 在城市污水BOD 预测中的应用[J]. 新型工业化,2019,9(4):94-98
Xue T L, Zhao D H, Han F, et al. Application of support vector regression machine in BOD prediction of urban sewage [J]. The Journal of New Industrialization,2019,9(4):94-98 (in Chinese)
[31]Borrero L A,Guette L S,Lopez E,et al.Predicting toxicity properties through machine learning [J].Procedia Computer Science,2020,170:1011-1016
[32]Caldwell D J, Mastrocco F, Hutchinson T H, et al. Derivation of an aquatic predicted no-effect concentration for the synthetic hormone,17 alpha-ethinyl estradiol [J]. Environmental Science & Technology, 2008, 42(19): 7046-7054
[33]刘娜, 金小伟, 王业耀, 等. 生态毒理数据筛查与评价准则研究[J]. 生态毒理学报,2016,11(3):1-10
Liu N, Jin X W, Wang Y Y, et al. Review of criteria for screening and evaluating ecotoxicity data [J]. Asian Journal of Ecotoxicology,2016,11(3):1-10 (in Chinese)
[34]Sheffield T Y, Judson R S. Ensemble QSAR modeling to predict multi species fish toxicity lethal concentrations and points of departure[J].Environmental Science&Technology,2019,53(21):12793-12802
[35]Yang L, Wang Y H, Chang J, et al. QSAR modeling the toxicity of pesticides against Americamysis bahia [J].Chemosphere,2020,258:127217
[36]Nolte T M, Peijnenburg W J G M, Hendriks A J, et al.Quantitative structure-activity relationships for green algae growth inhibition by polymer particles [J]. Chemosphere,2017,179:49-56
[37]Yap C W. PaDEL-descriptor: An open source software to calculate molecular descriptors and fingerprints [J]. Journal of Computational Chemistry,2011,32(7):1466-1474
[38]In Y Y,Lee S K,Kim P J,et al.Prediction of acute toxicity to fathead minnow by local model based QSAR and global QSAR approaches [J]. Bulletin of the Korean Chemical Society,2012,33(2):613-619
[39]Marzo M, Lavado G J, Como F, et al. QSAR models for biocides: The example of the prediction of Daphnia magna acute toxicity [J]. SAR and QSAR in Environmental Research,2020,31(3):227-243
[40]Hossain K A, Roy K. Chemometric modeling of aquatic toxicity of contaminants of emerging concern (CECs) in Dugesia japonica and its inter species correlation with Daphnia and fish: QSTR and QSTTR approaches [J]. Ecotoxicology and Environmental Safety,2018,166:92-101
[41]Lei B L, Li J Z, Liu H X, et al.Accurate prediction of aquatic toxicity of aromatic compounds based on genetic algorithm and least squares support vector machines [J].QSAR & Combinatorial Science,2008,27(7):850-865
[42]Niu B, Jin Y H, Lu W C, et al. Predicting toxic action mechanisms of phenols using AdaBoost Learner [J].Chemometrics and Intelligent Laboratory Systems, 2009, 96(1):43-48
[43]Ai H X,Wu X W,Zhang L,et al.QSAR modelling study of the bioconcentration factor and toxicity of organic compounds to aquatic organisms using machine learning and ensemble methods [J]. Ecotoxicology and Environmental Safety,2019,179:71-78
[44]Sun L, Zhang C, Chen Y J, et al. In silico prediction of chemical aquatic toxicity with chemical category approaches and substructural alerts [J]. Toxicology Research,2015,4(2):452-463
[45]Pedregosa F, Varoquaux G, Gramfort A, et al. Scikitlearn: Machine learning in python [J].Journal of Machine Learning Research,2011,12:2825-2830
[46]Guo H N, Wu S B, Tian Y J, et al. Application of machine learning methods for the prediction of organic solid waste treatment and recycling processes: A review [J].Bioresource Technology,2021,319:124114
[47]李云帆. 基于机器学习的石化废气污染物排放预测[D]. 北京: 中国石油大学(北京),2018:7-10
Li Y F. Prediction of petrochemical waste gas emissions based on machine learning [D]. Beijing: China University of Petroleum (Beijing),2018:7-10 (in Chinese)
[48]Cheng F X, Shen J, Yu Y, et al. In silico prediction of Tetrahymena pyriformis toxicity for diverse industrial chemicals with substructure pattern recognition and machine learning methods [J]. Chemosphere, 2011, 82(11):1636-1643
[49]Cao Q Q, Liu L, Yang H B, et al. In silico estimation of chemical aquatic toxicity on crustaceans using chemical category methods [J].Environmental Science Processes &Impacts,2018,20(9):1234-1243
[50]OECD. Guidance document on the validation of(quantitative) structure-activity relationship [(Q)SAR]models [R].Paris: OECD,2014
[51]Ertürk M D, Saçan M T, Novic M, et al. Quantitative structure-activity relationships (QSARs) using the novel marine algal toxicity data of phenols [J]. Journal of Molecular Graphics & Modelling,2012,38:90-100
[52]He L J, Xiao K Y, Zhou C, et al. Insights into pesticide toxicity against aquatic organism: QSTR models on Daphnia magna [J]. Ecotoxicology and Environmental Safety,2019,173:285-292
[53]de Morais e Silva L, Lorenzo V P, Lopes W S, et al.Predictive computational tools for assessment of ecotoxicological activity of organic micropollutants in various water sources in Brazil [J]. Molecular Informatics, 2019, 38(8-9): e1800156
[54]王园宁, 刘会会, 杨先海. 构建有机化合物斑马鱼雌激素干扰效应的二元分类模型[J]. 生态毒理学报, 2019,14(4):163-169
Wang Y N, Liu H H, Yang X H. Development of binary classification models for predicting estrogenic activity of organic compounds on zebrafish [J].Asian Journal of Ecotoxicology,2019,14(4):163-169 (in Chinese)
[55]米晓希, 汤爱涛, 朱雨晨, 等. 机器学习技术在材料科学领域中的应用进展[J]. 材料导报, 2021, 35(15):15115-15124
Mi X X, Tang A T, Zhu Y C, et al. Research progress of machine learning in material science [J]. Materials Reports,2021,35(15):15115-15124 (in Chinese)
[56]Song I S, Cha J Y,Lee S K.Prediction and analysis of acute fish toxicity of pesticides to the rainbow trout using 2D-QSAR [J]. Analytical Science and Technology, 2011,24(6):544-555
[57]Grigor’ ev V Y, Razdol’ skii A N, Zagrebin A O, et al.QSAR classification models of acute toxicity of organic compounds with respect to Daphnia magna [J]. Pharmaceutical Chemistry Journal,2014,48(4):242-245
[58]Su Q, Lu W C, Du D S, et al. Prediction of the aquatic toxicity of aromatic compounds to Tetrahymena pyriformis through support vector regression [J]. Oncotarget,2017,8(30):49359-49369
[59]Khan K, Khan P M, Lavado G, et al.QSAR modeling of Daphnia magna and fish toxicities of biocides using 2D descriptors [J]. Chemosphere,2019,229:8-17
[60]Boone K S, di Toro D M. Target site model: Predicting mode of action and aquatic organism acute toxicity using Abraham parameters and feature-weighted k-nearest neighbors classification [J]. Environmental Toxicology and Chemistry,2019,38(2):375-386
[61]Ren S J.Modeling the toxicity of aromatic compounds to Tetrahymena pyriformis:The response surface methodology with nonlinear methods [J].Journal of Chemical Information and Computer Sciences,2003,43(5):1679-1687
[62]Önlü S,Saçan M T.Toxicity of contaminants of emerging concern to Dugesia japonica:QSTR modeling and toxicity relationship with Daphnia magna [J]. Journal of Hazardous Materials,2018,351:20-28
[63]Xia B B,Liu K P,Gong Z G,et al.Rapid toxicity prediction of organic chemicals to Chlorella vulgaris using quantitative structure-activity relationships methods [J].Ecotoxicology and Environmental Safety, 2009, 72(3):787-794
[64]Meng Y B, Lin B L. A feed-forward artificial neural network for prediction of the aquatic ecotoxicity of alcohol ethoxylate [J]. Ecotoxicology and Environmental Safety,2008,71(1):172-186
[65]Agatonovic-Kustrin S, Morton D W, Razic S. In silico modelling of pesticide aquatic toxicity [J]. Combinatorial Chemistry & High Throughput Screening, 2014, 17(9):808-818
[66]Polishchuk P G,Muratov E N,Artemenko A G,et al.Application of random forest approach to QSAR prediction of aquatic toxicity [J]. Journal of Chemical Information and Modeling,2009,49(11):2481-2488
[67]Habibi-Yangjeh A, Danandeh-Jenagharad M. Application of a genetic algorithm and an artificial neural network for global prediction of the toxicity of phenols to Tetrahymena pyriformis [J]. Chemical Monthly, 2009, 140(11):1279-1288
[68]Louis B,Agrawal V K.QSAR modeling of aquatic toxicity of aromatic aldehydes using artificial neural network(ANN) and multiple linear regression (MLR) [J]. Journal of the Indian Chemical Society,2011,88(1):99-107
[69]Sestraş R E, Jäntschi L, Bolboacǎ S D. Poisson parameters of antimicrobial activity: A quantitative structure-activity approach [J].International Journal of Molecular Sciences,2012,13(4):5207-5229
[70]Sangion A,Gramatica P.Hazard of pharmaceuticals for aquatic environment:Prioritization by structural approaches and prediction of ecotoxicity [J].Environment International,2016,95:131-143