西藏农田土和农作物中多氯联苯的分布、环境行为和健康风险评估
Distribution, Environmental Behavior, and Health Risks of Polychlorinated Biphenyls in the Tibetan Agricultural Soil and Crops
-
摘要: 南亚排放的多氯联苯类污染物(PCBs)可随大气传输到西藏南部,并沉降到农田等区域。农田中的PCBs能够经食物链进入人体,从而可能对人体健康产生影响。但目前尚无西藏农田PCBs环境过程和农作物健康风险评估的研究。本研究通过同步采集西藏农田土壤和农作物,发现西藏农田土壤、青稞和油菜PCBs的浓度均值分别为5.1 pg·g-1 dw、13.5 pg·g-1 dw和10.9 pg·g-1 dw,低于全球其他地区。青稞和油菜对PCBs的生物富集系数都大于1,说明PCBs在农作物中发生了生物富集现象。模型模拟结果显示,农田土壤中99.6%的PCBs都富集在土壤有机质中,只有0.38%的PCBs进入了植物根系。因此,青稞直接从大气中吸收PCBs是其对PCBs积累和富集的主要途径。基于饮食结构(青稞、牛肉、牛奶和酥油),西藏人群PCBs摄入均值为0.75 ng·kg-1 bw·d-1,低于安全阈值约一个数量级。PCBs的食物摄入不会对西藏居民健康状况产生明显影响。Abstract: Polychlorinated biphenyls (PCBs) emitted in South Asia can undergo long-range atmospheric transport to reach the southern Tibetan Plateau. Once PCBs are deposited into the agricultural soil of this region they have the potential to accumulate in the human body via food chains, and ultimately increase the health risk for those consuming the produce from these lands. However, despite this important issue, few studies have examined the environmental processes involved, or assessed the health risk, of PCBs in agricultural soil and crops. In the present study, agricultural soil and crops were collected from the southern Tibetan Plateau. The average concentrations of PCBs in the soil, in hulless barley (Hordeum vulgare L. var nudum Hook. f), and in rape (Brassica campestris L.) were found to be 5.1, 13.5 and 10.9 pg·g-1 dw, respectively, which are at the low end of global levels. The bio-concentration factors of all the vegetation were greater than 1, suggesting PCB accumulation. Results from a fugacity model showed that 99.6% of soil PCBs accumulated in the soil organic matter, while only 0.38% of PCBs were taken up by the roots of crops, implying that the main source of PCBs in the crops was atmospheric deposition. The dietary intake of non-dioxin-like PCBs in the southern Tibetan Plateau was found to be 0.75 ng·kg-1 bw·d-1, which is more than one order of magnitude lower than the "guidance value". In conclusion, the health risks of PCBs via dietary exposure in the southern Tibetan Plateau are low.
-
Key words:
- polychlorinated biphenyls (PCBs) /
- Tibet /
- spatial distribution /
- fugacity model /
- health risk
-
环境DNA[1](environmental DNA, eDNA)通常指从环境样品(如水样、泥样等)直接获取的DNA。环境样品中一般包含一定区域内存在的生物组织、排泄物以及分泌黏液等,通常被认为是环境DNA的主要来源[2]。从环境样品中提取DNA之后,通过PCR技术(Polymerase chain reaction, 聚合酶链式反应)扩增目标DNA片段,进一步检测分析环境DNA所携带的信息,以达到获取某一区域环境生物信息的目的[3-4]。在过去的十几年,环境DNA技术已经被逐步应用于生态环境监测(如判断某些物种的存在与否)的研究中。RONDON等[5]从土壤样品中提取环境DNA研究土壤微生物的多样性,并首次提出了environmental DNA这一术语。严格来讲,环境DNA技术于2008年开始被研究者应用于监测大型生物[6]。2010年开始,随着实时荧光定量PCR技术(qPCR)和DNA条形码技术的引入,环境DNA技术的应用从定性分析物种存在与否逐步扩展到定量分析物种丰度[1-2, 7-9]。
近年来,有研究[7]表明,存在于某一区域的生物可以通过环境样品(如水样、泥样等)并采用环境DNA技术检测到,这一新兴检测技术有可能显著提升生态监测效率。与传统生态监测方法相比,环境DNA技术有如下优点:该技术不需要捕获、捕杀目标生物,因此,是更为环境行为友好的监测手段;该技术通过PCR技术扩增目标DNA片段,对DNA的检出限低于1 pg,与传统方法相比对低丰度生物有更灵敏的检出限;该技术对于物种的鉴别基于基因序列而非传统方法的人眼判断,与传统方法相比对于物种鉴别的准确性更强。由于采样方式与判别标准不同于传统生态监测技术,环境DNA技术有望大幅提升生态监测的可行性与准确性。然而作为新兴生态监测手段,环境DNA技术仍然存在一些缺陷,如样品处理与检测手段有待进一步优化,环境DNA在环境中的迁移变化行为尚不明确等。着力解决这些问题可促进该技术的推广与应用,这些问题也是相关研究领域的重点研究课题。而对环境DNA降解动力学及环境影响因素的研究,可提高环境DNA数据对于周边环境的指示意义,如在不同的地域环境下,可根据周边环境特征通过获取的环境DNA数据,更准确地推断周边环境生物的丰度。
作为新兴技术,目前大部分对环境DNA的研究局限于对环境水样的分析检测。有研究报道,温度[10-11]、pH[12]等环境变量是水体环境DNA变化的影响因素。底栖生物是重要的环境质量指示生物,多数底栖生物生活在表层沉积物中,因此,表层沉积物中的环境DNA同样值得关注。目前,对于表层沉积物中的环境DNA关注较少[9],环境DNA在表层沉积物中变化的主要影响因素仍然未知,需要进一步研究探索。本研究通过小试实验引入并培养底栖生物,以底栖生物为目标生物,通过采集表层沉积物提取环境DNA的方法,探究了模拟自然环境下水体表层沉积物中环境DNA含量变化与周边环境变量的关系,揭示了表层沉积物中的环境DNA对于周边环境变化的指示意义,为环境DNA变化动力学研究提供参考。
1. 材料与方法
1.1 实验设置
本研究以日本大螯蜚(Grandidierella japonica)作为目标生物,引入不同的生物丰度,共进行了4组小试规模的实验研究,每组实验包括3个平行样本和1个空白样本,结果见表1。实验设置与部分实验数据参考本课题组已有的研究[9]。本研究所使用的日本大螯蜚来自实验室水族箱养殖。
表 1 小试实验条件设置Table 1. Setup of lab-scale experiment实验组 被测生物数量/只 石英砂/g 人造海水/mL 平行样数量/个 A 10 70 180 3 B 20 70 180 3 C 30 70 180 3 D 50 70 180 3 空白样 0 70 180 4 每个小试装置为1个250 mL烧杯(DURAN,德国),内装有70 g石英砂(HARIO WS-10BR,日本)作为沉积物和180 mL人工海水(配方见US EPA人工合成海水配方[13])。石英砂和海水在使用之前均已在灭菌锅中进行高温灭菌。实验启动时,4组小试装置中的平行样分别引入10、20、30和50只日本大螯蜚并依次命名为A组、B组、C组和D组,所有小试装置放入25 ℃恒温箱中运行并通过空气泵持续曝气。日本大螯蜚在实验装置中养殖4 d后全部取出,之后启动实验。在实验启动后的第0、6、12、18、24、72、144、264、384小时进行取样,每次取样从每个实验组烧杯中取出1.5 g表面石英砂和0.3 mL水样。石英砂用于环境DNA提取与qPCR分析,水样用于细菌数分析。在实验开始后,烧杯内水中的pH、DO(溶解氧)、盐度、电导率等指标在实验启动后的第0、72、144、264、384小时各检测1次。4个空白样只含有高温灭菌过后的石英砂和人造海水,在实验开始和结束后进行取样分析检测。
1.2 DNA提取
从实验过程中所取的石英砂沉积物样品中,采用CTAB(cetyltrimethylammonium bromide)方法提取环境DNA,具体操作流程参见文献中的方法[8-9],最终将每个环境DNA样品分别溶于200 μL TE缓冲液。日本大螯蜚的动物组织DNA采用DNeasy Blood & Tissue Kit套件(QIAGEN, 德国)提取,具体操作参见厂商说明书;采用Qubit 3.0 Fluorometer仪器(Thermo Fisher Scientific, 美国)测定从动物组织提取的DNA浓度。
1.3 实时荧光定量PCR(qPCR)
在进行qPCR标准物制备时,本研究共使用3对PCR引物,通过美国国立生物技术信息中心(NCBI)数据库Primer-BLAST工具设计[8, 14],引物序列及PCR退火温度见表2。qPCR标准物为使用引物413F和413R通过传统PCR反应扩增纯化后的产物,标准物片段长度为413 bp。纯化后的标准物浓度通过Qubit 3.0 Fluorometer仪器测定,进而通过式(1)计算得出标准物中的DNA片段拷贝数[15],之后分成小份于-20 ℃保存备用。
表 2 引物序列及PCR退火温度Table 2. Nucleotide sequence of primers and PCR annealing temperature引物名称 核苷酸序列 (5′~3′) 产物长度/bp 扩增目标区 退火温度/ ℃ 126F GTTTTAGGTGCTTGGGCCAG 126 线粒体COI基因 60 126R AGCATGCGCTGTTACTGAGA 413F CTTCGTTTTAGGTGCTTGGGC 413 线粒体COI基因 55 413R AGGAGGCCCCTGCTAAATGA c=aNA660b (1) 式中:c为标准物中DNA拷贝数,copies·µL−1);a为纯化后的标准物浓度,g·µL−1;b为DNA片段长度,bp;NA为阿伏伽德罗常数。
在进行qPCR检测沉积物样品中环境DNA片段含量时,每个提取出的环境DNA样品与平行样均进行2次平行qPCR检测,每个96孔反应板上包括2组拷贝数为1、10、100、1 000、1 000的qPCR标准物和3个空白对照样品。每个qPCR反应体系为20 μL,包括10 μL LightCycler® 480 SYBR Green I Master (ROCHE, 瑞士),正反引物126F与126R各0.3 μmol L−1,5 μL经超纯水10倍稀释的环境DNA提取液。qPCR反应在LightCycler® 480 Instrument II仪器上进行,首先在95 ℃下变性3 min;之后进入扩增程序并循环50次:95 ℃下解旋30 s,在60 ℃下退火30 s,在72 ℃下延伸1 min;循环结束后,进入熔解曲线程序,根据熔解曲线筛选出有效的qPCR反应并计算原环境DNA样品的拷贝数。
1.4 水质检测
水样的盐度、电导率和温度通过Mother Tool CD-4307 SD便携式手持检测仪测定,溶解氧通过Fuso DO-5509溶氧仪测定,pH通过LAQUA Twin pH计测定,水样中活细菌总数通过BD Accuri™ C6流式细胞仪检测。
1.5 数据分析
为探究环境变量对环境DNA含量变化的影响,本研究采用广义线性模型(General linear model,GLM)进行回归分析。假设环境DNA降解规律服从指数降解模型,以qPCR检测出的表层沉积物环境DNA片段含量在t时刻的降解速率(由指数降解模型求得[9])作为因变量,以环境变量中的生物丰度、总活菌量、pH、盐度、溶解氧作为自变量,缺失数据由前后2个时间点均值代替。由于水质电导率与盐度为相关非独立关系变量,因此,回归分析中未将电导率纳入自变量中。本研究所涉及的数据分析均在R语言[16](版本3.6.0)及其集成开发环境R Studio[17]上进行。
2. 结果与讨论
2.1 实验期间环境DNA变化
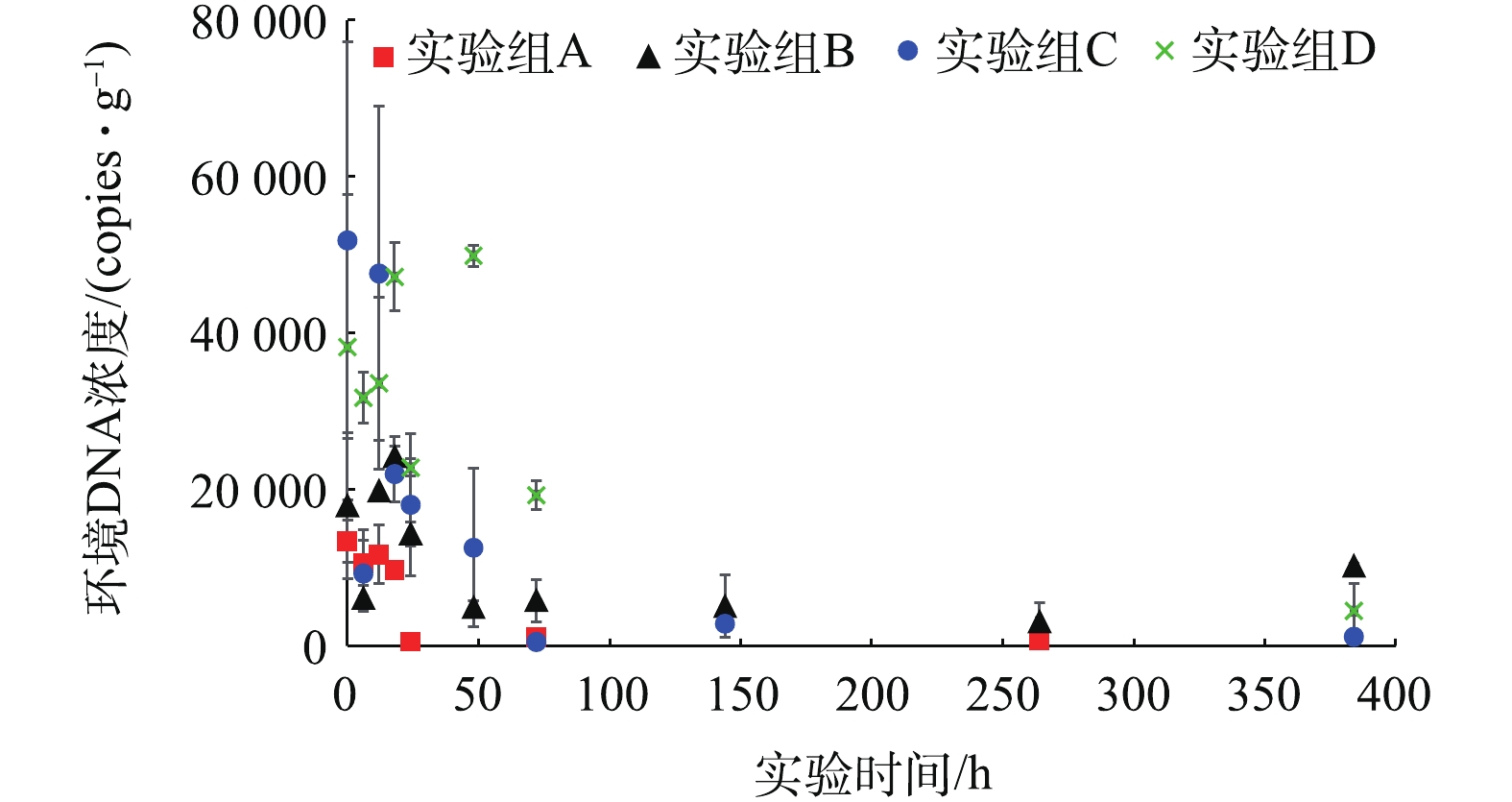
本研究在实验中设置的空白样经qPCR检测,反应均为阴性,因此,认为本研究中不同实验组之间未受到DNA污染。实验期间,由qPCR检测出的环境DNA片段变化如图1所示,服从指数降解特征,对于实验期间环境DNA指数降解动力学研究可参见已有报道[9],4组实验中环境DNA的指数降解系数分别为0.044、0.021、0.029和0.005 5 h−1。4组实验组表层沉积物中的环境DNA含量在日本大螯蜚移除之后(0 h)迅速下降,在实验进行72 h后,基本降低至5 000 copies·g−1以下,并保持在这一较低含量水平。除此以外,在生物丰度较低的实验组(A,B)表层沉积物中,环境DNA片段含量低于生物丰度较高的实验组(C,D)。
本研究中4组实验组表层沉积物中的环境DNA含量在日本大螯蜚移除之后迅速下降,在实验进行72 h后降低至较低水平。已有研究[18-20]报道,水体中环境DNA在目标生物移除后在1~14 d之后,降低至无法检出的水平,其变化速度与本研究基本处于同一水平。然而对于深层底泥中的环境DNA,有相关研究报道可持续2 000[21]、4 000[22]、6 000[23]、12 600 a[24-25]后仍被检出。本研究与以往研究的主要区别为,本研究中环境DNA载体介质为表层沉积物而非深层底泥,这说明表层沉积物中的环境DNA与水体中的环境DNA可能具有相似的变化特征。可能的原因是表层沉积物与水体直接接触,其所携带的环境DNA所处的自然环境与水环境相似,因此,二者所含有的环境DNA具有类似的变化特征。
2.2 实验期间水质变化
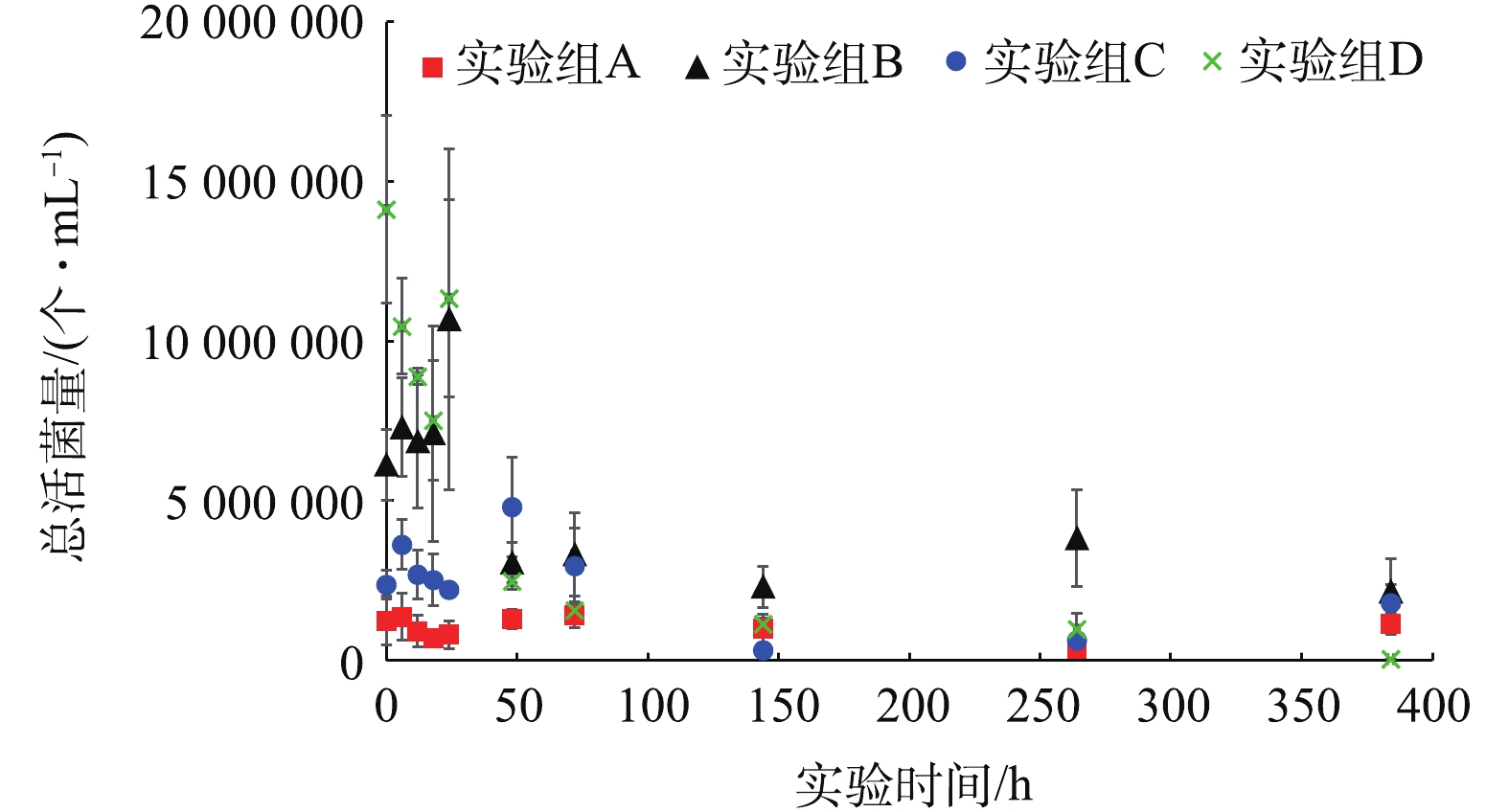
实验期间,各实验组水样中总活菌量变化如图2所示。各实验组水样中的活菌含量在日本大螯蜚移除之后(0 h)迅速下降,在实验进行72 h后基本降低至较低含量水平。除此以外,在生物丰度较低的实验组(A,B,C)水样中,总活菌含量低于生物丰度较高的实验组(D),这可能由于生物丰度较高的实验组内含有较多的有机质,导致活菌含量较高[9]。
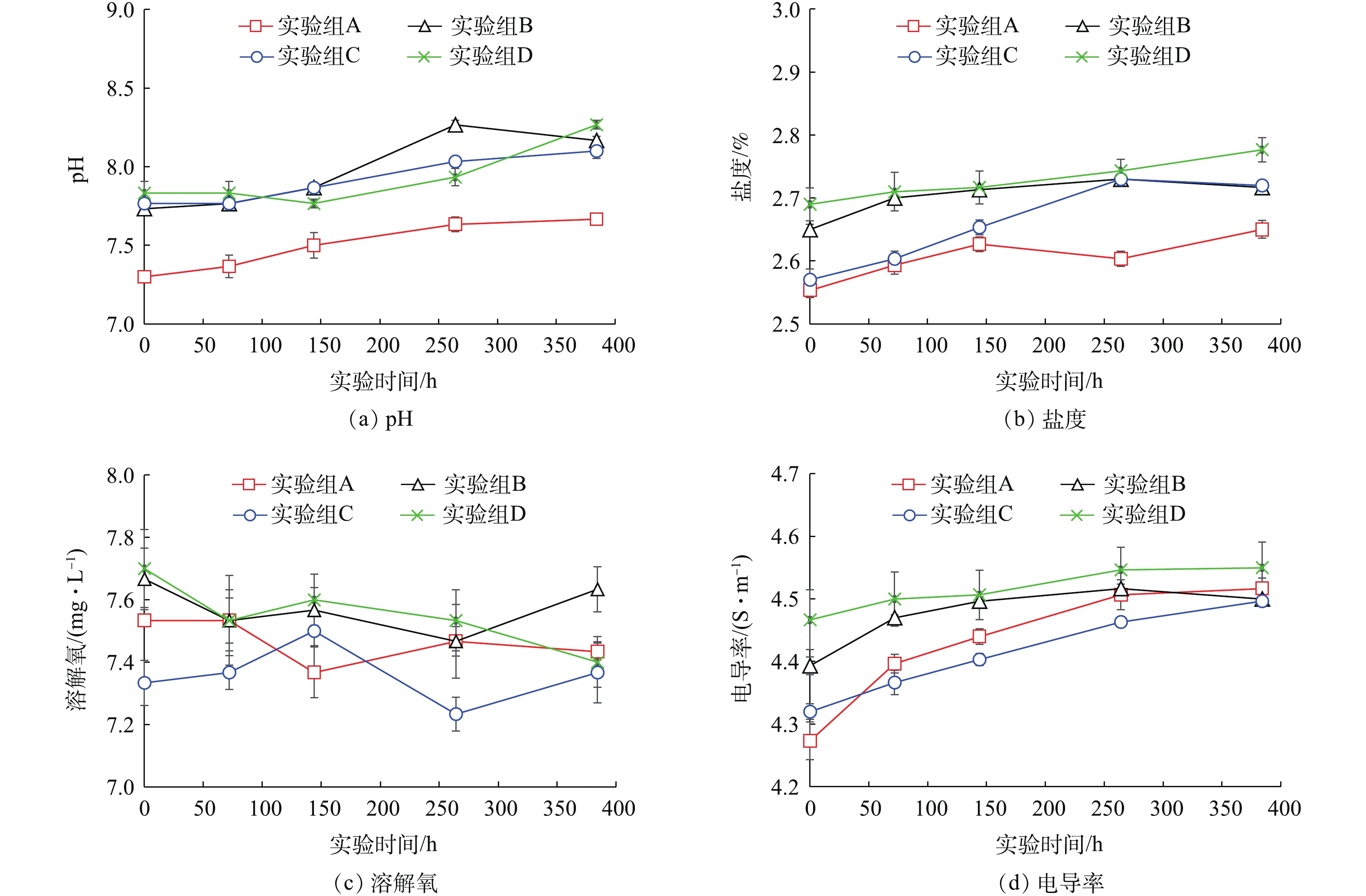
实验期间,各实验组水样中pH、盐度、溶解氧、电导率变化如图3所示。各实验组水样中的pH在日本大螯蜚移除之后(0 h)缓慢上升,在实验进行264 h后pH保持在8左右;除此以外,在生物丰度较低的实验组(A)水样中,pH低于生物丰度较高的实验组(B,C,D)。各实验组水样中的盐度在日本大螯蜚移除之后(0 h)缓慢上升,在实验进行264 h后,盐度保持在2.6%~2.7%左右;除此以外,生物丰度较低的实验组(A)水样中盐度低于生物丰度较高的实验组(B,C,D)。各实验组水样中的电导率在日本大螯蜚移除之后(0 h)缓慢上升,在实验进行264 h后,电导率保持在4.5 S·m−1左右;除此以外,在生物丰度较低的实验组(A)水样中,电导率低于生物丰度较高的实验组(B,C,D)。各实验组在实验期间一直处于曝气状态,因此,水样中的溶解氧在整个实验期间保持相对稳定,未表现出明显变化趋势。
在4组实验所检测的水质参数中,除溶解氧以外均表现出较明显的变化趋势。总活菌量表现出随时间下降的趋势,可能的原因是生物从反应器中移除后,反应器内部的有机物质随时间逐步被降解,导致微生物数量逐渐降低。pH、盐度、电导率随时间推移呈现初步上升趋势,可能的原因是反应器中的部分水分随着时间的推移蒸发,导致反应器内溶解性物质浓度上升。研究发现表层沉积物中环境DNA含量与实验组生物丰度呈正相关关系。有研究[18, 26-32]报道,物种丰度与环境DNA含量呈现一定程度的线性或指数正相关关系,可决系数在0.03~0.93之间。尽管环境DNA与生物丰度之间精确的数量关系目前尚未明确,但目前的研究发现至少证明了环境DNA存在定量估计生物丰度的可能性。
2.3 广义线性回归分析
本研究中表层沉积物中环境DNA片段降解速率与环境变量(包括生物丰度、总活菌量、pH、盐度、溶解氧)间回归关系如表3所示。水质参数中盐度与环境DNA片段降解速率呈显著负相关关系(回归系数=−3 673,P=0.005),pH与环境DNA片段降解速率呈显著正相关关系(回归系数=830.2,P=0.04)。生物丰度、总活菌量、溶解氧等水质参数与环境DNA片段降解速率呈正相关关系但不显著,回归系数分别为3.34、0.000 006、300。
表 3 环境DNA片段降解速率与环境变量间广义线性回归结果Table 3. Results of general linear regression between eDNA decay rate and environmental variables自变量 回归系数 标准差 t值 P值 生物丰度 3.34 4.02 1.830 0.41 总活菌量 0.000 006 0.000 017 0.366 0.72 pH 830.2 387.2 2.144 0.04 盐度 −6 022 1 549 −3.888 0.000 5 溶解氧 300 601 0.500 0.62 通过广义线性回归分析发现,环境DNA降解速率与水质盐度呈显著负相关关系,在盐度较高的环境下降解速率降低;与pH呈显著正相关关系,在pH较高的情况下降解速率上升。并与总活菌量、溶解氧等水质参数呈不显著正相关关系。由于实验模拟水质为常见海水水质,因此,本研究对环境变量的研究对于河口海湾底泥的环境DNA监测具有一定的参考价值。
2.4 表层沉积物中环境DNA的应用
环境DNA技术是近几年新兴生态环境监测手段,目前的相关研究主要集中于水样与鱼类监测,而对于沉积物与底栖生物的研究相对缺乏。底栖生物经常被认为是环境质量指标生物,但其具有难以辨认捕捉的缺点,通过提取分析表层沉积物样品中的环境DNA,可以克服底栖生物难以辨认捕捉的缺点,从而提升监测效率。本研究发现表层沉积物中的环境DNA与水体中的环境DNA具有相似的变化特征,因此,同样可以用来表征某一区域较短时间内的环境质量变化。WEI等[33]对日本东京湾表层沉积物中的环境DNA进行了为期1年的持续取样研究,也发现表层底泥环境DNA的变化对于1个月以内的自然环境有较强的指示意义。
目前,有关环境DNA的研究主要来自国外报道,来自国内的研究报道还较少。我国幅员辽阔,生态环境多样,随着社会经济的发展,我国环境监测的指标要求将逐步从理化性指标扩展到生物性指标,在这一趋势下,环境DNA技术有望对提高环境监测效率和效果起到较大的推进提升作用。
3. 结论
1)通过小试实验模拟海水底泥环境,研究了表层沉积物中环境DNA的变化特征与环境变量的关系。研究发现,水体表层沉积物中的环境DNA在源生物体移除后72 h内,降低至较低含量水平,变化特征与水体中的环境DNA较为相似。
2)通过广义线性回归分析发现,环境DNA降解速率与水质盐度存在显著负相关关系,与pH存在显著正相关关系。
3)表层沉积物中的环境DNA对于周边环境变化具有一定的指示意义,在未来的研究中需要对二者之间更精确的数量关系进行进一步探索。
致谢:感谢日本东京大学工学院都市工学系中岛典之教授、飞野智宏讲师提供的技术支持与论文写作指导!
-
 点击查看大图
点击查看大图
计量
- 文章访问数: 1707
- HTML全文浏览数: 1707
- PDF下载数: 58
- 施引文献: 0



 DownLoad:
DownLoad: